Equipe reconstrói características de vírus em nanoescala a partir de correlações de raios-X dispersos
 p Membros da CAMERA (da esquerda) Peter Zwart, Jeff Donatelli e Kanupriya Pande, co-autores de um artigo que descreve como o algoritmo M-TIP do grupo determinou estruturas de vírus 3D a partir de dados de difração de partícula única. Donatelli segura um modelo impresso em 3D de um dos vírus reconstruído pelo M-TIP. Crédito:Marilyn Chung, Berkeley Lab
p Membros da CAMERA (da esquerda) Peter Zwart, Jeff Donatelli e Kanupriya Pande, co-autores de um artigo que descreve como o algoritmo M-TIP do grupo determinou estruturas de vírus 3D a partir de dados de difração de partícula única. Donatelli segura um modelo impresso em 3D de um dos vírus reconstruído pelo M-TIP. Crédito:Marilyn Chung, Berkeley Lab
p Como parte de uma equipe de pesquisa internacional, Jeff Donatelli, Peter Zwart e Kanupriya Pande, do Centro de Matemática Avançada para Aplicações de Pesquisa de Energia (CAMERA) do Laboratório Nacional Lawrence Berkeley (Berkeley Lab), contribuíram com algoritmos importantes que ajudaram a atingir um objetivo proposto pela primeira vez há mais de 40 anos - usando correlações angulares de raios-X instantâneos de moléculas não cristalinas para determinar a estrutura 3D de objetos biológicos importantes. Esta técnica tem o potencial de permitir aos cientistas lançar luz sobre a estrutura e dinâmica biológica que antes eram impossíveis de observar com os métodos tradicionais de raios-X. p A descoberta resultou de um experimento de difração de partícula única conduzido na Fonte de Luz Coerente Linac (LCLS) do Departamento de Energia (DOE) pela Single-Particle Initiative organizado pelo SLAC National Accelerator Laboratory. Como parte desta iniciativa, a equipe CAMERA combinou esforços com Ruslan Kurta, um físico da instalação europeia XFEL (laser de elétrons livres de raios-X) na Alemanha, para analisar correlações angulares dos dados experimentais e usar o algoritmo de faseamento iterativo multicamadas (M-TIP) da CAMERA para realizar as primeiras reconstruções de vírus 3D bem-sucedidas a partir de correlações experimentais. Os resultados foram descritos em um artigo publicado em 12 de outubro em
Cartas de revisão física .
p "Nos últimos 40 anos, isso foi considerado um problema que não pôde ser resolvido, "disse Peter Zwart, co-autor no artigo e um biocientista físico que é membro da CAMERA com base na Divisão de Biofísica Molecular e Imagem Integrada do Laboratório de Berkeley. "Mas acontece que as ferramentas matemáticas que desenvolvemos são capazes de alavancar informações extras ocultas no problema que havia sido negligenciado anteriormente. É gratificante ver nossa abordagem teórica nos levar a uma ferramenta prática."
p
Novas oportunidades de pesquisa habilitadas por XFELs
p Durante grande parte do século passado, a técnica mais usada para determinar a estrutura molecular de alta resolução tem sido a cristalografia de raios-X, onde a amostra de interesse é organizada em uma grande rede periódica e exposta a raios-X que se espalham e formam padrões de difração que são coletados em um detector. Mesmo que a cristalografia tenha tido sucesso em determinar muitas estruturas de alta resolução, é um desafio usar esta técnica para estudar estruturas que não são suscetíveis à cristalização ou mudanças estruturais que não ocorrem naturalmente dentro de um cristal.
p A criação de instalações XFEL, incluindo o Linac Coherent Light Source (LCLS) e o X-FEL europeu, criaram oportunidades para a realização de novos experimentos que podem superar as limitações da cristalografia tradicional. Em particular, Os feixes XFEL são várias ordens de magnitude mais brilhantes e têm comprimentos de pulso muito mais curtos do que as fontes de luz de raio-X tradicionais, que permitem coletar o sinal de difração mensurável de amostras não cristalizadas menores e também estudar a dinâmica rápida. A difração de partícula única é uma dessas técnicas experimentais emergentes habilitadas pelo XFELS, onde se coleta imagens de difração de moléculas individuais em vez de cristais. Essas técnicas de partícula única podem ser usadas para estudar a estrutura e dinâmica molecular que têm sido difíceis de estudar com as técnicas de imagem tradicionais.
p
Superando limitações em imagens de partícula única por meio de correlações angulares
p Um grande desafio da imagem de partícula única é a determinação da orientação. "Em um experimento com uma única partícula, você não tem controle sobre a rotação das partículas à medida que são atingidas pelo feixe de raios-X, então, cada instantâneo de um hit bem-sucedido conterá informações sobre a amostra de uma orientação diferente, "disse o co-autor Jeff Donatelli, um matemático aplicado em CAMERA que desenvolveu muitos dos algoritmos na nova estrutura. "A maioria das abordagens de análise de partícula única tem se baseado na tentativa de determinar a orientação dessas partículas a partir das imagens; no entanto, a melhor resolução alcançável a partir dessas análises é restringida pela precisão com que essas orientações podem ser determinadas a partir de dados ruidosos. "
p Em vez de tentar determinar diretamente essas orientações, a equipe adotou uma abordagem diferente com base na ideia proposta originalmente na década de 1970 por Zvi Kam. "Em vez de examinar as intensidades de dados individuais na tentativa de encontrar a orientação correta para cada quadro medido, eliminamos essa etapa completamente usando as chamadas funções de correlação cruzada, "Kurta disse.
p Esta abordagem, conhecido como espalhamento de raios-X de flutuação, é baseado na análise das correlações angulares de ultracurto, pulsos intensos de raios-X espalhados por biomoléculas não cristalinas. "A beleza de usar dados de correlação é que eles contêm uma impressão digital abrangente da estrutura 3D de um objeto que estende as abordagens tradicionais de dispersão de solução, "Zwart disse.
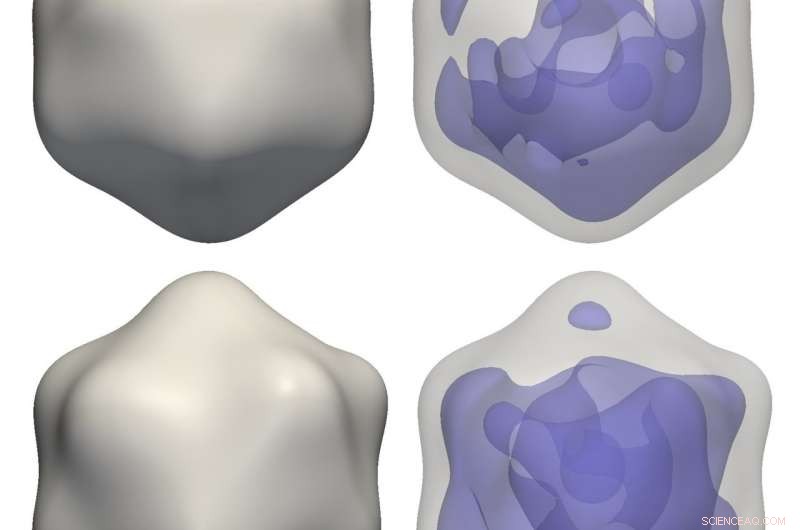 p Vírus reconstruídos:Reconstruções de um vírus do anão do arroz (parte superior) e um bacteriófago PR772 (parte inferior) a partir de dados de correlação experimental usando M-TIP. As imagens à direita mostram assimetrias no material genético interno para cada reconstrução de vírus. Crédito:Jeff Donatelli, Berkeley Lab
p Vírus reconstruídos:Reconstruções de um vírus do anão do arroz (parte superior) e um bacteriófago PR772 (parte inferior) a partir de dados de correlação experimental usando M-TIP. As imagens à direita mostram assimetrias no material genético interno para cada reconstrução de vírus. Crédito:Jeff Donatelli, Berkeley Lab
p
Reconstruindo a estrutura 3D a partir de correlações com o algoritmo M-TIP da CAMERA
p O avanço da equipe na reconstrução da estrutura 3D a partir dos dados de correlação foi possibilitado pelo algoritmo de faseamento iterativo de várias camadas (M-TIP) desenvolvido pela CAMERA. "Entre as vantagens proeminentes do M-TIP está sua capacidade de resolver a estrutura diretamente a partir dos dados de correlação, sem ter que depender de quaisquer restrições de simetria, e, mais importante, sem a necessidade de resolver o problema de determinação de orientação, "Disse Donatelli.
p Donatelli, O diretor da CAMERA, James Sethian e Zwart, desenvolveram sua estrutura M-TIP desenvolvendo uma generalização matemática de uma classe de algoritmos conhecidos como técnicas de faseamento iterativo, que são usados para determinar a estrutura em um problema mais simples, conhecido como recuperação de fase. Um artigo que descreve a estrutura M-TIP original foi publicado em agosto de 2015 no
Proceedings of the National Academy of Sciences .
p "Análises de correlação avançada em combinação com reconstruções ab-initio por M-TIP definem claramente uma rota eficiente para análise estrutural de objetos em nanoescala em XFELs, "Zwart disse.
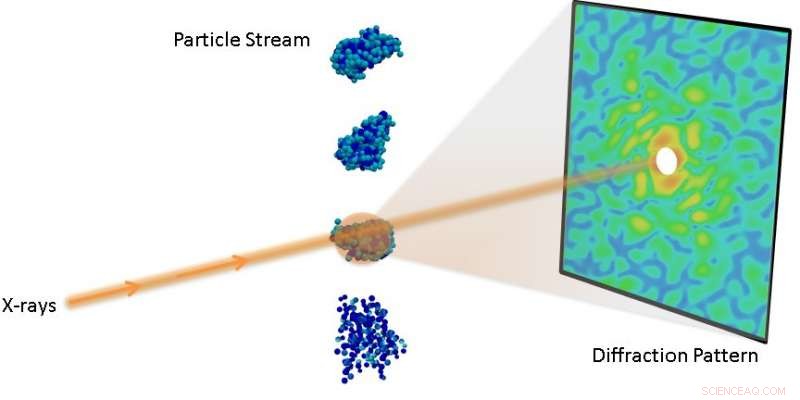 p Configuração experimental para um experimento de difração de partícula única. Crédito:Laboratório Nacional Lawrence Berkeley
p Configuração experimental para um experimento de difração de partícula única. Crédito:Laboratório Nacional Lawrence Berkeley
p
Orientações futuras para análise de correlação e M-TIP
p A equipe observa que os métodos usados nesta análise também podem ser aplicados para analisar dados de difração quando há mais de uma partícula por tiro.
p "A maioria dos algoritmos de imagem de partícula única pode lidar apenas com uma molécula de cada vez, limitando assim o sinal e a resolução. Nossa abordagem, por outro lado, é escalável para que possamos medir mais de uma partícula por vez, "disse Kurta. Imagens com mais de uma partícula por tiro permitirão aos cientistas atingir taxas de acerto muito mais altas, uma vez que é mais fácil usar um feixe largo e atingir muitas partículas de uma vez, e também evitará a necessidade de separar acertos de uma única partícula de acertos de várias partículas e tiros em branco, que é outro requisito desafiador na imagem tradicional de uma única partícula.
p Como parte do pacote de ferramentas computacionais CAMERA, eles também desenvolveram uma versão diferente do M-TIP que resolve o problema de orientação e pode classificar as imagens em estados conformacionais, e, conseqüentemente, pode ser usado para estudar pequenas diferenças biológicas na amostra medida. Esta versão alternativa do M-TIP foi descrita em um artigo publicado em 26 de junho de 2017 no
Proceedings of the National Academy of Sciences e faz parte de uma nova iniciativa de colaboração entre SLAC National Accelerator Laboratory, CÂMERA, the National Energy Research Scientific Computing Center (NERSC) and Los Alamos National Laboratory as part of DOE's Exascale Computing Project (ECP).


