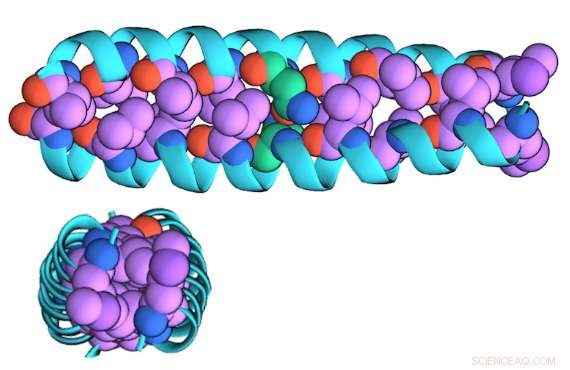
 p Uma ilustração da forma de dímero "compactada" de GCN4-p1 vista do lado (topo) e acima (parte inferior)
p Uma ilustração da forma de dímero "compactada" de GCN4-p1 vista do lado (topo) e acima (parte inferior)
p Pesquisadores da Universidade da Pensilvânia avançaram em direção a um novo método de sequenciamento de genes que uma fita de bases de DNA é lida enquanto passa por um orifício nanoscópico. p Em um novo estudo, eles mostraram que essa técnica também pode ser aplicada a proteínas como forma de aprender mais sobre sua estrutura.
p Os métodos existentes para este tipo de análise são trabalhosos, tipicamente envolvendo a coleta de grandes quantidades da proteína. Eles também costumam exigir a modificação da proteína, limitando a utilidade desses métodos para a compreensão do comportamento da proteína em seu estado natural.
p A técnica de translocação dos pesquisadores da Penn permite o estudo de proteínas individuais sem modificá-las. As amostras retiradas de um único indivíduo podem ser analisadas desta forma, abertura de aplicativos para diagnóstico e pesquisa de doenças.
p O estudo foi liderado por Marija Drndić, professor do Departamento de Física e Astronomia da Escola de Artes e Ciências; David Niedzwiecki, uma pesquisadora de pós-doutorado em seu laboratório; e Jeffery G. Saven, professor do Departamento de Química da Penn Arts &Sciences.
p Foi publicado na revista
ACS Nano .
p A técnica da equipe Penn deriva do trabalho de Drndić no sequenciamento do gene nanopore, que visa distinguir as bases em uma fita de DNA pelos diferentes percentuais de abertura que cada uma bloqueia ao passar por um poro nanoscópico. Diferentes silhuetas permitem que diferentes quantidades de um líquido iônico passem. A mudança no fluxo de íons é medida pela eletrônica em torno do poro; os picos e vales desse sinal podem ser correlacionados a cada base.
p Enquanto os pesquisadores trabalham para aumentar a precisão dessas leituras para níveis úteis, Drndić e seus colegas fizeram experiências com a aplicação da técnica a outras moléculas biológicas e estruturas em nanoescala.
p Colaborando com o grupo de Saven, eles começaram a testar seus poros em moléculas biológicas ainda mais complicadas.
p "Existem muitas proteínas que são muito menores e mais difíceis de manipular do que uma fita de DNA que gostaríamos de estudar, "Disse Saven." Estamos interessados em aprender sobre a estrutura de uma determinada proteína, como se existe como um monômero, ou combinado com outra cópia em um dímero, ou um agregado de múltiplas cópias conhecido como oligômero. "
p A detecção também costuma ser uma limitação.
p "Não há maneiras de amplificar peptídeos e proteínas como há para o DNA, "Drndić disse." Se você quiser estudar proteínas de uma fonte específica, você está preso com amostras muito pequenas. Com este método, Contudo, você pode simplesmente coletar a quantidade de dados de que precisa e o número de proteínas que deseja passar pelo poro e, em seguida, estudá-las uma de cada vez, visto que existem naturalmente no corpo. "
p Usando os nanoporos de nitreto de silício do grupo Drndić, que pode ser perfurado em diâmetros personalizados, a equipe de pesquisa decidiu testar sua técnica no GCN4-p1, uma proteína selecionada porque contém um motivo estrutural comum encontrado em fatores de transcrição e receptores intracelulares.
p "A versão do dimer é 'compactada', "Niedzwiecki disse, "É uma 'bobina enrolada' de hélices intercaladas que é aproximadamente cilíndrica. A versão do monômero é descompactada e provavelmente não é helicoidal; provavelmente é mais como uma corda."
p Os pesquisadores colocaram diferentes proporções de versões compactadas e descompactadas da proteína em um fluido iônico e as passaram pelos poros. Embora incapaz de dizer a diferença entre as proteínas individuais, os pesquisadores poderiam realizar essa análise em populações da molécula.
p "A forma de dímero e monômero da proteína bloqueia um número diferente de íons, então vemos uma queda diferente na corrente quando eles passam pelo poro, "Niedzwiecki disse." Mas temos uma gama de valores para ambos, como nem todo evento de translocação molecular é o mesmo. "
p Determinar se uma amostra específica desses tipos de proteínas está se agregando ou não poderia ser usado para entender melhor a progressão da doença.
p "Muitos pesquisadores, "Saven disse, "observaram esses longos emaranhados de peptídeos e proteínas agregados em doenças como Alzheimer e Parkinson, mas há um crescente corpo de evidências que sugere que esses emaranhados estão ocorrendo após o fato, que o que realmente está causando o problema são os conjuntos menores de proteínas. Descobrir o que são esses conjuntos e quão grandes são atualmente é muito difícil de fazer, então esta pode ser uma maneira de resolver esse problema. "
