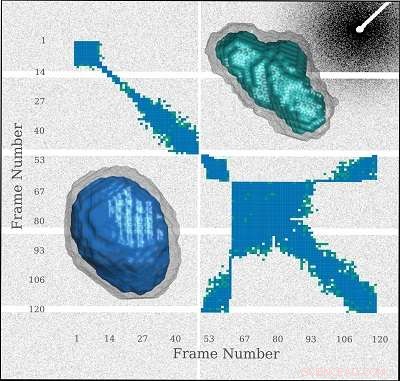
Envelopes SAXS de glicose isomerase gerados a partir de quadros coletados no início (canto inferior esquerdo) e no final (canto superior direito) no experimento. Crédito:Brooks-Bartlett et al
O espalhamento biológico de raios X a baixo ângulo (SAXS) é uma técnica experimental que fornece informações estruturais de baixa resolução sobre macromoléculas. O aumento da popularidade da técnica é resultado de melhorias recentes em software e hardware, permitindo coleta e análise de dados de alto rendimento, refletido no número crescente de linhas de luz SAXS dedicadas, como BM29 no ESRF, P12 em EMBL Hamburgo e B21 em Diamond Light Source.
Contudo, como para a maioria das outras técnicas estruturais macromoleculares, os danos da radiação ainda são um dos principais fatores que dificultam o sucesso dos experimentos. A alta proporção de solvente de amostras biológicas de SAXS significa que hidroxil, radicais hidroperoxila e elétrons hidratados são produzidos em abundância pela radiólise da água quando esta é irradiada com raios-X. Esses radicais podem interagir com as moléculas de proteína, em última análise, levando à agregação de proteínas, fragmentação ou desdobramento. Além disso, a repulsão molecular devido à carga de proteína também pode diminuir o espalhamento em ângulos baixos.
Os métodos comuns usados para reduzir os danos da radiação em amostras biológicas de SAXS estão geralmente preocupados em limitar a exposição aos raios X a qualquer determinado volume de amostra. De maneira análoga, Foi relatado que amostras de crio-resfriamento até 100 K para SAXS (cryoSAXS) aumentam a tolerância à dose de amostras de SAXS em pelo menos duas ordens de magnitude.
As aplicações das abordagens de mitigação de danos de radiação acima são incapazes de contornar completamente seus efeitos prejudiciais, em particular a mudança do perfil de espalhamento ao longo do experimento. É necessário determinar se quaisquer dois perfis de espalhamento são semelhantes para que o ruído possa ser reduzido pela média de curvas semelhantes.
Para que experimentos de diferentes pesquisadores sejam comparáveis, a progressão do dano por radiação é rastreada de maneira mais útil em função da dose absorvida pela amostra. RADDOSE-3D é um programa de software livre e de código aberto usado para calcular a distribuição tridimensional resolvida no tempo e no espaço da dose absorvida por um cristal de proteína em um experimento de cristalografia macromolecular; Contudo, não há software equivalente disponível para SAXS. Os estudos de danos de radiação em SAXS, portanto, atualmente requerem que os experimentadores parametrizem corretamente o experimento e calculem manualmente uma estimativa única da dose dentro da amostra.
Em um artigo publicado por Brooks-Bartlett et al. [(2017), J. Synchrotron. Rad. 24, doi:101107 / S1600577516015083], extensões para RADDOSE-3D são apresentadas, que permitem o cálculo conveniente de doses para experimentos SAXS, reduzindo a carga de realizar o cálculo manualmente. Adicionalmente, os autores criaram um pacote de visualização para avaliar a similaridade dos quadros SAXS e usaram essas ferramentas para avaliar a eficácia de vários compostos radioprotetores para aumentar a tolerância à radiação da amostra de proteína glicose isomerase.