Uma abordagem elétrica de molécula única para detecção de aminoácidos e reconhecimento de quiralidade
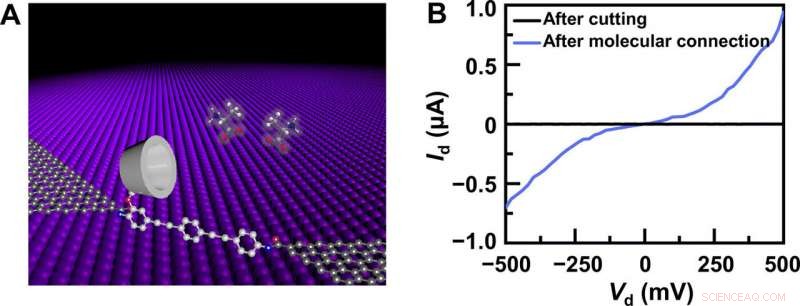 p Estrutura do dispositivo e caracterização elétrica. (A) Representação esquemática de um GMG-SMJ baseado em PM-β-CD. A máquina molecular com um PM-β-CD foi covalentemente conectada com pontos de contato de grafeno através de ligações amida. (B) Curvas I-V de GMG-SMJs após o corte por plasma de oxigênio e após a nova conexão molecular. Crédito: Avanços da Ciência , doi:10.1126 / sciadv.abe4365
p Estrutura do dispositivo e caracterização elétrica. (A) Representação esquemática de um GMG-SMJ baseado em PM-β-CD. A máquina molecular com um PM-β-CD foi covalentemente conectada com pontos de contato de grafeno através de ligações amida. (B) Curvas I-V de GMG-SMJs após o corte por plasma de oxigênio e após a nova conexão molecular. Crédito: Avanços da Ciência , doi:10.1126 / sciadv.abe4365
p A química analítica visa discriminar de forma eficiente entre dois aminoácidos. Em um novo relatório em
Avanços da Ciência , Zihao Liu e uma equipe de pesquisa em Química, Física, e Ciência de Materiais na China e no Japão, usou um método elétrico de molécula única baseado em nanocircuitos moleculares para formar junções de grafeno-molécula-grafeno de uma única molécula estáveis. A equipe desenvolveu essas junções moleculares através da ligação covalente de uma máquina molecular. Em seguida, eles usaram o pH para variar o tipo e a carga dos aminoácidos para encontrar flutuações de corrente multimodal distintas originadas de diversas interações hospedeiro-hóspede em relação aos cálculos teóricos. Os dados de condutância produziram tempos de nivelamento característicos e taxas de deslocamento para cada aminoácido para permitir medições in situ precisas e em tempo real. Os cientistas testaram quatro aminoácidos e seus enantiômeros (moléculas quirais que são imagens espelhadas umas das outras) para mostrar a capacidade de distingui-los em alguns microssegundos para fornecer um método simples e preciso para a identificação de aminoácidos, bem como proteína de molécula única sequenciamento. p
Blocos de construção de proteínas
p Os aminoácidos formam os blocos de construção das proteínas, moléculas com funções fisiológicas especiais e principais elementos estruturais em produtos farmacêuticos com aplicações em ciências da vida. Os pesquisadores podem reconhecer enantiômeros de aminoácidos para fornecer informações importantes relativas ao reconhecimento quiral e funções fisiológicas em sistemas biológicos. Detectando a estrutura, A pureza enantiomérica e o comportamento dinâmico dos aminoácidos podem promover a produção de novas técnicas de sequenciamento de proteínas e investigações farmacêuticas. A detecção e identificação de aminoácidos com diferentes estruturas e quiralidade são extremamente importantes na proteômica, produtos farmacêuticos e nanobiotecnologia. Contudo, as máquinas moleculares existentes funcionam apenas comparando os parâmetros termodinâmicos do conjunto, o que é insuficiente. Portanto, é importante desenvolver uma técnica molecular geral para revolucionar os métodos existentes e reconhecer as moléculas alvo com alta precisão. Nesse trabalho, Liu et al. demonstrou uma técnica de molécula única para identificar diretamente diferentes aminoácidos proteinogênicos e seus enantiômeros por meio de medições dinâmicas precisas de interações hospedeiro-hóspede com base em uma técnica de molécula única conhecida como junções de molécula de grafeno-molécula de grafeno abreviadas como GMG-SMJs para covalentemente incorporar sistemas moleculares individuais que se comportam como o canal condutor em um nanocircuito elétrico.
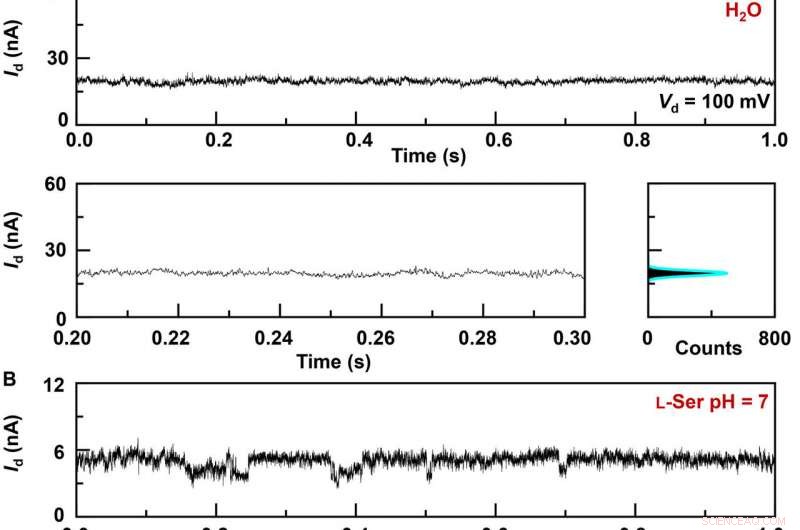 p Gravações atuais em tempo real da dinâmica de interação hospedeiro-convidado em GMG-SMJs. Curvas I-t de diferentes GMG-SMJs (topo), as partes ampliadas (canto inferior esquerdo), e seus histogramas correspondentes (canto inferior direito):(A) Um dispositivo de trabalho em água pura, (B) o mesmo dispositivo em uma solução aquosa de L-Ser 100 nM, e (C) outro dispositivo em uma solução aquosa de L-Ala 100 nM. As inserções nos histogramas são as partes aumentadas dos pequenos picos. pH =7, T =298 K, e a tensão de polarização =0,1 V. Crédito:Science Advances, doi:10.1126 / sciadv.abe4365
p Gravações atuais em tempo real da dinâmica de interação hospedeiro-convidado em GMG-SMJs. Curvas I-t de diferentes GMG-SMJs (topo), as partes ampliadas (canto inferior esquerdo), e seus histogramas correspondentes (canto inferior direito):(A) Um dispositivo de trabalho em água pura, (B) o mesmo dispositivo em uma solução aquosa de L-Ser 100 nM, e (C) outro dispositivo em uma solução aquosa de L-Ala 100 nM. As inserções nos histogramas são as partes aumentadas dos pequenos picos. pH =7, T =298 K, e a tensão de polarização =0,1 V. Crédito:Science Advances, doi:10.1126 / sciadv.abe4365
p A nova abordagem forneceu uma plataforma robusta para formar componentes eletrônicos de uma única molécula para criar dispositivos optoeletrônicos moleculares. Para desenvolver os construtos, Liu et al. covalentemente imprensou uma máquina molecular entre um par de contatos pontuais de grafeno com nano gap para detecção de aminoácidos e reconhecimento de quiralidade. Os resultados abrirão novas rotas para desenvolver a nanotecnologia do sequenciamento preciso de proteínas de uma única molécula para aplicações práticas.
p
Desenvolvimento de dispositivos, caracterização elétrica e medições em tempo real
p A equipe de pesquisa sintetizou o grafeno de camada única usando deposição química de vapor em folhas de cobre e o transferiu para dióxido de silício / pastilhas de silício e padronizou os eletrodos de metal usando fotolitografia. Para desenvolver os eletrodos de contato pontuais de grafeno com nano gap, eles usaram um método litográfico de linha tracejada. Eles então construíram GMG-SMJs (junções de molécula de grafeno-molécula de grafeno) conectando covalentemente uma única máquina molecular com eletrodos de grafeno por meio de ligações amida. A equipe mediu as curvas de corrente-tensão dos dispositivos em vários estágios para identificar a formação de GMG-SMJs. O transporte de carga através da junção resultou de conexões de uma única molécula. Eles então conduziram caracterizações elétricas dependentes do tempo para monitorar a condutância das junções moleculares únicas (SMJs) permetilada-β-ciclodextrina (PM-β-CD) em tempo real. Eles primeiro mediram as construções em água e depois em diversas soluções de diferentes aminoácidos. As trajetórias representativas do tempo atual revelaram distribuições de múltiplos picos para diferentes aminoácidos em solução, como L-serina e L-alanina. Os múltiplos estados distintos de cada aminoácido originaram-se apenas do processo de associação / dissociação entre o centro funcional PM-β-CD e os aminoácidos circundantes.
p
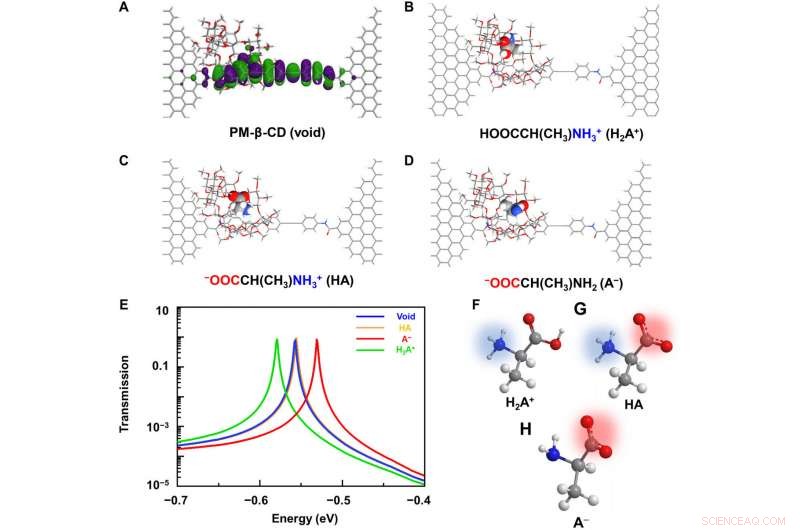 p Análises computacionais de L-Ala @ PM-β-CD SMJs. (A) Diagrama esquemático dos orbitais moleculares de fronteira calculados da máquina molecular. (B a D) Configurações moleculares típicas durante a associação com aminoácidos carregados de forma diferente:(B) cátion, (C) zwitterion, e (D) ânion. (E) Espectros de transmissão correspondentes em uma tensão de polarização zero. O vermelho, amarelo, e curvas verdes mostram a transmissão quando o ânion, zwitterion, e as formas catiônicas estavam na cavidade, respectivamente. A curva azul mostra a cavidade vazia. Os espectros completos são fornecidos na fig. S4. (F a H) Representação esquemática de aminoácidos em cátions, zwitterion, e formas de ânions. Crédito:Science Advances, doi:10.1126 / sciadv.abe4365
Análise teórica e estatística
p Análises computacionais de L-Ala @ PM-β-CD SMJs. (A) Diagrama esquemático dos orbitais moleculares de fronteira calculados da máquina molecular. (B a D) Configurações moleculares típicas durante a associação com aminoácidos carregados de forma diferente:(B) cátion, (C) zwitterion, e (D) ânion. (E) Espectros de transmissão correspondentes em uma tensão de polarização zero. O vermelho, amarelo, e curvas verdes mostram a transmissão quando o ânion, zwitterion, e as formas catiônicas estavam na cavidade, respectivamente. A curva azul mostra a cavidade vazia. Os espectros completos são fornecidos na fig. S4. (F a H) Representação esquemática de aminoácidos em cátions, zwitterion, e formas de ânions. Crédito:Science Advances, doi:10.1126 / sciadv.abe4365
Análise teórica e estatística
p Para entender melhor a correlação entre as interações hospedeiro-hóspede nas junções de molécula única PM-β-CD à base de L-alanina, Liu et al. calculou os espectros de transmissão do hospedeiro PM-β-CD com convidados L-Alanina com cargas diferentes. Para conseguir isso, eles usaram uma técnica de função de Green sem equilíbrio com base na teoria do funcional de densidade, conforme implícito no pacote Atomistix Toolkit. They noted the conductance contribution from the perturbed highest occupied molecular orbital (p-HOMO) to be dominant at low bias voltages as reflected with transmission spectra. The transmission spectra of the configurations were significantly different near the Fermi level of electrodes to afford different conductance stages. The researchers then analyzed the transitions between each level during amino acid recognition. They used L-alanine as an example to observe reversible transitions from the dissociation to form cations, zwitterions, and anion forms. The dissociation level dominated the device conductance, and the team collected the information using a four-state model, which described the transitions to understand the capacity to detect different structures of amino acids when interacting with PM-β-CD. The amino acids had at least two association processes for the carboxylic group and the other amino group. The greater the number of states recorded, the more accurate the recognition of results were.
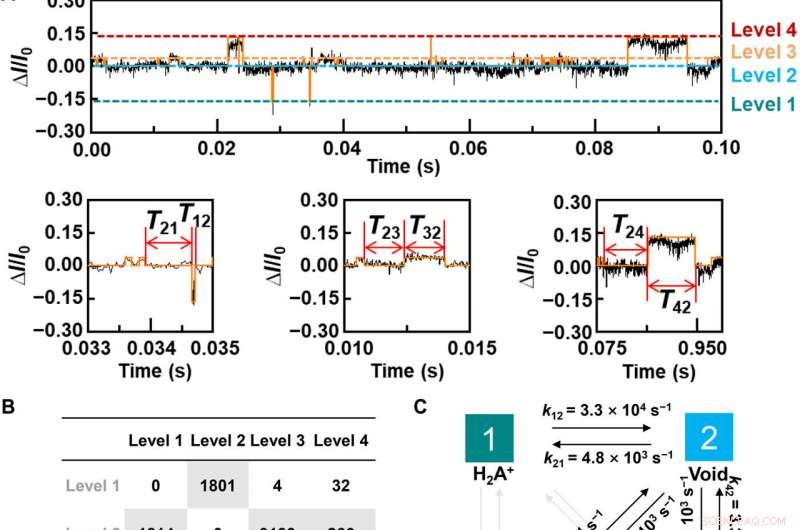 p Dynamic analysis for L-Alanine detection. (A) Plot of ΔI/I0 as a function of time during the host-guest interaction. The red curve is the idealized fit by using a QuB software. pH =7, T =298 K, and the bias voltage =0.1 V. (B) Transition statistics between each state. The horizontal columns mean the initial state. (C) Kinetic model for the L-Ala recognition process. (D to I), Plots of time intervals of (D) level 1 to level 2, (E) level 2 to level 1, (F) level 2 to level 3, (G) level 3 to level 2, (H) level 2 to level 4, and (I) level 4 to level 2 at 298 K. Credit:Science Advances, doi:10.1126/sciadv.abe4365
p Dynamic analysis for L-Alanine detection. (A) Plot of ΔI/I0 as a function of time during the host-guest interaction. The red curve is the idealized fit by using a QuB software. pH =7, T =298 K, and the bias voltage =0.1 V. (B) Transition statistics between each state. The horizontal columns mean the initial state. (C) Kinetic model for the L-Ala recognition process. (D to I), Plots of time intervals of (D) level 1 to level 2, (E) level 2 to level 1, (F) level 2 to level 3, (G) level 3 to level 2, (H) level 2 to level 4, and (I) level 4 to level 2 at 298 K. Credit:Science Advances, doi:10.1126/sciadv.abe4365
p
Enantiomer recognition and outlook
p Due to the similarity between enantiomers, it was more challenging to differentiate the structures compared to species identification. The associated energy differences were small and close to computational errors. To overcome these complexities, the team established a distinct "fingerprint database" for each amino acid to compare the conductance and kinetic data of different enantiomers. The team then achieved the current change and relaxation time data and compared them with the fingerprint database. Due to this universality, the method can also recognize chiral functional drug molecules to establish a broad approach for biomolecular detection at the single molecule-level.
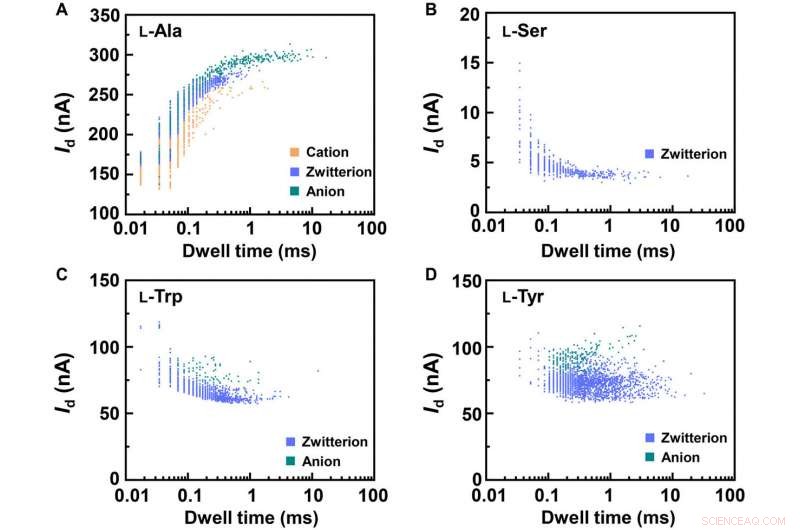 p Statistical analyses of the translocation events for different amino acids. Translocation events (current as a function of the dwell time) were analyzed for (A) L-Ala, (B) L-Ser, (C) L-Trp, and (D) L-Tyr, respectivamente. The two-dimensional (2D) contour plots are composed of all the events in 10-s recording at pH =7, showing the capability of detecting the different structures of amino acids when interacting with PM-β-CD. Crédito:Science Advances, doi:10.1126/sciadv.abe4365
p Statistical analyses of the translocation events for different amino acids. Translocation events (current as a function of the dwell time) were analyzed for (A) L-Ala, (B) L-Ser, (C) L-Trp, and (D) L-Tyr, respectivamente. The two-dimensional (2D) contour plots are composed of all the events in 10-s recording at pH =7, showing the capability of detecting the different structures of amino acids when interacting with PM-β-CD. Crédito:Science Advances, doi:10.1126/sciadv.abe4365
p Desta maneira, Zihao Liu and colleagues presented a practical single-molecule approach for real-time electrical recognition of amino acids with different structures and chirality within microseconds. The host-guest dynamic processes could be observed for diverse amino acids at the level of the anion, zwitterion and cation shuttling. Using the permethylated-β-cyclodextrin (PM-β-CD) complex, the team recognized differently charged states of amino acids based on current fluctuation range and thermodynamic/kinetic parameters. The technique can revolutionize existing methods to accurately sequence single-molecule gene/protein sequencing toward universal applications. The method can also offer a universal tool to recognize many important molecules in environmental or biological systems to understand the basis of life at the molecular level. p © 2021 Science X Network




