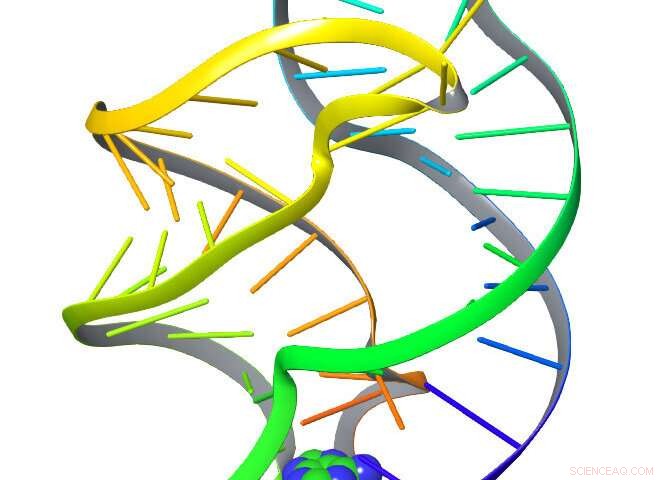
Cientistas da Universidade de Maryland aplicaram um sistema de processamento de linguagem aos movimentos de uma molécula de riboswitch retratada aqui, para entender como e quando a molécula assume diferentes formas. Crédito:Zachary Smith / UMD
Ao aplicar ferramentas de processamento de linguagem natural aos movimentos das moléculas de proteínas, Cientistas da Universidade de Maryland criaram uma linguagem abstrata que descreve as múltiplas formas que uma molécula de proteína pode assumir e como e quando ela faz a transição de uma forma para outra.
A função de uma molécula de proteína é frequentemente determinada por sua forma e estrutura, Portanto, compreender a dinâmica que controla a forma e a estrutura pode abrir uma porta para entender tudo, desde como uma proteína funciona até as causas da doença e a melhor maneira de desenvolver terapias medicamentosas direcionadas. Esta é a primeira vez que um algoritmo de aprendizado de máquina foi aplicado à dinâmica biomolecular desta forma, e o sucesso do método fornece insights que também podem ajudar no avanço da inteligência artificial (IA). Um artigo de pesquisa sobre este trabalho foi publicado em 9 de outubro, 2020, no jornal Nature Communications .
"Aqui, mostramos as mesmas arquiteturas de IA usadas para completar frases ao escrever e-mails que podem ser usadas para descobrir uma linguagem falada pelas moléculas da vida, "disse o autor sênior do jornal, Pratyush Tiwary, um professor assistente no Departamento de Química e Bioquímica da UMD e no Instituto de Ciência Física e Tecnologia. “Mostramos que o movimento dessas moléculas pode ser mapeado em uma linguagem abstrata, e que as técnicas de IA podem ser usadas para gerar histórias biologicamente verdadeiras a partir das palavras abstratas resultantes. "
As moléculas biológicas estão em constante movimento, balançando em seu ambiente. Sua forma é determinada pela forma como são dobrados e torcidos. Eles podem permanecer em uma determinada forma por segundos ou dias antes de repentinamente se abrirem e se redobrarem em uma forma ou estrutura diferente. A transição de uma forma para outra ocorre de forma muito semelhante ao alongamento de uma bobina emaranhada que se abre em estágios. À medida que diferentes partes da bobina são liberadas e desdobradas, a molécula assume diferentes conformações intermediárias.
Mas a transição de uma forma para outra ocorre em picossegundos (trilionésimos de segundo) ou mais rápido, o que torna difícil para métodos experimentais, como microscópios de alta potência e espectroscopia, capturarem exatamente como o desdobramento acontece, quais parâmetros afetam o desdobramento e quais formas diferentes são possíveis. As respostas a essas perguntas formam a história biológica que o novo método de Tiwary pode revelar.
Tiwary e sua equipe aplicaram as leis do movimento de Newton - que podem prever o movimento dos átomos dentro de uma molécula - com poderosos supercomputadores, incluindo Deepthought2 da UMD, para desenvolver modelos estatísticos de física que simulam a forma, movimento e trajetória de moléculas individuais.
Em seguida, eles alimentaram esses modelos em um algoritmo de aprendizado de máquina, como aquele que o Gmail usa para completar frases automaticamente enquanto você digita. O algoritmo abordou as simulações como uma linguagem em que cada movimento molecular forma uma letra que pode ser encadeada com outros movimentos para formar palavras e frases. Ao aprender as regras de sintaxe e gramática que determinam quais formas e movimentos se sucedem e quais não, o algoritmo prevê como a proteína se desemaranha à medida que muda de forma e a variedade de formas que assume ao longo do caminho.
Para demonstrar que seu método funciona, a equipe o aplicou a uma pequena biomolécula chamada riboswitch, previamente analisado por espectroscopia. Os resultados, que revelou as várias formas que o riboswitch poderia assumir conforme era esticado, corresponderam aos resultados dos estudos de espectroscopia.
"Um dos usos mais importantes disso, Espero, é desenvolver medicamentos muito direcionados, "Tiwary disse." Você quer drogas potentes que se liguem muito fortemente, mas apenas para o que você deseja que eles se liguem. Podemos conseguir isso se pudermos entender as diferentes formas que uma determinada biomolécula de interesse pode assumir, porque podemos fazer medicamentos que se ligam apenas a uma dessas formas específicas no momento apropriado e apenas pelo tempo que quisermos. "
Uma parte igualmente importante desta pesquisa é o conhecimento adquirido sobre o sistema de processamento de linguagem usado por Tiwary e sua equipe, que geralmente é chamada de rede neural recorrente, e, neste caso específico, uma longa rede de memória de curto prazo. Os pesquisadores analisaram a matemática subjacente à rede enquanto ela aprendia a linguagem do movimento molecular. Eles descobriram que a rede usava um tipo de lógica semelhante a um importante conceito da física estatística chamada entropia de caminho. Compreender isso abre oportunidades para melhorar as redes neurais recorrentes no futuro.
"É natural perguntar se existem princípios físicos governantes que tornam as ferramentas de IA bem-sucedidas, "Tiwary disse." Aqui nós descobrimos que, na verdade, é porque a IA é a entropia do caminho de aprendizagem. Agora que sabemos disso, abre mais botões e engrenagens que podemos ajustar para fazer uma IA melhor para a biologia e talvez, ambiciosamente, até mesmo melhorar a própria IA. Sempre que você entende um sistema complexo como IA, torna-se menos uma caixa preta e oferece novas ferramentas para usá-lo de forma mais eficaz e confiável. "