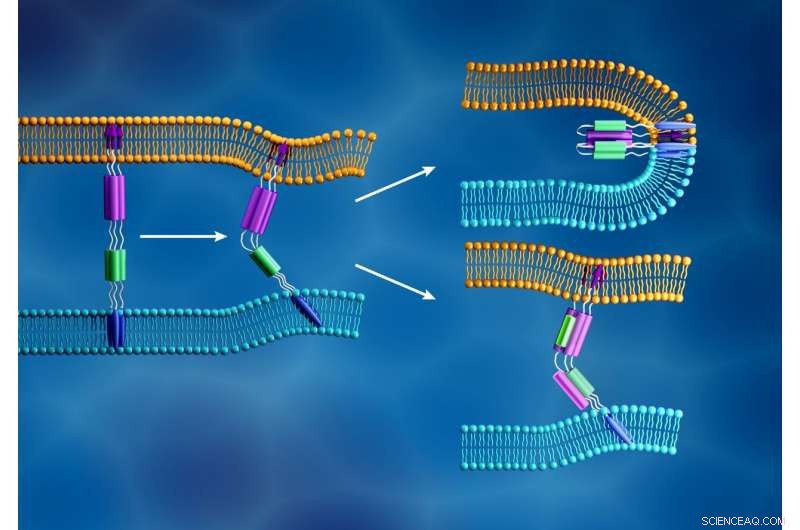
A nova membrana do coronavírus (azul claro) e a membrana da célula humana (laranja) se fundem quando o peptídeo de fusão da subunidade S2 viral (setas roxas) se insere na membrana celular e um componente diferente da subunidade S2 (roxo e verde) se dobra para formar uma estrutura compacta, conforme mostrado no canto superior direito. Em contraste, conforme ilustrado no canto inferior direito, os inibidores de fusão são projetados para prevenir a infecção viral interrompendo esse processo. Crédito:ORNL / Jill Hemman
SARS-CoV-2, o coronavírus responsável pela doença COVID-19, está infectando o mundo em um ritmo rápido. Entender como essa infecção funciona em nível molecular pode ajudar os especialistas a descobrir maneiras de moderar ou interromper a propagação.
Uma equipe de cientistas do Laboratório Nacional Oak Ridge (ORNL) do Departamento de Energia (DOE) está usando refletometria de nêutrons para fazer exatamente isso.
Os nêutrons são capazes de sondar materiais biológicos sob condições fisiológicas sem danificá-los. Aproveitando essas propriedades, os pesquisadores podem medir a dinâmica da infecção do vírus à medida que ela ocorre.
Sua missão é obter uma visão detalhada de alguns dos primeiros estágios da infecção que ocorrem na membrana celular. Essas descobertas ajudarão a equipe a testar candidatos a medicamentos antivirais que podem interromper esse processo. Os dados obtidos com esses experimentos também podem servir de subsídio para outros estudos voltados para o desenvolvimento de terapêuticas e vacinas.
Os pesquisadores estão concentrando suas análises nas proteínas de pico SARS-CoV-2, proteínas estruturais do tipo barb que cobrem a superfície do vírus e desencadeiam o processo de infecção. A proteína spike se liga a um receptor na camada externa da célula hospedeira e facilita a fusão entre as membranas viral e celular, permitindo que o vírus entre na célula e libere seu material genético. O maquinário de produção de proteínas da célula usa essa informação genética para fazer novas cópias do vírus.
Quando o SARS-CoV-2 sequestra uma célula hospedeira, sua proteína spike se divide em duas subunidades, chamados S1 e S2. As duas partes são essenciais para a infecção. A subunidade S1 contém um domínio de ligação ao receptor que reconhece e se liga a um receptor celular. Os receptores celulares são proteínas embutidas na membrana celular que podem se ligar a moléculas específicas fora da célula. Esta conexão pode fazer com que os componentes mudem de forma, o que, por sua vez, pode induzir mudanças em cascata dentro da célula. Para a proteína spike SARS-CoV-2, esta conexão ativa a subunidade S2, que ajuda o vírus a fundir sua membrana com a da célula. Portanto, a função da proteína de pico é semelhante a abrir uma porta trancada, com S1 como a chave que destranca a porta e S2 como a força que empurra a porta aberta.
Aprendendo com epidemias anteriores
A estrutura geral da proteína de pico SARS-CoV-2 é muito semelhante à do SARS-CoV, um coronavírus anterior que causou a síndrome respiratória aguda grave (SARS), e essa semelhança ajudou a equipe a desenvolver sua estratégia de pesquisa.
A subunidade S1 é o foco de muitos estudos de desenvolvimento de drogas, já que esta parte da proteína do pico provoca uma resposta imunológica no corpo humano. Contudo, estudos anteriores de SARS-CoV descobriram que a subunidade S1 experimenta altas taxas de mutação, permitindo que o vírus evite os tratamentos baseados em anticorpos, enquanto mantém sua capacidade de infectar as células. "Esta é a lição que aprendemos com a epidemia original de SARS, "disse Minh Phan, um associado de pesquisa de pós-doutorado no ORNL e investigador principal deste projeto.
Phan e seus colegas estão estudando a subunidade S2 porque esse componente da proteína spike não sofre mutação tão rapidamente. Os tratamentos que têm sucesso em inibir a função S2 podem permanecer eficazes por mais tempo.
Uma visão em nanoescala do coronavírus
Para entender melhor a dinâmica das subunidades S2 virais e das membranas das células hospedeiras, os pesquisadores estão empregando o refletômetro de líquidos (LIQREF) na Spallation Neutron Source (SNS) do ORNL. Ao medir como os nêutrons refletem em diferentes ângulos ao passar por diferentes tipos de matéria, o instrumento pode ajudar a esclarecer a estrutura de materiais biológicos em escala molecular.
A equipe primeiro sintetizou uma membrana lipídica que imita a membrana externa das células que revestem as superfícies dentro dos pulmões humanos, onde esta infecção viral pode ocorrer. Eles identificaram como os lipídios foram organizados dentro da membrana e como esse arranjo muda quando as membranas são expostas a diferentes condições, como temperatura, pressão, e acidez.
No instrumento LIQREF, os pesquisadores espalharam a membrana lipídica sobre uma fina camada de água em um aparelho chamado cocho de Langmuir. Eles então introduzem a subunidade S2 nessas membranas para observar em detalhes como as membranas S2 e lipídicas mudam de forma quando interagem.
Os nêutrons também são ideais para este estudo porque são sensíveis ao elemento hidrogênio, comum a todas as moléculas biológicas, e seus isótopos. Ao substituir alguns átomos de hidrogênio por átomos de deutério, os cientistas podem criar contraste em suas amostras e focalizar seletivamente em diferentes características estruturais. Esta técnica é útil para estudar amostras que envolvem vários componentes com densidades semelhantes, como membranas lipídicas.
"Geralmente, essas membranas não são membranas lipídicas simples, "disse John Ankner, an instrument scientist involved with this study. "They consist of lipids of a certain structure, lipids of another structure, colesterol, proteínas, and things that come in contact with them."
To capture this complexity, the research team is investigating multiple versions of the membrane, changing the contrast of the sample with deuterium each time.

Researchers at ORNL are using neutron scattering at the Spallation Neutron Source to better understand how spike proteins help the COVID-19 virus infect human cells and what drugs could be effective in stopping them. This research team includes John Ankner (left) and Minh Phan (right). Crédito:ORNL / Genevieve Martin
"By taking multiple measurements and assembling all of this information together, you can create a single picture of how these different components go together, " said Ankner.
The information derived from these experiments will then help steer the team's efforts in selecting and testing drug candidates that could block this interaction, such as fusion inhibitors that successfully blocked original SARS-CoV infections. If these inhibitors can stop the new coronavirus from invading healthy cells, existing drugs could potentially be repurposed to treat COVID-19 patients. The results may also help guide the design of new fusion inhibitors.
Capturing infection
While other studies have used protein crystallography to better understand the atomic structure of the coronavirus S2 subunit alone, this project is analyzing how S2 changes shape when interacting with a lipid membrane. A shape change could be important for inducing actions within a cell after the spike S1 subunit binds to the cell receptor. Phan also notes that the LIQREF instrument allows the team to measure these dynamics under physiological conditions, whereas protein crystallography only allows researchers to capture what the S2 subunit looks like in a crystallized form.
"At ORNL, we have the right tools to study the dynamics of the interaction under physiological conditions. This allows us to better understand how the S2 subunit moves and changes shape naturally in a wet environment, " said Phan. "Such information could complement what experts already know about the protein from crystallography. If we can help verify what this mechanism looks like, then we may have a clearer understanding to guide the development of drugs that block the fusion process.
Collaboration is key
Claro, learning more about the S2 subunit and its certain behaviors depends on the ability to grow quality samples, which involves synthesizing S2 subunit proteins, purifying them, and preparing them for experimentation.
Phan and Ankner note that this part of their research has been made possible only through collaboration with labs across ORNL and at outside institutions.
The S2 subunit protein was synthesized in mammalian cell cultures by Steve Foster, a biomedical researcher at the University of Tennessee Medical Center in Knoxville, Tennessee. Through this method, he can develop S2 proteins for research that retain several aspects of its natural structure and function.
"In our lab we routinely use mammalian cell cultures for protein production, so we hope we've produced an S2 protein best suited for this research analysis. Our proximity to ORNL also works well in that the sample doesn't have to travel far, meaning less risk of damaging the protein or distorting its original structure, which is critical for this work, " said Foster.
Following its synthesis, the sample was purified by Jessy Labbé and Michael Melesse Vergara from ORNL's Biosciences Division. Scientists from the ORNL Neutron Sciences Directorate then performed a series of tests to confirm the structure of the sample protein and check its purity. This effort was implemented by Yichong Fan and Wellington Leite from the Bio-Labs team, and Jacob Kinnun and Mary Odom from the SNS team.
"We put an enormous effort into making sure the protein has the right properties going into the experiment. If it does not, we could get spurious results and misinterpret what we're doing, " said Hugh O'Neill, director of ORNL's Center for Structural and Molecular Biology and lead researcher for the Bio-Labs team.
"This virus is extremely delicate in its components, and it's a big challenge to get these materials to the neutron instrument, " said Ankner. "That's why involving various ORNL labs and the University of Tennessee is so crucial. Each step that eventually gets the sample onto our instrument requires the expertise of lots of people."
This project also relied on efforts from the LIQREF instrument staff, who were instrumental in developing the systems, protocols, and modeling frameworks necessary to run the experiments and interpret the data.
"Experts across the division, across ORNL, and from partner institutes have come together for this project, " said Phan. "We couldn't have done this without their support, and it's greater motivation to fulfill our mission."