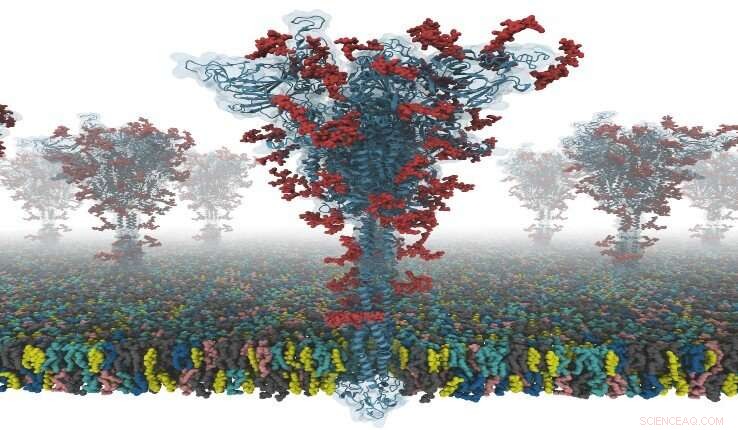
Um modelo de uma proteína S. Crédito:Dr. Yeolkyo Choi / Lehigh
O vírus SARS coronavirus 2 (SARS-CoV-2) é a causa conhecida da doença coronavírus 2019 (COVID-19). O "pico" ou proteína S facilita a entrada viral nas células hospedeiras.
Agora, um grupo de pesquisadores da Universidade Nacional de Seul, na Coreia do Sul, Universidade de Cambridge, no Reino Unido, e a Lehigh University nos EUA, trabalharam juntos para produzir os primeiros modelos de todos os átomos de código aberto de uma proteína S de comprimento total. Os pesquisadores dizem que isso é de particular importância porque a proteína S desempenha um papel central na entrada do vírus nas células, tornando-se um alvo principal para o desenvolvimento de vacinas e medicamentos antivirais.
Os detalhes podem ser encontrados em um jornal, "O desenvolvimento de um modelo de proteína de pico SARS-CoV-2 de comprimento total totalmente glicosilado em uma membrana viral" acaba de ser publicado online em The Journal of Physical Chemistry B .
Este vídeo de demonstração ilustra como construir este sistema de membrana a partir de seus modelos de proteína SARS-CoV-2 S. O programa de construção de modelos é de acesso aberto e pode ser encontrado na página inicial do CHARMM-GUI clicando no link COVID-19 Archive, ou clicando no link do arquivo no cabeçalho, em seguida, o link das proteínas COVID-19 na barra lateral esquerda.
Desenvolvido por Wonpil Im, um professor do Departamento de Ciências Biológicas e Departamento de Bioengenharia da Lehigh University, CHARMM-GUI (GUI =interface gráfica do usuário) é um programa que simula sistemas biomoleculares complexos de forma simples, com precisão e rapidez. Im o descreve como um "microscópio computacional" que permite aos cientistas entender as interações em nível molecular que não podem ser observadas de outra forma. Mais informações sobre o CHARMM-GUI podem ser encontradas neste vídeo.
"Nossos modelos são os primeiros modelos de proteína de pico (S) SARS-CoV-2 de comprimento total totalmente glicosilada que estão disponíveis para outros cientistas, "diz Im." Tive a sorte de colaborar com o Dr. Chaok Seok da Universidade Nacional de Seul na Coréia e o Dr. Tristan Croll da Universidade de Cambridge, no Reino Unido. Nossa equipe passou dias e noites construindo esses modelos com muito cuidado a partir do conhecido crio- Porções da estrutura EM. A modelagem foi muito desafiadora porque havia muitas regiões onde a modelagem simples não fornecia modelos de alta qualidade. "
Os cientistas podem usar os modelos para realizar pesquisas de simulação inovadoras e inovadoras para a prevenção e tratamento de COVID-19, de acordo com Im.
A estrutura da proteína S foi determinada com crio-EM com o RBD up (PDB ID:6VSB), e com o RBD desativado (PDB ID:6VXX). Mas, este modelo tem muitos resíduos ausentes. Então, eles primeiro modelaram os resíduos de aminoácidos ausentes, e, em seguida, outros domínios ausentes. Além disso, eles modelaram todos os glicanos (ou carboidratos) potenciais ligados à proteína S. Esses glicanos impedem o reconhecimento de anticorpos, o que torna difícil desenvolver uma vacina. Eles também construíram um sistema de membrana viral de uma proteína S para simulação de dinâmica molecular.