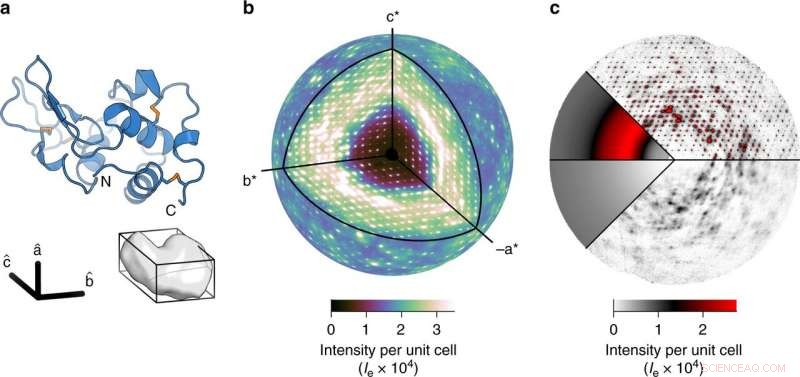
um diagrama de fita da lisozima (parte superior) e a célula unitária triclínica contendo uma proteína (parte inferior). b Um mapa tridimensional altamente detalhado de espalhamento difuso foi obtido. A esfera externa é desenhada com resolução de 2 Å. c O espalhamento total é feito de três componentes:espalhamento Compton inelástico (canto inferior esquerdo), um amplo anel isotrópico que domina o sinal difuso (canto superior esquerdo), e características variacionais no espalhamento difuso (direita). Halos intensos são visíveis nas camadas contendo picos de Bragg (plano l =0, canto superior direito). O espalhamento nublado é melhor visualizado nos planos a meio caminho entre os picos de Bragg (plano l =1∕2, inferior direito). Crédito: Nature Communications (2020). DOI:10.1038 / s41467-020-14933-6
Os biólogos estruturais de Cornell adotaram uma nova abordagem para usar um método clássico de análise de raios X para capturar algo que o método convencional nunca havia considerado:o movimento coletivo das proteínas. E eles fizeram isso criando software para costurar meticulosamente os fragmentos de dados que geralmente são desconsiderados no processo.
Seu papel, "Espalhamento difuso de raios-X a partir de movimentos correlacionados em um cristal de proteína, "publicado em 9 de março em Nature Communications .
Como biólogo estrutural, Nozomi Ando, EM. '04, Ph.D. '08, professor assistente de química e biologia química, está interessado em mapear o movimento das proteínas, e suas partes internas, para entender melhor a função da proteína. Este tipo de movimento é bem conhecido, mas tem sido difícil de documentar porque a técnica padrão para imagens de proteínas é a cristalografia de raios-X, que produz essencialmente instantâneos estáticos.
"Porque estamos estudando sistemas biológicos realmente desafiadores, o grupo muitas vezes tem que ser pioneiro em novos métodos estruturais também, "disse o pesquisador de pós-doutorado Steve Meisburger, Ph.D. '14, o autor principal do artigo. "Uma das questões que nos interessam desde o início é como os movimentos sutis de respiração de uma proteína dirigem a função bioquímica."
Os pesquisadores trouxeram seu projeto para a Fonte Síncrotron de Alta Energia Cornell (CHESS), onde eles aproveitaram o detector de matriz de pixels Pilatus 6M da instalação, o que lhes permitiu fazer imagens de alta resolução.
Para este trabalho, como na cristalografia regular, Os raios X foram emitidos em um cristal de amostra. O detector de matriz de pixels registrou a intensidade dos raios-X que foram difratados pelas proteínas do cristal, codificando assim a estrutura atômica. Qualquer distúrbio, ou seja, movimento - dentro do cristal fez com que fótons adicionais saltassem, criando um sinal de fundo muito fraco chamado espalhamento difuso. Essas informações são tradicionalmente descartadas durante o processamento de dados.
"Os fótons vão para todo lugar, e o sinal parece extremamente fraco porque está espalhado, "disse Ando, o autor sênior do artigo. "Por décadas, as pessoas não conseguiam medir com precisão, e eles não sabiam como interpretá-lo. "
Meisburger criou um software para processar cerca de 50 milhões de pontos de dados exclusivos, resultando em um mapa tridimensional de alta qualidade. Para surpresa dos pesquisadores, o mapa revelou que um componente significativo desse padrão de espalhamento difuso era, na verdade, resultado da vibração da estrutura da proteína. Este movimento oscilante era tão dominante, parecia obscurecer qualquer movimento dentro das proteínas, o que inicialmente foi uma decepção para os pesquisadores.
Mas depois de levar em conta essas vibrações de rede em simulações, os pesquisadores identificaram movimentos internos de proteínas também. Esses movimentos incluíam a abertura e o fechamento do sítio ativo da proteína.
"Imagine o cristal sendo como uma fileira de pessoas tentando andar juntas de mãos dadas, mas ao mesmo tempo, cada indivíduo pode estar fazendo algo ligeiramente diferente, "Ando disse." O sinal de todos se movendo juntos é dominante, então não podíamos discernir o sinal sutil que vinha dos indivíduos. Isso era algo que nunca tinha sido contabilizado. "
Esta nova abordagem para espalhamento difuso pode ajudar os pesquisadores a obter uma imagem mais clara da estrutura e dinâmica da proteína e, em última análise, uma melhor compreensão das reações bioquímicas.
"Nós realmente queremos levar isso em uma direção em que muitas pessoas possam usar a técnica e aprender algo novo sobre sua proteína, "Meisburger disse." Uma grande coisa sobre isso é que você consegue espalhamento difuso de graça sempre que você faz um experimento regular de cristalografia. Essa técnica realmente adiciona informações ao que você normalmente obteria. "