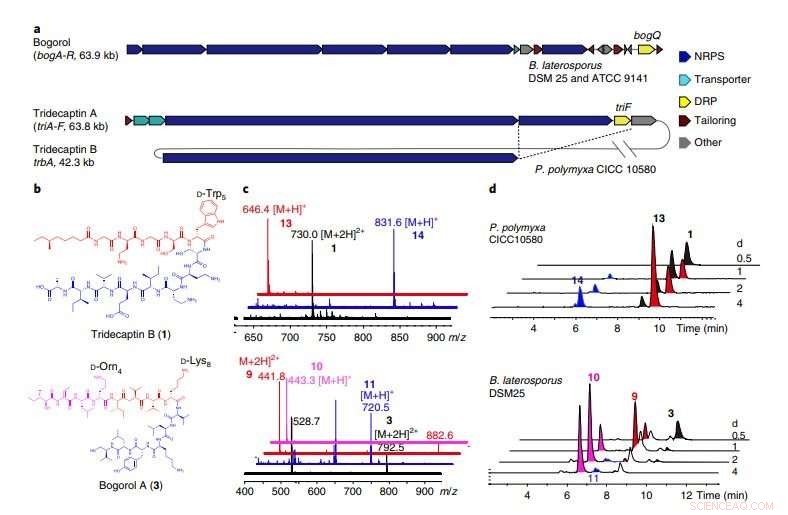
uma, Os BGCs de bogorol de B. laterosporus DSM 25 e ATCC 9141, bem como tridecaptina A e tridecaptina B de P. polymyxa CICC 10580. BogQ da cepa DSM 25 e da cepa ATCC 9141 compartilham 85 por cento de identidade de sequência de aminoácidos. As linhas pontilhadas que se cruzam indicam genes compartilhados por dois grupos de genes no mesmo hospedeiro (Fig. 10 suplementar). b, Estruturas de DNRPs tridecaptina B e bogorol A, com motivos de reconhecimento DRP destacados. c, Sobreposição empilhada do espectro de massa (ionização por electrospray) de compostos originais (preto), Fragmentos C-terminais (azul), e fragmentos N-terminais (vermelho). Os dados são representativos de dois experimentos independentes. Principal, tridecaptina B; fundo, bogorol A. d, Análises ao longo do tempo de compostos correspondentes produzidos por P. polymyxa CICC 10580 (topo) e B. laterosporus DSM 25 (parte inferior) em diferentes tempos de fermentação (representativos de três experimentos independentes). Crédito:Divisão de Ciências da Vida, HKUST
Antibióticos peptídicos não ribossomais, incluindo polimixina, vancomicina, e teixobactina, a maioria dos quais contém D-aminoácidos, são altamente eficazes contra bactérias multirresistentes. Contudo, O uso excessivo de antibióticos, ignorando o risco de surgimento de resistência, levou inexoravelmente ao surgimento generalizado de bactérias resistentes. Elucidar os mecanismos pouco conhecidos de resistência aos antibióticos peptídicos é crítico ao implementar antibióticos peptídicos e melhoraria efetivamente a eficiência.
Em um estudo recente, um grupo de cientistas da Universidade de Ciência e Tecnologia de Hong Kong revela a ampla distribuição e o potencial de resistência de amplo espectro das D-peptidases estereoespecíficas, fornecendo um potencial indicador precoce de resistência aos antibióticos a antibióticos peptídicos não ribossomais.
Suas descobertas foram publicadas no jornal Nature Chemical Biology em 26 de fevereiro, 2018.
"Aplicamos uma abordagem a 5, 585 genomas bacterianos completos abrangendo todo o domínio das bactérias, "disse Pei-Yuan Qian, professor catedrático da Divisão de Ciências da Vida, HKUST, e autor principal do artigo. "Com as análises químicas e enzimáticas subsequentes, demonstramos um mecanismo de resistência a antibióticos de peptídeos não ribossômicos que se baseia na clivagem hidrolítica por D-estereopeptidases específicas. "
A equipe identificou uma família de D-peptidases de resistência estereoespecífica (DRPs) que são filogeneticamente amplamente distribuídas na natureza. A descoberta de DRPs estava envolvida no combate a antibióticos amplamente distribuídos contendo D-aa para a sobrevivência de seu hospedeiro, que foi validado experimentalmente por uma combinação de edição de genes baseada em CRISPR / Cas9, análises químicas e enzimáticas.
"Dado o potencial dos DRPs para resistência de amplo espectro e seu potencial para direcionar antibióticos clinicamente importantes contendo D-aa, esses genes de resistência amplamente distribuídos são provavelmente particularmente perigosos se forem transferidos para patógenos oportunistas, "disse o Prof. Qian." A descoberta de DRPs na natureza constitui apenas a ponta do iceberg, que levará a pesquisas intensas sobre o uso e desenvolvimento de antibióticos peptídicos para combater a resistência aos antibióticos. "
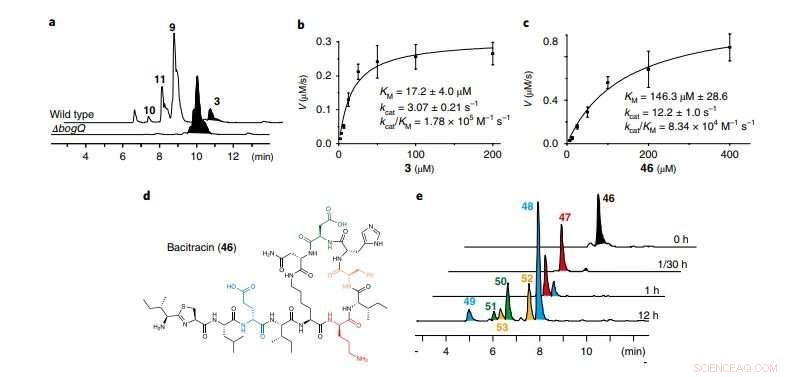
uma, Traços de LC-MS comparando B. laterosporus ATCC 9141 de tipo selvagem e o mutante ΔbogQ (representativo de três experimentos independentes). b, c, Análises cinéticas de hidrólise catalisada por BogQ de bogorol A (b; 3) e bacitracina (c; 46) v, velocidade da reação. Os dados são médios? ±? S.d .; n =? 3 experiências independentes. d, Estrutura do antibiótico bacitracina DNRP; as cores destacam os locais de clivagem de BogQ. e, Traços de LC-MS de ensaios in vitro de BogQ (2,0? ΜM) contra 46 (200? ΜM; representante de dois experimentos independentes). Produtos de clivagem de curso de tempo (47-53) de 46 são rotulados usando o mesmo código de cor que seus locais de clivagem d-aa em d. Para padrões de clivagem enzimática, veja a Fig Suplementar 22. Crédito:Divisão de Ciências da Vida, HKUST