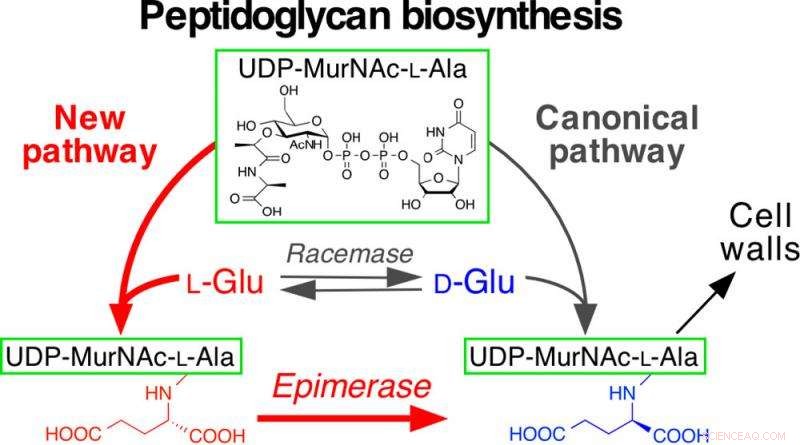
A via recém-descoberta envolve dois genes que facilitam a ligação de L-Glu a UDP-MurNAc-L-Ala e, em seguida, epimeriza (isomeriza) L-Glu a D-Glu. Crédito:Dairi T. et al., Journal of the American Chemical Society, 15 de março, 2017
Os cientistas descobriram uma via biossintética única para o D-Glutamato, um importante componente da parede celular em bactérias patogênicas, que pode levar ao desenvolvimento de medicamentos e produtos químicos agrícolas.
As paredes celulares (peptideglicano) são essenciais para que os microrganismos mantenham a integridade celular. A equipe liderada pelo professor Toru Dairi, da Escola de Graduação em Engenharia da Universidade de Hokkaido, focada na biossíntese de D-Glutamato (Glu), que é um componente das paredes celulares. Em geral, D-Glu é fornecido por uma enzima chamada Glu racemases. Mas a equipe descobriu, por meio de sua genômica comparativa de vários microrganismos, que a Xanthomonas oryzae - uma bactéria patogênica que faz com que as folhas das plantas de arroz fiquem brancas antes de matar a planta - não possui genes Glu racemase.
Para descobrir o que está compensando a peça que falta, os pesquisadores conduziram experimentos de clonagem shotgun nos quais os genes de X. oryzae foram introduzidos em mutantes de Escherichia coli que não podem produzir D-Glu. A equipe descobriu que os dois genes criaram um caminho que contornou o defeito, permitindo a produção de D-Glu. Em geral, D-Glu se liga a um complexo molecular denominado UDP-MurNAc-L-Ala, mas neste microorganismo patogênico, L-Glu se ligou ao complexo mediado por um dos dois genes que eles identificaram. O outro gene então epimeriza (isomeriza) L-Glu em D-Glu.
"Como X.oryzae, outras bactérias patogênicas, como Stenotrophomonas, que desencadeiam infecções hospitalares, também podem seguir o caminho recém-descoberto. Esperamos que o caminho forneça novos alvos para o desenvolvimento de medicamentos e produtos químicos agrícolas contra bactérias, "diz Toru Dairi.