Primeiros genomas de referência em nível cromossômico da banana ornamental e da banana rosa
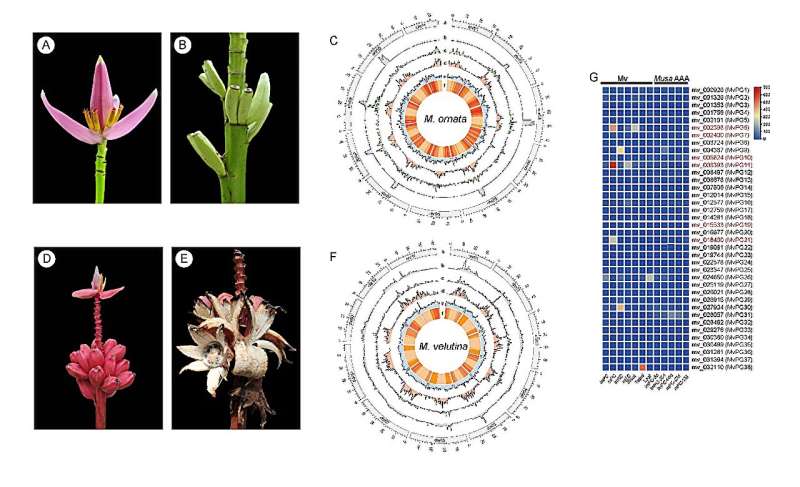 A e B:Flor e fruto de Musa ornata; D e E:Flor e fruto de Musa velutina . C e F:Características do genoma; G:Nível de expressão dos genes da poligalacturonase em diferentes tecidos e espécies. imPC:pericarpos imaturos; mPC:pericarpos maduros; imSC:sarcocarpos imaturos; mSC:sarcocarpos maduros; imPC-0d:pericarpos imaturos de frutos recém-emergindo do cacho; imPC-35d:pericarpos imaturos de frutos com 35 dias; imPC-60d:pericarpos imaturos de frutos com 60 dias; mPC-GM:pericarpos maduros de frutos verdes maduros; mPC-YM:pericarpos maduros de frutos amarelos maduros (6 dias após tratamento com etileno). Crédito:Os autores
A e B:Flor e fruto de Musa ornata; D e E:Flor e fruto de Musa velutina . C e F:Características do genoma; G:Nível de expressão dos genes da poligalacturonase em diferentes tecidos e espécies. imPC:pericarpos imaturos; mPC:pericarpos maduros; imSC:sarcocarpos imaturos; mSC:sarcocarpos maduros; imPC-0d:pericarpos imaturos de frutos recém-emergindo do cacho; imPC-35d:pericarpos imaturos de frutos com 35 dias; imPC-60d:pericarpos imaturos de frutos com 60 dias; mPC-GM:pericarpos maduros de frutos verdes maduros; mPC-YM:pericarpos maduros de frutos amarelos maduros (6 dias após tratamento com etileno). Crédito:Os autores O gênero Musa, abrangendo aproximadamente 70 espécies herbáceas, é encontrado predominantemente nas regiões tropicais e subtropicais da Ásia e Oceania. Este gênero é conhecido por ser uma das culturas alimentares mais importantes do mundo e por ser uma planta ornamental popular nos mercados.
Apesar das contribuições significativas feitas pelas tecnologias de sequenciação de terceira geração na produção de genomas de alta qualidade, continua a existir uma escassez de recursos genómicos para os cultivares de banana, os seus parentes selvagens e as espécies ornamentais dentro de Musa, o que dificultou a melhoria tanto da cultura como da ornamentação. variedades.
Musa ornata W. Roxburgh (Mo) e Musa velutina H. Wendl. &Drude (Mv) pertencem à seção Musa da família Musaceae. Eles estão intimamente relacionados com Musa acuminata e são nativos de Bangladesh, Mianmar e nordeste da Índia. Estas espécies não são apenas amplamente cultivadas como plantas ornamentais populares em regiões tropicais, mas também contribuem para a dieta local através dos seus frutos.
Portanto, Mo e Mv são candidatos desejáveis para sequenciamento do genoma de alta qualidade para melhorar o futuro melhoramento molecular. Neste estudo, os conjuntos de genoma em nível cromossômico de Mo e Mv foram gerados usando leituras longas de Oxford Nanopore e leituras de Hi-C. Os genomas de Mo e Mv foram montados em 11 pseudocromossomos com tamanhos de genoma de 427,85 Mb e 478,10 Mb, respectivamente.
Sequências repetitivas compreenderam 46,70% e 50,91% do total dos genomas de Mo e Mv, respectivamente. Avaliações de qualidade do genoma confirmaram a contiguidade (LAI:13,68 e 16,81), precisão (taxas de mapeamento de leituras da Illumina:95,55% e 94,29%) e integridade (BUSCO:98,08% e 98,51%) dos dois genomas.
De acordo com as previsões genéticas, um total de 39.177 e 31.256 genes codificadores de proteínas de alta confiança foram anotados para Mo e Mv, respectivamente. Em comparação com Musa acuminata e Mv, várias grandes inversões e translocações foram observadas em chr04 de Mo. Análises de enriquecimento de gene diferencialmente expresso (DEG) e Gene Ontology (GO) indicaram que genes regulados positivamente nos pericarpos maduros de Mv estavam principalmente associados ao metabolismo sacarídeo processos, particularmente na parede celular e na região extracelular.
Além disso, foram identificados vários genes de poligalacturonase (PG) que apresentaram níveis de expressão mais elevados em pericarpos maduros de Mv em comparação com outros tecidos, o que pode ser responsável pela deiscência do pericarpo. Além disso, este estudo também identificou genes associados à via de biossíntese de antocianinas.
Tomados em conjunto, os conjuntos do genoma de Mo e Mv em nível cromossômico fornecem informações valiosas sobre o mecanismo de deiscência do pericarpo e a biossíntese de antocianinas na banana, o que contribuirá significativamente para futuros esforços de melhoramento genético e molecular.
O artigo "Assembléias do genoma em nível cromossômico de Musa ornata e Musa velutina fornecem insights sobre a deiscência do pericarpo e a biossíntese de antocianina na banana" foi publicado em Horticulture Research .
