Os genes se organizam espacialmente para um splicing eficiente de mRNA, mostra o estudo
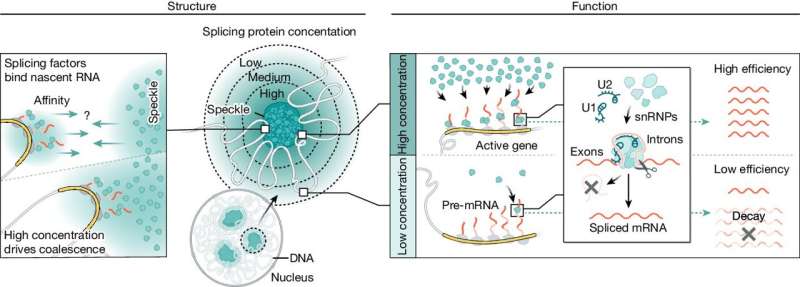 Modelo de como a organização do genoma 3D impulsiona o splicing de mRNA. Como os pré-mRNAs nascentes têm alta afinidade por fatores de splicing e como as regiões densas de PolII contêm as concentrações mais altas de pré-mRNAs nascentes, essas regiões genômicas podem alcançar contatos multivalentes com fatores de splicing que são enriquecidos em manchas nucleares. Como as manchas nucleares contêm a maior concentração desses fatores dentro do núcleo, esses contatos multivalentes podem conduzir à coalescência (automontagem) desses locais de DNA genômico com as manchas nucleares. Regiões genômicas e pré-mRNAs próximas a manchas nucleares apresentam níveis mais elevados de spliceossomos do que regiões mais distantes. A concentração local de pré-mRNAs, DNA genômico e spliceossomos em regiões proximais de speckle leva ao aumento da eficiência de splicing, enquanto um gene speckle-far transcrito no mesmo nível não é processado de forma tão eficiente. Modelo criado por Inna-Marie Strazhnik, Caltech. Crédito:Natureza (2024). DOI:10.1038/s41586-024-07429-6
Modelo de como a organização do genoma 3D impulsiona o splicing de mRNA. Como os pré-mRNAs nascentes têm alta afinidade por fatores de splicing e como as regiões densas de PolII contêm as concentrações mais altas de pré-mRNAs nascentes, essas regiões genômicas podem alcançar contatos multivalentes com fatores de splicing que são enriquecidos em manchas nucleares. Como as manchas nucleares contêm a maior concentração desses fatores dentro do núcleo, esses contatos multivalentes podem conduzir à coalescência (automontagem) desses locais de DNA genômico com as manchas nucleares. Regiões genômicas e pré-mRNAs próximas a manchas nucleares apresentam níveis mais elevados de spliceossomos do que regiões mais distantes. A concentração local de pré-mRNAs, DNA genômico e spliceossomos em regiões proximais de speckle leva ao aumento da eficiência de splicing, enquanto um gene speckle-far transcrito no mesmo nível não é processado de forma tão eficiente. Modelo criado por Inna-Marie Strazhnik, Caltech. Crédito:Natureza (2024). DOI:10.1038/s41586-024-07429-6 O núcleo de cada uma das suas células contém toda a informação genética (o genoma) necessária para construir cada tipo de célula e proteína em todo o seu corpo. Como uma biblioteca complexa num espaço minúsculo 50 vezes menor que a largura de um fio de cabelo humano, os genes são organizados em regiões precisas em três dimensões.
Além do genoma, o núcleo contém estruturas chamadas corpos nucleares que contêm altas concentrações de proteínas e ácidos nucléicos específicos. O papel dos corpos nucleares, no entanto, tem sido um mistério há quase um século. Um novo estudo do laboratório de Mitch Guttman, professor de biologia da Caltech, mostra que estes corpos nucleares podem agir como fábricas em miniatura para permitir a produção eficiente de mRNA.
O splicing do mRNA é uma parte fundamental do processo que ocorre quando as instruções codificadas do DNA são transformadas em uma proteína funcional. Depois que um gene é transcrito do DNA para o RNA, seções estranhas (chamadas íntrons) devem ser cortadas, um processo facilitado pelo splicing de enzimas.
No novo estudo, os pesquisadores do Caltech examinaram um tipo específico de corpo nuclear, chamado de mancha nuclear, que contém altas concentrações de enzimas de splicing. A equipe de Guttman descobriu que o genoma se desloca fisicamente, de modo que genes altamente transcritos ficam próximos de manchas, permitindo um splicing mais eficiente.
As descobertas têm implicações tanto para a compreensão científica fundamental da relação entre a estrutura nuclear e a função celular e, em última análise, para o futuro desenvolvimento terapêutico para doenças nas quais o splicing do mRNA é afetado, inclusive para condições neurodegenerativas como a ELA.
O artigo que descreve as descobertas foi publicado na revista Nature em 8 de maio. O título é "A organização do genoma em torno de manchas nucleares impulsiona a eficiência do splicing de mRNA".
Durante uma década, os pesquisadores do laboratório Guttman estudaram como o núcleo é organizado espacialmente – em outras palavras, o layout da biblioteca. As estruturas 3D nas quais o DNA é ordenado tornam certos genes mais ou menos acessíveis à maquinaria que converte o DNA em mRNA, e o novo estudo mostra que a estrutura física do genoma liga a transcrição ao processo de splicing.
Uma célula muscular, por exemplo, mudará o seu genoma de modo que genes altamente transcritos para a atividade muscular estejam fisicamente próximos de manchas nucleares, onde altas concentrações de enzimas de splicing tornam o splicing do RNA particularmente eficiente. Uma célula neuronal, por outro lado, irá reorientar o seu genoma no espaço para que os genes necessários para produzir células especializadas para a função neurológica estejam mais próximos das manchas.
“Sabemos que o splicing depende de enzimas, e qualquer mutação que interrompa a concentração de enzimas tem efeitos dramáticos”, diz Prashant Bhat, pesquisador associado de pós-doutorado em biologia e engenharia biológica e primeiro autor do estudo.
"Anteriormente, tendíamos a pensar no splicing como uma constante - você faz um pré-mRNA; você o une. Agora sabemos que a organização física do núcleo faz uma grande diferença na eficiência do splicing e, por extensão, de outros processos ."
Muitas doenças, incluindo doenças neurodegenerativas e cancro, têm uma característica comum:splicing disfuncional do ARN. Embora não se saiba se o splicing inadequado é a principal causa da doença ou um efeito, em última análise, o processo de splicing é um alvo promissor para a terapêutica.
Por exemplo, doenças nas quais é produzido um número inadequado de proteínas (conhecidas como doenças de haploinsuficiência) podem ser tratadas reposicionando a cópia normal do gene dessa proteína perto de manchas nucleares para aumentar o splicing e restaurar níveis saudáveis de proteínas.
