Resiliência genética e adaptação da avelã chinesa ameaçada de extinção
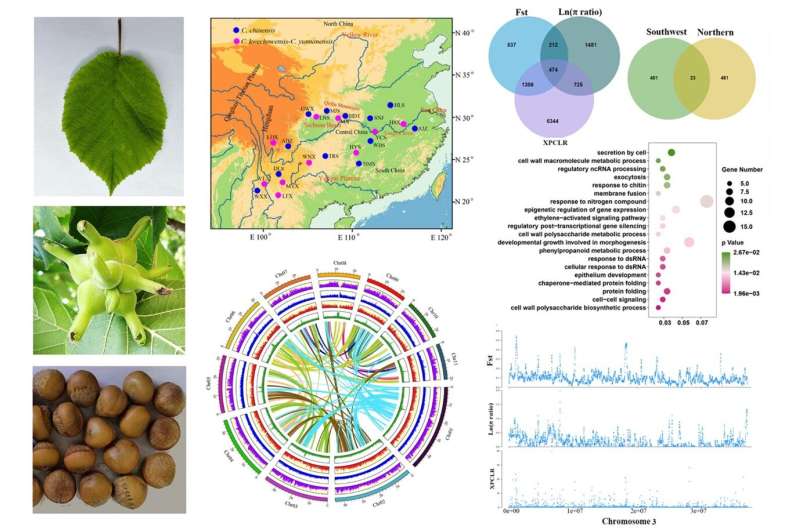 (A) Folha madura. (B) Frutos com invólucros bractados. (C) Sementes maduras. (D) Mapa mostrando a distribuição das 22 populações utilizadas neste estudo. (E) Gráfico Circos mostrando as características genômicas de C. chinensis. (F) Diagrama de Venn de PSGs:a esquerda indicava PSGs dentro de CCH_SW identificados pelos métodos FST, razão π e XP-CLR, e a direita indicava PSGs compartilhados entre CCH_SW e CCH_N. (G) Gráfico de dispersão de enriquecimento GO de PSGs em CCH_SW. (H) Assinaturas de seleção específicas para Chr03. Crédito:Yang, Z., et al.
(A) Folha madura. (B) Frutos com invólucros bractados. (C) Sementes maduras. (D) Mapa mostrando a distribuição das 22 populações utilizadas neste estudo. (E) Gráfico Circos mostrando as características genômicas de C. chinensis. (F) Diagrama de Venn de PSGs:a esquerda indicava PSGs dentro de CCH_SW identificados pelos métodos FST, razão π e XP-CLR, e a direita indicava PSGs compartilhados entre CCH_SW e CCH_N. (G) Gráfico de dispersão de enriquecimento GO de PSGs em CCH_SW. (H) Assinaturas de seleção específicas para Chr03. Crédito:Yang, Z., et al. As espécies ameaçadas são frequentemente caracterizadas por um grave declínio populacional ou mesmo por um estado de quase extinção durante as suas histórias demográficas. Tais ameaças resultam geralmente de alterações climáticas históricas e de atividades humanas.
Para populações pequenas e isoladas, a deriva genética pode levar à depressão por endogamia, à erosão da diversidade genética e à acumulação de mutações deletérias, o que diminui ainda mais o potencial adaptativo e aumenta o risco de extinção quando enfrenta habitats naturais ou climáticos voláteis. Elucidar a história demográfica das espécies ameaçadas e o impacto da erosão genómica no perigo é fundamental para implementar uma gestão e conservação eficazes.
Corylus chinensis (CCH), a avelã chinesa, foi amplamente distribuída pela Ásia durante o período terciário. No entanto, está agora ameaçado e distribuído apenas de forma disjuntiva nas montanhas médias e altas da China subtropical. Notavelmente, as suas características genéticas, história demográfica e potencial adaptativo permanecem em grande parte desconhecidos.
Para esse fim, uma equipe de pesquisadores da China gerou o primeiro genoma em nível de cromossomo do ameaçado C. chinensis, realizou estudos de genômica e genômica de conservação e comparou as assinaturas genômicas do colapso populacional, adaptação local e recuperação genética com sua disseminação simpátrica. C. kwechhowensis–C. complexo yunnanensis (CKY).
O estudo foi publicado na revista Plant Diversity .
“Encontramos grandes rearranjos do genoma em todas as espécies de Corylus e identificamos famílias de genes expandidos específicos de espécies em C. chinensis que podem estar envolvidas na adaptação”, diz o co-autor Zhaoshan Wang. "A genômica populacional revelou que tanto o CCH quanto o CKY divergiram em duas linhagens genéticas, formando um padrão consistente de diferenciação sudoeste-norte (SW vs. N)."
Especificamente, a inferência demográfica sugeriu que o tamanho da população das linhagens SW estreitas (CCH_SW e CKY_SW) diminuiu continuamente desde o final do Mioceno, enquanto as linhagens N generalizadas permaneceram estáveis (CCH_N) ou mesmo recuperadas (CKY_N) de gargalos populacionais durante o Quaternário.
Comparado com o CKY, o CCH mostrou diversidade genômica e heterozigosidade comparáveis, mas níveis mais elevados de endogamia medidos por séries de homozigosidade. No entanto, o CCH transportou significativamente menos mutações deletérias do que o CKY, uma vez que a selecção de purga mais eficaz reduziu a acumulação de variantes homozigóticas.
"Também detectamos sinais significativos de seleção positiva e introgressão adaptativa em diferentes linhagens, o que facilitou o acúmulo de variantes favoráveis e a formação de adaptação local. Portanto, ambos os tipos de seleção e introgressão exógena poderiam ter mitigado a endogamia e facilitado a sobrevivência e persistência do cruzamento CCH ", acrescenta Wang.
No geral, o estudo fornece insights críticos sobre a diferenciação da linhagem, adaptação local e potencial para recuperação futura de árvores ameaçadas.
