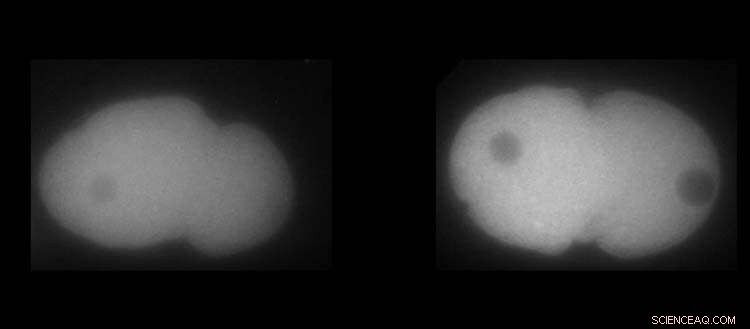
À esquerda, proteína MEX-5. À direita, proteína PLK-1 em embriões de C. elegans. O branco mostra a maior ou menor presença das proteínas. Crédito:UNIGE / Laboratoire Monica Gotta
Cada uma de nossas células contém cerca de 40 milhões de proteínas que, juntas, realizam todas as tarefas que a célula precisa para sobreviver. Para uma ação suave, as proteínas certas devem ser concentradas em quantidades específicas, em um momento específico e em um local específico. No entanto, estabelecer uma distribuição tão delicada requer um processo extremamente preciso, ocorrendo em pequenas resoluções espaciais que as ferramentas padrão de biologia celular geralmente não conseguem detectar. Para entender como esse mecanismo funciona, pesquisadores da Universidade de Genebra (UNIGE) desenvolveram uma nova abordagem combinando experimentos de genética e biologia celular com modelagem física. Usando algoritmos específicos, eles simularam a formação de gradientes de proteínas em 3D e ao longo do tempo e conseguiram explicar esses mecanismos complexos. Além disso, seu modelo inovador pode ser adaptado a outros sistemas de biologia para investigar a dinâmica de proteínas. Esses resultados podem ser lidos nos
Proceedings of the National Academy of Sciences .
Como uma gota de tinta em um copo de água, as proteínas podem se difundir e distribuir uniformemente por toda a célula. No entanto, para algumas tarefas, as proteínas precisam formar gradientes. "Gradientes de proteínas, que surgem da distribuição desigual de proteínas em áreas celulares específicas, são centrais para muitas funções celulares e do organismo", explica Monica Gotta, professora do Departamento de Fisiologia e Metabolismo Celular e do Centro de Pesquisa Translacional em Onco- hematologia (CRTOH) da Faculdade de Medicina da UNIGE, que dirigiu este trabalho. "Por exemplo, os gradientes de proteínas são importantes para a diferenciação celular, o processo pelo qual os diferentes tipos de células que constituem um organismo complexo emergem de uma única célula, o ovo fertilizado."
Um uso de aleatoriedade A proteína PLK-1, um regulador chave da divisão celular, é conhecida por estar mais concentrada no lado anterior do embrião. Mas como esse mecanismo pode ser implementado e qual seria a consequência se o menor detalhe desse errado? Como as ferramentas usuais da biologia não foram suficientes para responder a essa pergunta, Monica Gotta teve o prazer de receber em sua equipe uma física, Sofia Barbieri, pesquisadora de pós-doutorado no Departamento de Fisiologia Celular e Metabolismo da Faculdade de Medicina da UNIGE.
"Compilando todo o conhecimento sobre esse processo biológico e novas hipóteses sobre os mecanismos, desenvolvi um modelo estatístico de formação de gradiente proteico baseado em matemática probabilística", explica Sofia Barbieri. "Recorri a algoritmos computacionais específicos, chamados simulações de Monte Carlo, em homenagem à famosa cidade do jogo." Esses algoritmos são usados para modelar fenômenos com alto nível de complexidade, como finanças, negociação ou física de partículas.
A equipe conseguiu simular gradientes de proteínas, não apenas em 3D, mas também ao longo do tempo. Tal modelo exigiu, no entanto, várias iterações entre otimização de parâmetros e comparação com dados biológicos. Os pesquisadores construíram uma primeira versão do modelo incorporando todos os elementos físicos e biológicos conhecidos do sistema e, em seguida, introduziram parâmetros específicos necessários para testar várias hipóteses sobre as variáveis desconhecidas. Eles simularam possíveis resultados físicos e biológicos que reproduziam computacionalmente a dinâmica da proteína e o estabelecimento do gradiente na célula e os testaram na vida real com experimentos in vivo usando os embriões de um pequeno verme, o nematóide C. elegans.
Interações complexas de proteínas em jogo Graças à interação contínua entre modelagem e biologia celular, os pesquisadores da UNIGE foram capazes de determinar como o gradiente PLK-1 foi estabelecido e mantido ao longo do tempo. De fato, a PLK-1 deve se ligar e se desvincular dinamicamente da MEX-5, outra proteína crucial para o desenvolvimento do embrião de C. elegans, para neutralizar sua tendência natural de se difundir homogeneamente na célula. MEX-5 tem de fato a capacidade de alterar sua difusividade dependendo de sua posição dentro da célula e interagir com outras proteínas, o que é essencial para enriquecer PLK-1 quando necessário. "Mas, surpreendentemente, o MEX-5 não é tão eficiente em sua tarefa, pois uma grande quantidade de PLK-1 não está vinculada ao MEX-5", ressalta Sofia Barbieri.
Este estudo fornece um modelo quantitativo único para entender as interações dinâmicas entre proteínas e pode ser adaptado a outras células ou proteínas para as quais os mecanismos complexos não podem ser testados com experimentos usuais de biologia celular. "Nosso trabalho mostra que as colaborações interdisciplinares são cada vez mais importantes para avançar nas pesquisas", conclui Monica Gotta.
