Como um antibiótico de espectro estreito visa C. difficile
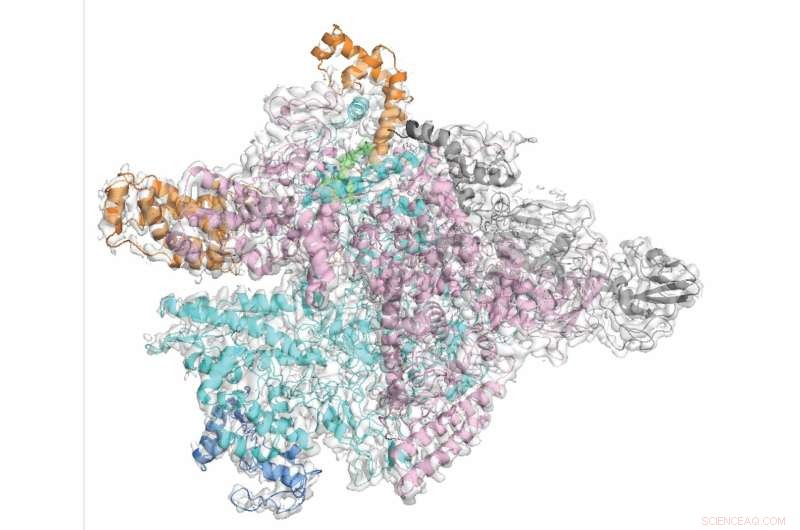
Os pesquisadores demonstraram em um nível molecular como a fidaxomicina atinge seletivamente o C. diff enquanto poupa as inocentes bactérias. Crédito:Universidade Rockefeller
A maioria dos antibióticos são facas de dois gumes. Além de matar o patógeno para o qual são prescritos, eles também dizimam bactérias benéficas e alteram a composição do microbioma intestinal. Como resultado, os pacientes tornam-se mais propensos à reinfecção e as cepas resistentes aos medicamentos têm maior probabilidade de surgir.
A resposta para esse problema pode ser antibióticos de espectro estreito que matam apenas uma ou algumas espécies de bactérias, minimizando o risco de danos colaterais. Em um estudo recente, os cientistas da Rockefeller examinaram de perto um desses antibióticos, a fidaxomicina, usada para tratar Clostridium difficile, ou C. diff, uma das infecções associadas à saúde mais comuns. Os pesquisadores demonstraram em um nível molecular como a fidaxomicina atinge seletivamente o C. diff enquanto poupa as inocentes bactérias.
As descobertas, detalhadas em
Nature , pode ajudar os cientistas na corrida para desenvolver novos antibióticos de espectro estreito contra outros patógenos.
"Quero que pessoas, cientistas e médicos pensem de maneira diferente sobre antibióticos", diz Elizabeth Campbell, professora associada de pesquisa do Rockefeller. “Como nosso microbioma é crucial para a saúde, as abordagens de espectro estreito têm um papel importante a desempenhar na forma como tratamos infecções bacterianas no futuro”.
Enigmáticamente seletivo C. diff é uma bactéria produtora de toxinas que pode inflamar o cólon e causar diarreia grave. Ele infecta cerca de meio milhão de pessoas nos Estados Unidos, principalmente em ambiente hospitalar, e cerca de um em cada 11 pessoas com mais de 65 anos que morrem em um mês.
Durante anos, os médicos usaram antibióticos de amplo espectro para tratar C. diff. A fidaxomicina é uma alternativa relativamente nova que recebeu aprovação da FDA em 2011.
Como vários outros antibióticos, incluindo o medicamento para tuberculose rifampicina, a fidaxomicina tem como alvo uma enzima chamada RNA polimerase (RNAP), que a bactéria usa para transcrever seu código de DNA em RNA. Para entender exatamente por que a fidaxomicina inibe seletivamente a RNAP em C. diff e não na maioria das outras bactérias, Campbell juntou-se ao bioquímico Robert Landick da Universidade de Wisconsin-Madison para visualizar C. diff RNAP usando microscopia crioeletrônica, uma poderosa técnica de imagem que pode revelar a forma 3D das moléculas e capturar a molécula da droga e seu alvo em ação. "Embora a arquitetura geral do RNAP em diversas bactérias seja semelhante, ainda existem diferenças consideráveis", diz Campbell.
Espionando RNAP Um grande desafio, no entanto, foi primeiro produzir grandes quantidades de C. diff, um germe anaeróbico que não cresce na presença de oxigênio. O primeiro autor do estudo, Xinyun Cao, do Landick Lab, passou dois anos desenvolvendo um sistema para produzir mais facilmente C. diff RNAP usando E. Coli, uma bactéria de fácil crescimento frequentemente usada no laboratório.
Usando este material, o co-primeiro autor Hande Boyaci, um pós-doc na equipe de Campbell, gerou imagens de C. diff RNAP bloqueado com fidaxomicina em resolução quase atômica. Presos em uma dobradiça entre duas subunidades de RNAP, os congestionamentos de fidaxomicina abrem a pinça da enzima, impedindo que ela se agarre ao material genético e inicie o processo de transcrição.
Ao examinar de perto os pontos de contato entre RNAP e fidaxomicina, os pesquisadores identificaram um aminoácido no RNAP que se liga à fidaxomicina, mas está ausente nos principais grupos de micróbios intestinais que são poupados pela fidaxomicina. Uma versão geneticamente alterada de C. diff que não possuía esse aminoácido não era perturbada pela fidaxomicina, assim como outras bactérias comensais no intestino. Por outro lado, as bactérias que o adicionaram ao seu RNAP tornaram-se sensíveis à fidaxomicina.
As descobertas sugerem que este um aminoácido entre os 4.000 aminoácidos desta máquina de transcrição robusta e essencial é o calcanhar de Aquiles, responsável pela morte das bactérias pela fidaxomicina.
A abordagem usada neste estudo propõe um roteiro para o desenvolvimento de antibióticos novos e mais seguros, dizem os pesquisadores. Ao elucidar ainda mais a estrutura RNAP de diversas bactérias, os cientistas podem projetar antibióticos que visam cada patógeno de forma mais seletiva e eficaz.
