
Modelo molecular mostrando dCas9 ligado a uma molécula de fusão RNA-RNA origami guia que traz fatores de transcrição para uma sequência promotora. Crédito:Cody Geary, Universidade de Aarhus
O desenvolvimento de ferramentas para controle preciso de processos biológicos tem sido um dos principais pilares do agora maduro campo da biologia sintética. Essas ferramentas científicas emprestam princípios de uma infinidade de campos de pesquisa que, quando combinados, permitem aplicações únicas e potencialmente transformadoras para a sociedade moderna.
Traduzir as inovações modernas da nanotecnologia de RNA no contexto biológico possui imenso potencial devido à compatibilidade com o dobramento e expressão nas células, mas também impõe desafios únicos, como condições de desempenho rígidas e instabilidade inerente das moléculas de RNA.
No entanto, uma abordagem de design de RNA estrutural recente desenvolvida no laboratório Andersen, denominada "RNA origami", está tentando resolver isso. Essa abordagem tenta gerar dispositivos complexos baseados em RNA feitos pelo homem que são estáveis nas células, interagem com outras biomoléculas, incluindo outros RNA e proteínas, e permitem aplicações únicas, particularmente no contexto da regulação gênica.
Demonstrado por duas abordagens distintas recentemente publicadas em
Nucleic Acids Research , o origami de RNA é apresentado como uma plataforma sofisticada de design de RNA que, quando aplicada no contexto celular, gera moléculas únicas para regulação baseada em biologia sintética.
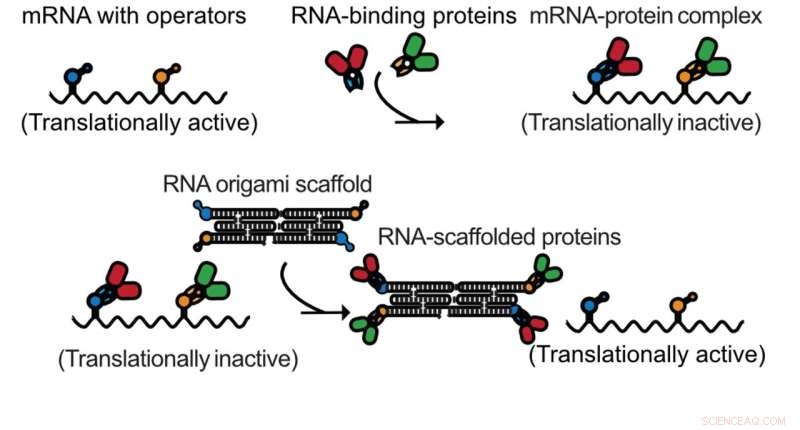
Um mRNA com operadores é inibido pelas proteínas que expressam. As moléculas de origami de RNA servem como esponjas que se ligam às proteínas e tornam os mRNAs translacionalmente ativos novamente. Crédito:ACS Synthetic Biology (2022)
As esponjas de RNA regulam a produção de enzimas em bactérias Na primeira abordagem, o origami de RNA foi usado para obter um controle preciso dos níveis de produção de proteínas quando expressos em bactérias. Cassetes de expressão de proteína autoinibidora foram feitos instalando um sítio de ligação forte para a proteína expressa em seu próprio gene. Depois, origami de RNA decorado com os mesmos sítios de ligação a proteínas foi expresso em grande excesso.
Desta forma, o origami de RNA serve como uma esponja de proteína que sequestra proteínas na célula e permite a expressão da proteína auto-inibida. Este conceito geral foi mostrado para permitir a regulação de várias proteínas simultaneamente e ativar as vias enzimáticas para melhores rendimentos do produto.
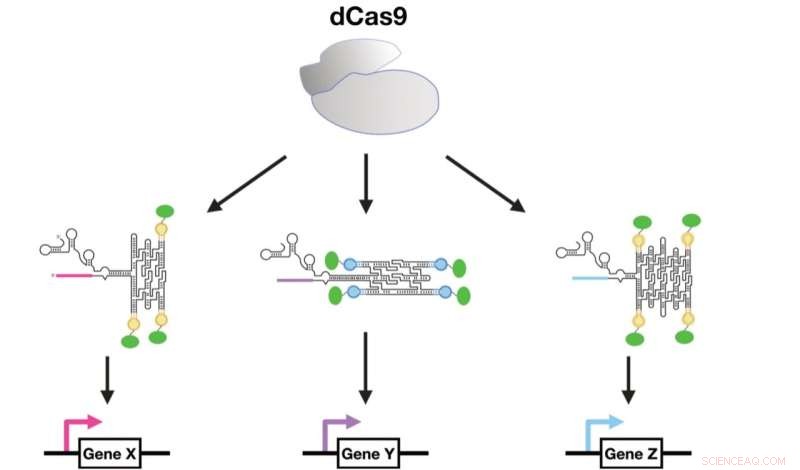
CRISPR-dCas9 funciona como um regulador mestre de moléculas de fusão sgRNA – RNA origami que trazem fatores de transcrição para uma sequência promotora. Gráficos por George Pothoulakis. Crédito:Pesquisa de Ácidos Nucleicos (2022). DOI:10.1093/nar/gkac470
Reguladores baseados em CRISPR para fábricas de produtos químicos de levedura Na segunda abordagem, o origami de RNA foi combinado com CRISPR, uma das técnicas modernas de biologia molecular mais populares, para regular a expressão gênica em leveduras. Os origamis de RNA foram integrados nos pequenos RNAs que guiam o CRISPR-Cas9 para direcionar sequências específicas no genoma de DNA.
Os andaimes de origami de RNA foram decorados com sítios de ligação a proteínas capazes de recrutar fatores de transcrição. Ao direcionar os andaimes de RNA para regiões promotoras, os fatores de transcrição ativaram a expressão gênica. Foi demonstrado que a força de expressão pode ser ajustada pela orientação do scaffold e pela quantidade de fatores de transcrição recrutados. Finalmente, foi demonstrado que as vias multienzimáticas podem ser controladas para a produção de alto rendimento da droga anticâncer violaceína.
+ Explorar mais Sistema de controle de qualidade de proteínas sintéticas em bactérias


