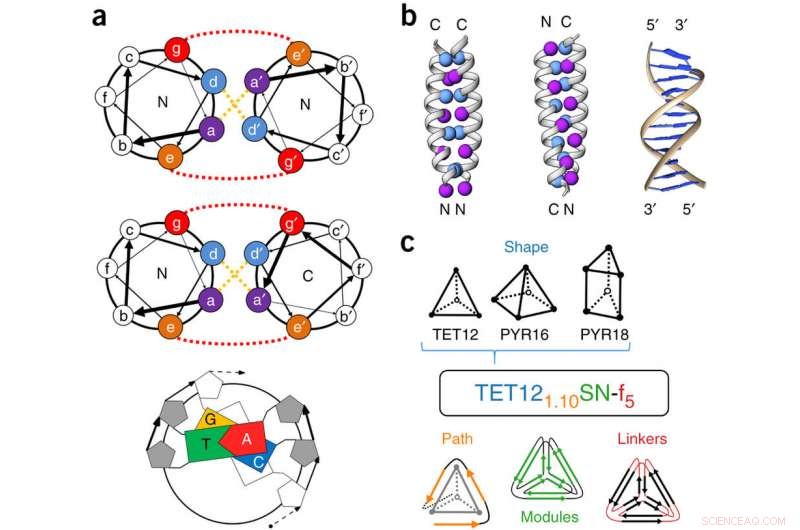
 p Estrutura do módulo CC e nomenclatura CCPO. (a) Coiled-coils (CC) são definidos por uma unidade de repetição regular de sete resíduos de aminoácidos, rotulado como, b, c, d, e, f, ou g (uma repetição do heptal). A especificidade da interação é codificada por interações hidrofóbicas nas posições a e d (linhas tracejadas amarelas) e interações eletrostáticas entre as posições eeg (linhas tracejadas vermelhas). As regras de complementaridade do DNA são comparativamente simples, uma vez que apenas quatro elementos de construção estão presentes. (b) Dímeros CC podem emparelhar em uma orientação paralela ou antiparalela. As posições a e d são mostradas como esferas. O DNA de fita B pode emparelhar apenas em uma orientação antiparalela. (c) Um exemplo de nome (TET12 1,10 SN-f 5 ) é mostrado no meio do painel. Os nomes completos dos poliedros são definidos pelo tipo de poliedro (TET =tetraedro; PYR =pirâmide; TRIP =prisma trigonal) seguido pelo número de segmentos CC (mostrado em azul). O subscrito (em laranja) denota a topologia e a permutação circular de cada poliedro, ou seja, o caminho duplo euleriano que a cadeia polipeptídica faz sobre o poliedro. Os próximos rótulos (verde) denotam o tipo de módulos CC usados (S =solúvel; SN =solúvel, cobrado negativamente), tipo de linker (f =flexível; c =carregado), e, na forma subscrita (vermelho), o comprimento do vinculador. Nos casos em que duas variantes têm o mesmo nome (por exemplo, pedidos diferentes de módulos CC), as letras b, c, d, e assim por diante são anexados. Os poliedros mais extensivamente caracterizados são referidos por nomes mais curtos:TET12SN (TET12 1,10 SN-f5), TET12S (TET12 1,10 S-f 5 ), TET12 SScr (TET12ScrS-f5), PYR16SN (PYR16 4,6 SN-f 5 ), e TRIP18SN (TRIP187.5RSN-f5). Crédito: Nature Biotechnology (2017). DOI:10.1038 / nbt.3994
p Estrutura do módulo CC e nomenclatura CCPO. (a) Coiled-coils (CC) são definidos por uma unidade de repetição regular de sete resíduos de aminoácidos, rotulado como, b, c, d, e, f, ou g (uma repetição do heptal). A especificidade da interação é codificada por interações hidrofóbicas nas posições a e d (linhas tracejadas amarelas) e interações eletrostáticas entre as posições eeg (linhas tracejadas vermelhas). As regras de complementaridade do DNA são comparativamente simples, uma vez que apenas quatro elementos de construção estão presentes. (b) Dímeros CC podem emparelhar em uma orientação paralela ou antiparalela. As posições a e d são mostradas como esferas. O DNA de fita B pode emparelhar apenas em uma orientação antiparalela. (c) Um exemplo de nome (TET12 1,10 SN-f 5 ) é mostrado no meio do painel. Os nomes completos dos poliedros são definidos pelo tipo de poliedro (TET =tetraedro; PYR =pirâmide; TRIP =prisma trigonal) seguido pelo número de segmentos CC (mostrado em azul). O subscrito (em laranja) denota a topologia e a permutação circular de cada poliedro, ou seja, o caminho duplo euleriano que a cadeia polipeptídica faz sobre o poliedro. Os próximos rótulos (verde) denotam o tipo de módulos CC usados (S =solúvel; SN =solúvel, cobrado negativamente), tipo de linker (f =flexível; c =carregado), e, na forma subscrita (vermelho), o comprimento do vinculador. Nos casos em que duas variantes têm o mesmo nome (por exemplo, pedidos diferentes de módulos CC), as letras b, c, d, e assim por diante são anexados. Os poliedros mais extensivamente caracterizados são referidos por nomes mais curtos:TET12SN (TET12 1,10 SN-f5), TET12S (TET12 1,10 S-f 5 ), TET12 SScr (TET12ScrS-f5), PYR16SN (PYR16 4,6 SN-f 5 ), e TRIP18SN (TRIP187.5RSN-f5). Crédito: Nature Biotechnology (2017). DOI:10.1038 / nbt.3994
p (Phys.org) —Uma grande equipe de pesquisadores com membros da Eslovênia, o Reino Unido, Sérvia, A França e a Espanha desenvolveram uma técnica que faz com que as proteínas se automontem em formas geométricas sob demanda. Em seu artigo publicado na revista
Nature Biotechnology, o grupo descreve sua técnica e possíveis usos para as minúsculas gaiolas. p Nos últimos anos, cientistas manipularam fitas de DNA para fazer com que se unissem em formas úteis (origami de DNA). Neste novo esforço, os pesquisadores fizeram algo semelhante usando proteínas. Os profissionais da área acreditam que tais objetos podem ser úteis para aplicações como a construção de pacotes para entrega de drogas em locais específicos do corpo humano.
p Para fazer com que as proteínas se automontem, os pesquisadores torceram fitas duplas de seções de aminoácidos (bobinas enroladas) em fitas de aminoácidos que foram colocadas em seções cortadas de fitas de proteínas de ocorrência natural. Os pesquisadores descrevem as bobinas enroladas como semelhantes a segmentos de fio. As seções que foram introduzidas na corrente foram escolhidas especificamente porque testes anteriores mostraram que elas se flexionariam da maneira desejada quando expostas a fatores como eletricidade ou água. O resultado foi um único fio com dobras que faziam com que o fio tomasse a forma de um objeto geométrico. A equipe relata que eles foram capazes de criar tetraedros, pirâmides de quatro lados e prismas triangulares. Os testes das estruturas mostraram que são solúveis em soluções aquosas e que podem assumir suas formas dentro e fora das células. A equipe também mostrou que as estruturas podem ser formadas e possivelmente usadas em ratos vivos.
p Os pesquisadores observam que parâmetros como a carga das bobinas enroladas e a capa que se forma em suas extremidades podem ser ajustados para criar diferentes formas e ditar as condições sob as quais as proteínas se auto-montam. Eles ainda relatam que foi possível criar uma caixa de ferramentas de blocos de construção das estruturas, permitindo maior, estruturas mais complexas das formas básicas. Eles sugerem que tais construções podem ser usadas para transportar drogas ou vacinações através do corpo ou para criar outras estruturas que incorporem a funcionalidade de aminoácidos. p © 2017 Phys.org
