A pesquisa combina origami de DNA e fotolitografia para dar um passo mais perto dos computadores moleculares
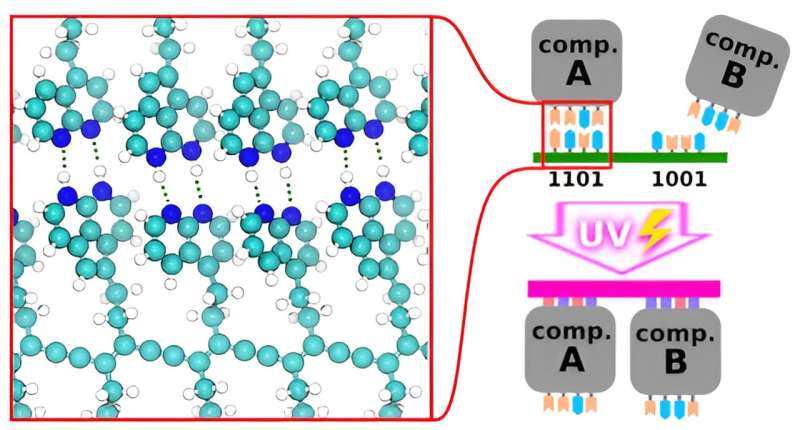 Resumo gráfico. Crédito:ACS Nano (2024). DOI:10.1021/acsnano.3c10575
Resumo gráfico. Crédito:ACS Nano (2024). DOI:10.1021/acsnano.3c10575 Os componentes moleculares dos computadores poderão representar uma nova revolução informática e ajudar-nos a criar computadores mais baratos, mais rápidos, mais pequenos e mais potentes. No entanto, os investigadores lutam para encontrar formas de os montar de forma mais fiável e eficiente.
Para ajudar a conseguir isso, cientistas do Instituto de Física da Academia Tcheca de Ciências investigaram as possibilidades de automontagem de máquinas moleculares com base em soluções aprimoradas pela evolução natural e usando sinergia com a fabricação atual de chips.
Há um limite para a miniaturização dos atuais chips de computador baseados em silício. A electrónica molecular, utilizando interruptores e memórias do tamanho de uma única molécula, poderia proporcionar uma revolução no tamanho, velocidade e capacidades dos computadores, ao mesmo tempo que reduzia o seu crescente consumo de energia, mas a sua produção em massa é um desafio. A nanofabricação e montagem dos componentes em larga escala, com poucos defeitos e acessíveis permanece indefinida. A inspiração retirada da natureza viva poderia mudar este status quo.
Pequenos protótipos de circuitos moleculares compostos por algumas moléculas estão sendo produzidos atualmente por microscopia de varredura por sonda, que os manipula uma molécula por vez por meio de um cantilever macroscópico lento e pesado.
Prokop Hapala, que liderou o estudo publicado no ACS Nano , compara isso à construção de um delicado mosaico usando um enorme guindaste, um ladrilho de cada vez. A automontagem poderia resolver este problema, mas cria outros desafios. Por exemplo, como produzimos uma variedade de estruturas quando apenas uma pequena quantidade de informação estrutural pode ser codificada em interações entre alguns grupos funcionais?
Pesquisadores do Instituto de Física da Academia Tcheca de Ciências inspiraram-se na natureza, onde componentes funcionais e estruturais são dissociados em modelos de polímeros como DNA ou RNA. Lá, os açúcares-fosfatos representam a estrutura e as nucleobases, ligadas por ligações de hidrogênio, fornecem o armazenamento da informação.
Graças a estas ligações, estes polímeros informativos podem auto-montar-se em formas complexas e conduzir a auto-replicação ou síntese de outras moléculas mais pequenas. Essa abordagem já foi utilizada no "origami de DNA", que pode produzir moléculas complexas com as formas e funções desejadas. Mas como podemos ampliar o processo e alcançar maior variedade?
“Pares de bases de DNA conhecidos – que ingenuamente se pode pensar que seriam a melhor escolha – não podem ser usados como estão”, explica Paolo Nicolini, um dos autores. “Eles funcionam muito bem na célula, mas isso se deve ao ambiente e ao restante da maquinaria celular. Em condições compatíveis com a nanofabricação, eles simplesmente não são seletivos o suficiente”.
Mithun Manikandan, Paolo Nicolini e Prokop Hapala decidiram combinar as possibilidades oferecidas pelo origami de DNA e pela fotolitografia para criar estruturas complexas de chips contemporâneos. Isto poderia abrir caminho à produção em massa de circuitos moleculares revolucionários integrados com a tecnologia contemporânea de fabrico de chips – algo que poderia permitir uma transição suave da actual maquinaria informática para o próximo nível.
Para permitir isso, os pesquisadores propuseram a substituição da estrutura açúcar-fosfato por diacetileno fotossensível. Eles usaram simulações detalhadas para rastrear grupos finais complementares ligados a hidrogênio que conduziriam a automontagem em uma rede sob as condições usadas na produção de chips.
Derivados de diacetileno foram usados como base porque podem polimerizar eficientemente sob essas condições quando estimulados por luz UV ou injeção de elétrons, e unidades análogas às bases de DNA/RNA (as "letras" do código genético) foram investigadas in silico como grupos finais conduzindo a montagem dos componentes nas formas pretendidas.
O objectivo era encontrar pares complementares, onde duas unidades se ligassem de forma fiável uma à outra e não a outras unidades – esta característica, novamente análoga à forma como o ADN funciona, permitiria a criação de padrões de circuitos complexos determinísticos. Os pesquisadores descobriram que as unidades contendo grupos finais doadores de hidrogênio puro eram especialmente adequadas. Foram encontradas dezesseis unidades candidatas promissoras, abrindo caminho para pesquisas experimentais e eventuais aplicações industriais.
Os resultados têm implicações interessantes para a computação de DNA e análogos artificiais de DNA. Os alfabetos de quatro letras mais viáveis encontrados na triagem ocorreram em uma região muito estreita de energias de ligação de 15 a 25 kcal/mol, e todos dependiam de um pequeno subconjunto dos grupos finais testados.
Embora apenas um pequeno subconjunto do possível espaço de letras pudesse ser testado com alta precisão, isto sugere que o alfabeto do DNA pode não ser apenas o resultado de um “acidente congelado no tempo”, mas poderia ter sido uma opção estável e energeticamente favorável. Nenhum alfabeto de seis letras foi encontrado no espaço testado, mas novos mecanismos de seletividade e ligações não covalentes diferentes das ligações de hidrogênio (como ligações de halogênio) poderiam potencialmente habilitá-los. De maneira semelhante, as possibilidades de análogos terapêuticos e farmacêuticos de DNA poderiam ser testadas.
Este trabalho vai melhorar ainda mais a disponibilidade sintética das moléculas e superar limitações experimentais. Embora a maioria de nós provavelmente esteja lendo isso sobre máquinas que dependem de transistores baseados em silício, em breve poderemos começar a fazer uma transição suave para máquinas que usam parcialmente nanoeletrônica molecular. Este trabalho representa mais um passo em direção a esse futuro.
