Microscopia de força atômica de alta velocidade assume proteínas intrinsecamente desordenadas
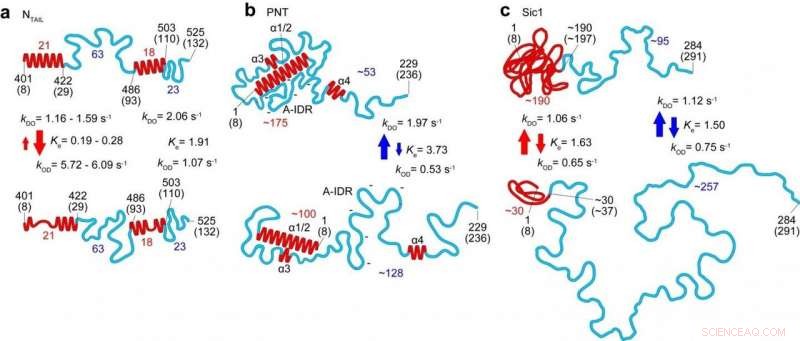 p Características estruturais e dinâmicas de três IDPs (N CAUDA , PNT e Sic1) revelado pela imagem HS-AFM. Os painéis superior e inferior correspondem aos estados mais ordenado e menos ordenado, respectivamente. Os números em vermelho e azul representam o número de aminoácidos contidos nas respectivas regiões dobradas e regiões totalmente desordenadas, respectivamente. As setas vermelha e azul indicam a natureza cinética das mudanças na altura das regiões dobradas e na distância de ponta a ponta de regiões totalmente desordenadas, respectivamente. Crédito:Kanazawa University
p Características estruturais e dinâmicas de três IDPs (N CAUDA , PNT e Sic1) revelado pela imagem HS-AFM. Os painéis superior e inferior correspondem aos estados mais ordenado e menos ordenado, respectivamente. Os números em vermelho e azul representam o número de aminoácidos contidos nas respectivas regiões dobradas e regiões totalmente desordenadas, respectivamente. As setas vermelha e azul indicam a natureza cinética das mudanças na altura das regiões dobradas e na distância de ponta a ponta de regiões totalmente desordenadas, respectivamente. Crédito:Kanazawa University
p Nossa compreensão das proteínas biológicas nem sempre se correlaciona com o quão comuns ou importantes elas são. Metade de todas as proteínas, moléculas que desempenham um papel fundamental nos processos celulares, são intrinsecamente desordenados, o que significa que muitas das técnicas padrão para sondar biomoléculas não funcionam com elas. Agora, pesquisadores da Universidade de Kanazawa, no Japão, demonstraram que sua tecnologia de microscopia de força atômica de alta velocidade desenvolvida em casa pode fornecer informações não apenas sobre as estruturas dessas proteínas, mas também sobre sua dinâmica. p Entender como uma proteína é formada fornece pistas valiosas sobre suas funções. O desenvolvimento da cristalografia de proteínas nas décadas de 1930 e 1950 trouxe várias estruturas de proteínas à vista pela primeira vez, mas tornou-se gradualmente aparente que uma grande fração das proteínas carece de uma estrutura única, tornando-as intratáveis à cristalografia de raio-x. Como eles são muito finos para a microscopia eletrônica, as únicas alternativas viáveis para muitas dessas proteínas intrinsecamente desordenadas (IDPs) são a imagem por ressonância magnética nuclear e o espalhamento de raio X de pequeno ângulo. Os dados coletados a partir dessas técnicas são calculados em conjuntos e, portanto, não fornecem uma indicação clara das conformações de proteínas individuais ou com que frequência elas ocorrem. A microscopia de força atômica, por outro lado, é capaz de gerar imagens biológicas de resolução em nanoescala em alta velocidade, para que ele possa capturar dinâmicas, bem como estruturas de proteínas.
p Neste último trabalho, pesquisadores da Universidade de Kanazawa ao lado de colaboradores no Japão, França e Itália aplicaram a técnica ao estudo de vários deslocados internos e identificaram parâmetros que definem a forma, tamanho e comprimento da cadeia de regiões de proteína, bem como uma lei de potência relacionando o tamanho da proteína ao comprimento da proteína, e uma descrição quantitativa do efeito da superfície da mica nas dimensões da proteína. A dinâmica das conformações da proteína capturada graças às capacidades de alta velocidade da técnica revelou glóbulos que aparecem e desaparecem, e transformações entre conformações totalmente não estruturadas e fracamente dobradas em segmentos de até 160 aminoácidos de comprimento.
p Os estudos da nucleoproteína do vírus do sarampo, em particular, ajudaram a identificar não apenas a forma e as dimensões, mas também as características das transições ordem-distúrbio na região responsável pelo reconhecimento molecular, que permite que os vírus identifiquem os fatores do hospedeiro para que possam se reproduzir. Eles também poderiam determinar estruturas em escala maior da fosfoproteína do vírus que não são acessíveis à ressonância magnética nuclear (que só pode dar uma indicação das distâncias entre aminoácidos separados por menos de 2 nm). Os pesquisadores sugerem que a formação de certas formas compactas observadas pode explicar a resistência à proteólise - quebra de proteínas.
p Em seu relatório do trabalho, os pesquisadores destacam que, além de uma ferramenta poderosa por si só, "Quando todas as características moleculares reveladas por HS-AFM são combinadas com a estrutura local dobrada dada por NMR, as informações combinadas permitem um delineamento quantitativo dos caracteres estruturais e dinâmicos dos deslocados internos, de uma forma mais realista em comparação com as imagens representadas individualmente, como demonstrado para PNT [fosfoproteína do vírus do sarampo]. "
p
Microscopia de força atômica de alta velocidade
p A microscopia de força atômica foi desenvolvida na década de 1980 e trouxe a resolução em escala atômica alcançada pela microscopia de tunelamento de varredura (que ganhou o Prêmio Nobel de Física de 1986) para amostras não condutoras. Ele funciona usando um pequeno cantilever com uma ponta em nanoescala no final, que ou sente a superfície como uma agulha de disco de vinil ou bate nela. Seja ajustando a altura da ponta ou a frequência ressonante do toque, as interações entre a ponta e a superfície fornecem um sinal que pode ser usado para gerar uma imagem.
p Enquanto as imagens AFM trouxeram enormes benefícios para a pesquisa biológica, these studies were able to move up a gear again when Toshio Ando and his team at Kanazawa University reported an atomic force microscope that operated at high speed. Atomic scale resolution images became movies bringing not just structures but also dynamics within grasp. Previous work on ordered proteins, which are reasonably well understood, as well as the IDP facilitates chromatin transcription (FACT) protein, has established that the technique can be used to image these biomolecules without effects from contact between tip and sample distorting the data.
p
Intrinsically disordered proteins
p The arrival of xray crystallography gave researchers a clear view of vast numbers of biomolecule structures for the first time. But with the hundreds of thousands of biomolecule structures analyzed using protein crystallography since the technique first came into use in the 1930s and 1950s, a mounting body of evidence began to build that not all proteins have a single set structure. The observations ran counter to the prevailing paradigm of protein function determined by a fixed structure.
p Over the past ten to twenty years the ubiquity of these intrinsically disorderd proteins and their importance in cell functions from signaling to the regulation of transcription and subsequent translation has become widely recognized. In the current work the researchers study IDPs including polyglutamine tract binding protein-1 (PQBP-1, involved in different processes, such as pre-mRNA splicing, transcription regulation, innate immunity and neuron development), autophagy proteins (which are invovolved in removing dysfunctional cell components) containing intrinsically disordered regions (IDRs) and the measles virus nucleoprotein.
