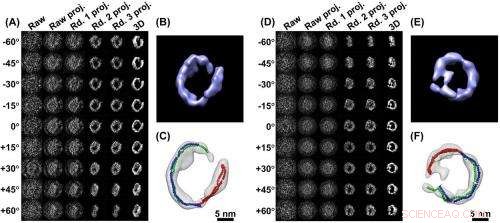
 p Imagens 3-D de uma única partícula (A) uma série de imagens de uma partícula de proteína ApoA-1, tomadas de diferentes ângulos, conforme indicado. Uma sucessão de quatro aprimoramentos de computador (projeções) esclarece o sinal. Na coluna da direita está a imagem 3-D compilada a partir dos dados esclarecidos. B) é um close-up da imagem 3-D reconstruída. C) A análise mostra como a estrutura da partícula é formada por três proteínas ApoA-1 (vermelho, verde, modelos semelhantes a macarrão azul)
p Imagens 3-D de uma única partícula (A) uma série de imagens de uma partícula de proteína ApoA-1, tomadas de diferentes ângulos, conforme indicado. Uma sucessão de quatro aprimoramentos de computador (projeções) esclarece o sinal. Na coluna da direita está a imagem 3-D compilada a partir dos dados esclarecidos. B) é um close-up da imagem 3-D reconstruída. C) A análise mostra como a estrutura da partícula é formada por três proteínas ApoA-1 (vermelho, verde, modelos semelhantes a macarrão azul)
p (PhysOrg.com) - Quando Gang Ren gira os controles de seu microscópio crio-eletrônico, ele compara isso a ajustar a marcha e os freios de uma bicicleta de corrida. Mas esta máquina no Laboratório Nacional Lawrence Berkeley (Laboratório de Berkeley) do Departamento de Energia dos EUA (DOE) é um pouco mais complexa. Custa quase US $ 1,5 milhão, opera na temperatura fria do nitrogênio líquido, e está permitindo que os cientistas vejam o que ninguém viu antes. p Na Fundição Molecular, Aclamado centro de pesquisa em nanotecnologia do Berkeley Lab, Ren levou seu microscópio Zeiss Libra 120 Cryo-Tem a resoluções nunca imaginadas por seus fabricantes alemães, produzindo instantâneos detalhados de moléculas individuais. Hoje, ele e seu colega Lei Zhang estão relatando as primeiras imagens 3-D de uma proteína individual já obtidas com clareza suficiente para determinar sua estrutura.
p Os cientistas criam rotineiramente modelos de proteínas usando difração de raios-X, ressonância magnética nuclear, e imagem de microscópio crioeletrônico convencional (cryoEM). Mas esses modelos requerem uma "média" de computador de dados de análise de milhares, ou mesmo milhões de moléculas semelhantes, porque é muito difícil determinar as características de uma única partícula. Ren e Zhang fizeram exatamente isso, geração de modelos detalhados usando imagens microscópicas eletrônicas de uma única proteína.
p Ele chama sua técnica de "tomografia de elétrons de partículas individuais, ”Ou IPET. O trabalho é descrito na edição de 24 de janeiro da
PLoS One , o jornal científico de código aberto, em um artigo intitulado “IPET e FETR:Abordagem Experimental para o Estudo da Dinâmica da Estrutura Molecular por Tomografia Crio-Eletrônica de uma Estrutura de uma Única Molécula”.
p As imagens 3-D relatadas no artigo incluem as de um único anticorpo IgG e apolipoproteína A-1 (ApoA-1), uma proteína envolvida no metabolismo humano. O objetivo de Ren é produzir imagens 3D individuais de proteínas medicamente significativas, como o HDL - o “colesterol bom” protetor do coração, cuja estrutura tem escapado aos esforços de legiões de cientistas armados com ferramentas de modelagem de proteínas muito mais poderosas. “Estamos no bom caminho, ”Diz Ren.
Uma animação por computador demonstra a dinâmica flexível - as partes móveis - do anticorpo IgG humano. Imagens 3-D de duas partículas de anticorpos individuais (cinza) foram geradas usando tomografia EM com IPET. A demonstração mostra como as mesmas cadeias moleculares (vermelho, laranja, e modelos semelhantes a macarrão verde) da partícula de anticorpo # 1 pode caber precisamente na partícula # 2, que foi encontrado sob o microscópio em uma pose totalmente diferente. p Ren tem as credenciais de quem sabe o que pode fazer. Ele foi recrutado para trabalhar no Berkeley Lab em agosto de 2010 na Universidade da Califórnia em San Francisco, onde ele usou um microscópio crioeletrônico e técnicas de cálculo de média mais convencionais para discernir a estrutura 3-D do LDL - o “colesterol ruim” considerado um importante fator de risco para doenças cardíacas.
p Suas imagens de proteínas individuais são um pouco confusas, mesmo depois de serem limpos por filtros complexos de computador, mas muito informativo para o observador treinado. Essas partículas individuais são extraordinariamente minúsculas, exigindo que Ren zere em um ponto de menos de 20 nanômetros. Ele relatou imagens de proteínas tão pequenas quanto 70 kDa. Isso é kilodaltons, uma escala liliputiana (expressa em unidades de massa) reservada para medir os átomos, moléculas, e fragmentos de DNA. É uma maneira mais útil de dimensionar objetos moles, como proteínas que podem ser agrupadas, pegajoso, ou disquete.
p Ao contrário das imagens esculturais de modelos de proteínas, um conjunto dessas fotografias pode transmitir uma ideia dessas partículas em toda a sua flexibilidade em nanoescala. Dentro da estrutura complexa dessas proteínas estão os segredos de sua função, e talvez chaves para drogas que bloqueiam os maus e promovem os bons. Com alguma filtragem de computador adicional, um modelo de proteína de alto contraste pode ser gerado a partir das imagens e animado para mostrar suas partes móveis em 3-D.
p “Isso permite que você veja a personalidade de cada proteína, ’’ Diz Ren. “É uma prova de conceito para algo que as pessoas pensavam ser impossível.”
p Ao observar a estrutura de proteínas individuais, é possível entender sua flexibilidade, partes móveis. “Isso abre uma porta para o estudo da dinâmica das proteínas, ”Ren diz. “Anticorpos, por exemplo, não são sólidos. Eles são muito flexíveis, muito dinâmico. ”
p Como Ren conseguiu tanta versatilidade em seu Libra 120? “Não é um modelo muito sofisticado, ’’ Ele admite. Muito tem a ver com os acessórios que ele aparafusa na máquina, e com sua própria arte e paciência. Ele equipou o microscópio com um $ 300, Câmera CCD 000, algum software de processamento de imagem poderoso, agentes contrastantes especiais, e um dispositivo chamado “filtro de energia” que analisa os dados digitalizados da câmera e seleciona os sinais fracos. Completamente familiarizado com sua máquina personalizada, ele também emprega um elemento de graxa de cotovelo, trabalhando muitas horas para extrair as imagens poderosas de uma torrente de ruído digital.
 p O cientista Gang Ren (em pé) e seu colega de pós-doutorado Lei Zhang podem verificar imagens de proteínas individuais em seu microscópio crioeletrônico na Fundição Molecular do Berkeley Lab.
p O cientista Gang Ren (em pé) e seu colega de pós-doutorado Lei Zhang podem verificar imagens de proteínas individuais em seu microscópio crioeletrônico na Fundição Molecular do Berkeley Lab.
p Os múltiplos ângulos usados para criar o retrato 3D ajudam a resolver a imagem molecular tênue. “Todas as imagens são barulhentas, ”Ren explica. “Na física, o ruído é inconsistente entre as imagens, mas o sinal - o objeto ou proteína - é consistente. Usando essa abordagem, descobrimos que a parte consistente (o sinal) pode ser aprimorada, enquanto a parte inconsistente (o ruído) será reduzida substancialmente. ”
p Os microscópios eletrônicos focalizam fluxos de elétrons em vez de luz para ver coisas incrivelmente minúsculas. O curto comprimento de onda de um feixe de elétrons permite uma resolução e ampliação muito maiores do que a luz visível. Microscópios eletrônicos poderosos têm sido usados há décadas para sondar materiais em escala atômica; e bem ao lado da Fundição Molecular fica o Centro Nacional de Microscopia Eletrônica do Berkeley Lab, que abriga os microscópios mais poderosos do mundo. O microscópio TEAM 0.5 pode distinguir objetos tão pequenos quanto o raio de um átomo de hidrogênio. Mas esses microscópios pesados produzem essa resolução em escala atômica com pulsos de energia que obliterariam a maioria das proteínas biológicas moles. Os microscópios eletrônicos de alta potência são usados principalmente para sondar a estrutura atômica de fortes, materiais sólidos, como o grafeno - uma rede de carbono com apenas um átomo de espessura.
p O laboratório de Ren é especializado em crioEM, que examina objetos congelados a -180 ° C (-292 ° F). Um banho de nitrogênio líquido congela as amostras tão rapidamente que nenhum cristal de gelo se forma. “É amorfo, como vidro, __ Ren diz. As amostras de proteínas são congeladas em um disco do tamanho da unha do bebê, preenchido com pequenos poços de 2 mícrons de diâmetro. O disco é inserido no microscópio em um suporte giratório que pode inclinar a amostra até 140 ° dentro do vácuo - ângulos de câmera suficientes para produzir uma perspectiva 3-D. “O desafio é isolá-lo do ar, e girá-lo sem vibrações, até mesmo as vibrações do borbulhar de nitrogênio líquido, ’’ Diz Ren.
p A temperatura extremamente baixa fixa as amostras e evita que sequem no vácuo necessário para a varredura de elétrons. Ele cria condições favoráveis para imagens em doses muito mais baixas de elétrons - baixas o suficiente para manter uma única proteína suave intacta, enquanto mais de 100 imagens são tiradas em um período de uma a duas horas.

