Lendo os blocos de construção da vida:pesquisadores desenvolvem ferramentas para acelerar o sequenciamento de DNA
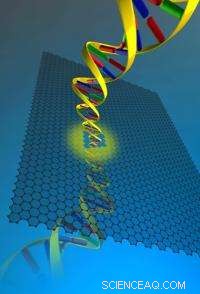 p Em um artigo de capa da Nature Nanotechnology, pesquisadores de Harvard e do Instituto de Tecnologia de Massachusetts demonstraram que uma longa molécula de DNA pode ser puxada através de um nanoporo de grafeno. Crédito:Slaven Garaj, Jene Golovchenko, Jing Kong, Daniel Branton, W. Hubbard, e A. Reina
p Em um artigo de capa da Nature Nanotechnology, pesquisadores de Harvard e do Instituto de Tecnologia de Massachusetts demonstraram que uma longa molécula de DNA pode ser puxada através de um nanoporo de grafeno. Crédito:Slaven Garaj, Jene Golovchenko, Jing Kong, Daniel Branton, W. Hubbard, e A. Reina
p Os cientistas estão um passo mais perto de uma revolução no sequenciamento de DNA, após o desenvolvimento em um laboratório de Harvard de um minúsculo dispositivo projetado para ler as mínimas mudanças elétricas produzidas quando as fitas de DNA são passadas por minúsculos orifícios - chamados de nanoporos - em uma membrana eletricamente carregada. p Conforme descrito em
Nature Nanotechnology em 11 de dezembro, uma equipe de pesquisa liderada por Charles Lieber, o professor de química Mark Hyman Jr., conseguiram, pela primeira vez, criar um detector de nanoporos integrado, um desenvolvimento que abre a porta para a criação de dispositivos que podem usar matrizes de milhões de buracos microscópicos para sequenciar o DNA de forma rápida e barata.
p Descrita pela primeira vez há mais de 15 anos, O sequenciamento nanopore mede as mudanças sutis da corrente elétrica produzidas à medida que as quatro moléculas básicas que compõem o DNA passam pelo poro. Ao ler essas mudanças, os pesquisadores podem sequenciar o DNA com eficácia.
p Mas ler essas mudanças sutis na corrente está longe de ser fácil. Uma série de desafios - desde como registrar as pequenas mudanças na corrente até como escalar o processo de sequenciamento - significou que o processo nunca foi possível em grande escala. Lieber e sua equipe, Contudo, acreditam que encontraram uma solução unificada para a maioria desses problemas.
p “Até desenvolvermos nosso detector, não havia maneira de medir localmente as mudanças na corrente, ”Disse Lieber. “Nosso método é ideal porque é extremamente localizado. Podemos usar todo o trabalho existente que foi feito em nanoporos, mas com um detector local estamos um passo mais perto de revolucionar completamente o sequenciamento. ”
 p “O transistor de nanofio mede a mudança de potencial elétrico no poro e amplifica efetivamente o sinal, ”Disse Charles Lieber. “Além de um sinal maior, isso nos permite ler as coisas muito mais rapidamente. "Crédito:Kris Snibbe / Harvard Staff Photographer
p “O transistor de nanofio mede a mudança de potencial elétrico no poro e amplifica efetivamente o sinal, ”Disse Charles Lieber. “Além de um sinal maior, isso nos permite ler as coisas muito mais rapidamente. "Crédito:Kris Snibbe / Harvard Staff Photographer
p O detector desenvolvido por Lieber e sua equipe surgiu de trabalhos anteriores com nanofios. Usando os fios ultrafinos como um transistor em nanoescala, eles são capazes de medir as mudanças na corrente de forma mais local e precisa do que nunca.
p “O transistor de nanofio mede a alteração do potencial elétrico no poro e amplifica efetivamente o sinal, ”Disse Lieber. “Além de um sinal maior, isso nos permite ler as coisas muito mais rapidamente. Isso é importante porque o DNA é tão grande [que] o rendimento de qualquer método de sequenciamento precisa ser alto. Em princípio, este detector pode funcionar em frequências gigahertz. ”
p A medição altamente localizada também abre a porta para o sequenciamento paralelo, que usa matrizes de milhões de poros para acelerar drasticamente o processo de sequenciamento.
p Além do potencial para melhorar significativamente a velocidade de sequenciamento, o novo detector tem a promessa de reduzir drasticamente o custo do sequenciamento de DNA, disse Ping Xie, associado do Departamento de Química e Biologia Química e coautor do artigo que descreve a pesquisa.
p Os métodos de sequenciamento atuais geralmente começam com um processo chamado reação em cadeia da polimerase, ou amplificação de DNA, que copia uma pequena quantidade de DNA milhares de milhões de vezes, tornando mais fácil sequenciar. Embora extremamente importante para a biologia, o processo é caro, exigindo suprimentos químicos e equipamentos de laboratório caros.
p No futuro, Xie disse, será possível construir a tecnologia de sequenciamento nanopore em um chip de silício, permitindo médicos, pesquisadores, ou mesmo a pessoa média a usar o sequenciamento de DNA como ferramenta de diagnóstico.
p O avanço da equipe de Lieber pode em breve fazer a transição do laboratório para o produto comercial. O Harvard Office of Technology Development está trabalhando em uma estratégia para comercializar a tecnologia de forma adequada, incluindo o licenciamento para uma empresa que planeja incorporá-lo em sua plataforma de sequenciamento de DNA.
p “Agora mesmo, somos limitados em nossa capacidade de realizar o sequenciamento de DNA, ”Xie disse. “A tecnologia de sequenciamento atual é onde os computadores estavam nos anos 50 e 60. Requer muitos equipamentos e é muito caro. Mas apenas 50 anos depois, computadores estão por toda parte, mesmo em cartões de felicitações. Nosso detector abre a porta para fazer uma coleta de sangue e saber imediatamente com o que o paciente está infectado, e tomar decisões de tratamento muito rapidamente. ” p
Esta história foi publicada como cortesia da Harvard Gazette, Jornal oficial da Universidade de Harvard. Para notícias adicionais da universidade, visite Harvard.edu.

