Novo kit de ferramentas torna as simulações de dinâmica molecular mais acessíveis
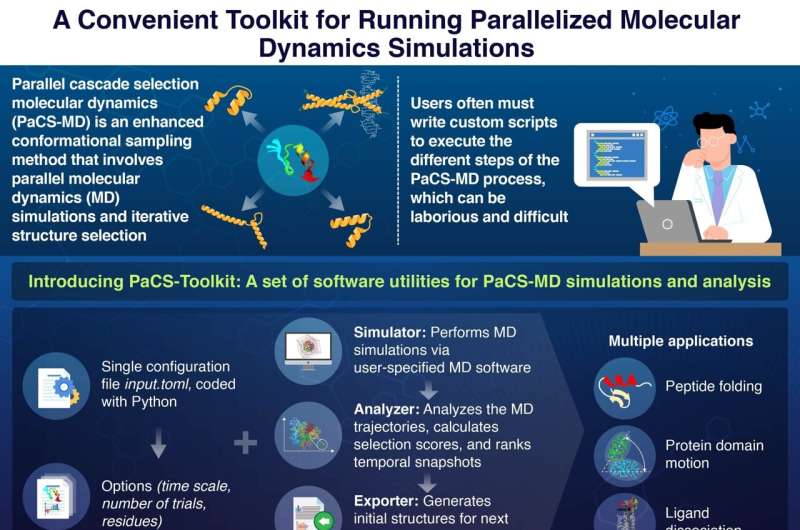 O PaCS-Toolkit visa melhorar a acessibilidade às simulações e análises do PaCS-MD, acelerando assim o progresso na pesquisa de biologia molecular e no desenvolvimento de medicamentos. Crédito:tecnologia de Tóquio
O PaCS-Toolkit visa melhorar a acessibilidade às simulações e análises do PaCS-MD, acelerando assim o progresso na pesquisa de biologia molecular e no desenvolvimento de medicamentos. Crédito:tecnologia de Tóquio Simulações de dinâmica molecular (MD) tornaram-se uma ferramenta poderosa nos campos cada vez maiores da biologia molecular e do desenvolvimento de medicamentos. Embora existam muitas técnicas de simulação MD, a seleção em cascata paralela MD (PaCS-MD) é particularmente útil ao estudar o dobramento (ou "conformação") de proteínas ou as interações entre proteínas e ligantes.
O ponto crucial deste método consiste em executar múltiplas simulações MD em paralelo, explorando assim diferentes conformações possíveis simultaneamente. Usando critérios de seleção cuidadosamente elaborados, conformações promissoras podem ser detectadas automaticamente em "instantâneos temporais" e investigadas posteriormente. Esta estratégia acelera enormemente a descoberta de interações moleculares importantes e processos dinâmicos que ajudam os cientistas a compreender os movimentos funcionais das proteínas.
No entanto, um dos obstáculos do PaCS-MD é que os usuários devem escrever scripts personalizados para executar as simulações MD desejadas. Nestes scripts, eles devem especificar as condições iniciais e os recursos alvo, selecionar o software MD a ser usado, implementar um procedimento de classificação de instantâneos e preparar as estruturas iniciais para o próximo ciclo de simulação. Este processo, que pode ser bastante complexo e sujeito a erros, cria uma barreira de entrada considerável para cientistas interessados em usar o PaCS-MD.
Felizmente, uma equipa de investigadores da Escola de Ciências e Tecnologia da Vida do Instituto de Tecnologia de Tóquio, no Japão, decidiu recentemente abordar esta questão. Em seu último estudo, publicado no The Journal of Physical Chemistry B e liderada pelo professor Akio Kitao, a equipe desenvolveu um pacote de software chamado PaCS-Toolkit para tornar o PaCS-MD mais acessível e conveniente de usar.
Uma vantagem notável do PaCS-Toolkit é que todo o processo de simulação é configurado através de um único arquivo de configuração. Neste arquivo, os usuários especificam parâmetros importantes para as simulações, incluindo o tipo de PaCS-MD, o número de simulações MD a serem executadas em paralelo e os resíduos de proteínas ou átomos a serem rastreados como critérios de seleção para ramificações paralelas.
O PaCS-Toolkit pega esse arquivo de configuração, junto com os arquivos de entrada MD padrão, e executa simulações PaCS-MD de acordo com o software MD especificado. Vale ressaltar que, como o pacote é um software aberto escrito em Python – uma linguagem de programação popular – os usuários podem ajudar a melhorar o PaCS-Toolkit e ampliar suas funcionalidades.
"Nosso kit de ferramentas mantém flexibilidade para que novos recursos, bibliotecas e software MD possam ser adicionados através da introdução de classes responsáveis em Python. Os usuários que podem programar nesta linguagem devem ser capazes de modificar o código do PaCS-Toolkit e implementar novos métodos conforme necessário, " diz o Prof. Kitao.
Outra vantagem crucial do PaCS-Toolkit reside na sua otimização e compatibilidade com diferentes ambientes computacionais, seja um conjunto de supercomputadores com interface de passagem de mensagens (MPI), servidores equipados com múltiplas placas gráficas (GPUs) ou computadores pessoais, como laptops.
“O PaCS-Toolkit incorpora fortemente paralelização usando MPI, GPU e o pacote de multiprocessamento Python, permitindo a otimização do tempo de computação dependendo dos recursos computacionais disponíveis”, explica o Prof.
Para mostrar o potencial do seu kit de ferramentas, os pesquisadores conduziram simulações PaCS-MD adaptadas para três aplicações diferentes. Estes incluíram o dobramento da miniproteína chignolin, o movimento de domínios proteicos em uma enzima SARS-CoV-2 e a dissociação do ligante de um importante receptor de adenosina.
“Em conjunto, nossos resultados indicam que o PaCS-Toolkit pode ser facilmente utilizado para simular uma ampla variedade de dinâmicas para diferentes tipos de sistemas moleculares”, conclui o Prof.
Este kit de ferramentas pode ajudar a desbloquear o verdadeiro potencial das simulações PaCS-MD em vários campos, permitindo que pesquisadores interessados esclareçam processos moleculares complexos e acelerem a descoberta de medicamentos.
