Novo método para gerar proteínas de ligação potentes e específicas para novos medicamentos
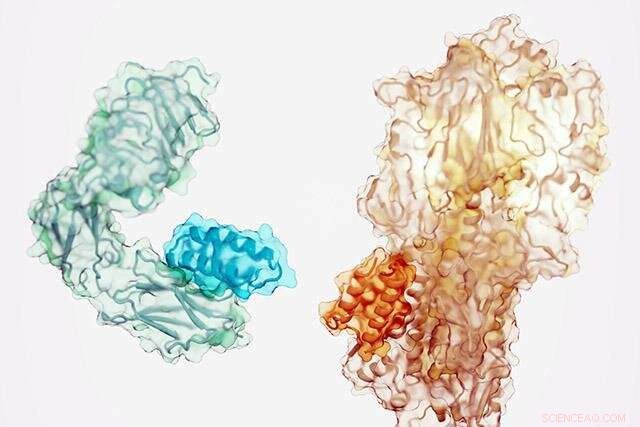
Pequenas proteínas (tonalidade mais escura) projetadas para se ligarem ao receptor de insulina (esquerda) e a um componente do vírus influenza (direita). Crédito:Ian C. Haydon/Institute for Protein Design
Uma equipe de cientistas criou um novo método poderoso para gerar drogas proteicas. Usando computadores, eles projetaram moléculas que podem atingir proteínas importantes no corpo, como o receptor de insulina, bem como proteínas vulneráveis na superfície dos vírus. Isso resolve um desafio de longa data no desenvolvimento de medicamentos e pode levar a novos tratamentos para câncer, diabetes, infecção, inflamação e outros.
A pesquisa, publicada hoje na revista
Nature , foi liderado por cientistas no laboratório de David Baker, professor de bioquímica da Escola de Medicina da Universidade de Washington e ganhador do Prêmio Revelação de 2021 em Ciências da Vida.
"A capacidade de gerar novas proteínas que se ligam de forma firme e específica a qualquer alvo molecular que você deseja é uma mudança de paradigma no desenvolvimento de medicamentos e na biologia molecular de forma mais ampla", disse Baker.
Os anticorpos são os medicamentos à base de proteínas mais comuns de hoje. Eles normalmente funcionam ligando-se a um alvo molecular específico, que então se torna ativado ou desativado. Os anticorpos podem tratar uma ampla gama de distúrbios de saúde, incluindo COVID-19 e câncer, mas gerar novos é um desafio. Os anticorpos também podem ser caros de fabricar.
Uma equipe liderada por dois pesquisadores de pós-doutorado no laboratório Baker – Longxing Cao e Brian Coventry – combinou avanços recentes no campo do design computacional de proteínas para chegar a uma estratégia para criar novas proteínas que se ligam a alvos moleculares de maneira semelhante aos anticorpos. Eles desenvolveram um software que pode escanear uma molécula-alvo, identificar potenciais locais de ligação, gerar proteínas direcionadas a esses locais e, em seguida, selecionar milhões de proteínas de ligação candidatas para identificar aquelas com maior probabilidade de funcionar.
A equipe usou o novo software para gerar proteínas de ligação de alta afinidade contra 12 alvos moleculares distintos. Esses alvos incluem receptores celulares importantes, como TrkA, EGFR, Tie2 e o receptor de insulina, bem como proteínas na superfície do vírus influenza e SARS-CoV-2 (o vírus que causa o COVID-19).
"Quando se trata de criar novos medicamentos, há alvos fáceis e alvos difíceis", disse Cao, que agora é professor assistente na Westlake University. "Neste artigo, mostramos que mesmo alvos muito difíceis são suscetíveis a essa abordagem. Fomos capazes de fazer proteínas de ligação a alguns alvos que não tinham parceiros de ligação ou anticorpos conhecidos", disse.
No total, a equipe produziu mais de meio milhão de proteínas de ligação candidatas para os 12 alvos moleculares selecionados. Os dados coletados neste grande conjunto de proteínas de ligação candidatas foram usados para melhorar o método geral.
"Estamos ansiosos para ver como essas moléculas podem ser usadas em um contexto clínico e, mais importante, como esse novo método de projetar drogas proteicas pode levar a compostos ainda mais promissores no futuro", disse Coventry.
+ Explorar mais Rumo a um medicamento para tratar todos os coronavírus
