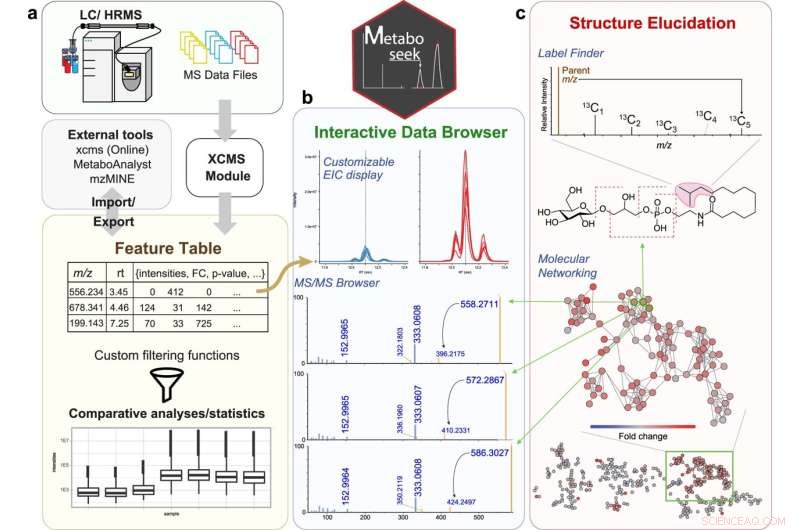
Metabolômica comparativa com Metaboseek. a Metaboseek inclui um módulo XCMS integrado para detecção de recursos e agrupamento de recursos (com anotação CAMERA) e aceita tabelas de recursos geradas por outro software. b Os recursos podem ser anotados e priorizados usando amplas opções de filtragem e ferramentas de estatísticas integradas. Os dados brutos para cada recurso molecular podem ser pesquisados rapidamente, incluindo EICs associados, espectros MS1 e MS/MS. c O navegador de dados interage com um conjunto de ferramentas de elucidação de estrutura, por exemplo, fórmula molecular baseada em SIRIUS e previsão de estrutura, o Label Finder para identificar compostos marcados com isótopos e o localizador de padrões MS/MS para identificar recursos de MS com padrões de fragmentação característicos. Crédito:Comunicação da Natureza (2022). DOI:10.1038/s41467-022-28391-9
Como pesquisador associado de pós-doutorado no laboratório do membro do corpo docente do BTI, Frank Schroeder, Max Helf viu seus colegas de laboratório lutarem continuamente quando estavam analisando dados. Então, ele decidiu fazer algo a respeito e desenvolveu um aplicativo gratuito e de código aberto chamado Metaboseek, que agora é essencial para o trabalho do laboratório.
O laboratório Schroeder estuda a lombriga Caenorhabditis elegans, um dos sistemas modelo de maior sucesso para a biologia humana, para descobrir novos metabólitos que governam as vias de sinalização conservadas evolutivamente e podem ser úteis como pistas para o desenvolvimento de novos produtos farmacêuticos ou agroquímicos. Os pesquisadores realizam essa tarefa comparando os metabólitos entre duas populações diferentes de vermes – um processo chamado metabolômica comparativa.
Dado que as amostras rotineiramente contêm mais de 100.000 compostos, abordagens computacionais são essenciais para realizar a análise.
A equipe contava com pacotes de software que não ofereciam o nível de flexibilidade necessário para personalizar facilmente os parâmetros de análise. Essa limitação e a falta de uma interface gráfica de usuário adequada significavam que os colegas de Helf enfrentaram a incômoda tarefa de inspecionar visualmente montes de dados - por exemplo, para detectar possíveis falsos positivos - e alternar entre várias outras ferramentas de software para confirmar e filtrar aqueles sem sentido. resultados.
"Parecia muito ineficiente para mim e não consegui superar as deficiências de outras soluções de software para esse problema", disse Helf. "Achei que deveria haver uma maneira mais fácil, então comecei a escrever código para meu próprio software."
Helf desenvolveu a versão inicial de seu software em 2017 e continuou a aprimorá-lo nos dois anos seguintes. "Além de abordar os problemas que meus colegas de laboratório já estavam enfrentando, conversei com eles sobre o que mais os impedia - o que eles queriam fazer, mas nem estavam tentando - e construí esses recursos no aplicativo", disse Helf, que agora é um gerente de produtos de bioinformática na empresa de proteômica Biognosys AG. "Eu queria que esta nova ferramenta fosse fácil de usar e acessível a qualquer pessoa que faça biologia química."
O resultado foi o Metaboseek, um aplicativo com interface gráfica que incorpora várias ferramentas de análise de dados que pesquisadores não codificadores não teriam. O aplicativo simplifica a análise de dados metabolômicos comparativos, ajudando o pesquisador a determinar quais recursos de dados são reais e permitindo que eles se aprofundem nesses recursos - tudo na mesma ferramenta.
"Max fez isso sem que eu pedisse", disse Schroeder. "Antes que eu soubesse que isso estava acontecendo, havia o Metaboseek. Começamos a usá-lo e agora nosso laboratório e muitos colaboradores não poderiam existir sem ele."
Em um estudo publicado na
Nature Communications em 10 de fevereiro, a equipe de Schroeder forneceu uma prova de conceito para Metaboseek, aplicando-o a uma importante via de metabolismo de gordura que ainda não havia sido estudada:a via de α-oxidação em C. elegans que ajuda a quebrar uma classe de ácidos graxos.
Usando Metaboseek, a equipe descobriu que as lombrigas sem um gene-chave na via de α-oxidação acumulavam centenas de metabólitos não relatados anteriormente. As descobertas são importantes porque a α-oxidação é uma via bioquímica básica em vermes que é conservada em humanos, disse Schroeder.
"Bennett Fox fez o trabalho de química, então este estudo foi uma boa colaboração entre os dois pós-doutorandos", acrescentou Schroeder, que também é professor do Departamento de Química e Biologia Química da Universidade de Cornell.
De acordo com Schroeder e Helf, existem algumas razões pelas quais não existem muitas boas ferramentas analíticas para comparar dados metabolômicos. Primeiro, a metabolômica comparativa é um campo relativamente jovem em comparação com outros campos da biologia com muitos dados, como genômica (que se concentra no DNA) e proteômica (que se concentra em proteínas), então não houve tempo suficiente para desenvolver ferramentas de software e infraestrutura de banco de dados .
Além disso, na última década, o advento de espectrômetros de massa acessíveis e de resolução ultra-alta para coletar dados metabolômicos aumentou talvez mais de dez vezes a quantidade de dados que uma amostra pode gerar - criando uma necessidade ainda maior de ferramentas sofisticadas que podem manter com a enxurrada de dados.
O Metaboseek atende a essas necessidades com uma variedade de recursos para analisar vários tipos de dados para auxiliar na identificação de compostos, determinação de estrutura, atribuição de metabólitos a famílias com base em semelhanças estruturais, rastreamento de compostos radiomarcados e muito mais.
+ Explorar mais Um pacote R para análise abrangente de dados de proteômica de baixo para cima centrada em peptídeos e proteínas
