Crédito:Universidade de Kanazawa
Pesquisadores da Universidade de Kanazawa relatam no
Journal of Physical Chemistry Letters como simular imagens de microscopia de força atômica 3D de sistemas fora de equilíbrio envolvendo biomoléculas. A abordagem faz uso de uma célebre equação da termodinâmica aplicável a situações de não equilíbrio.
A microscopia de força atômica tridimensional (3D-AFM) é uma técnica usada para sondar a distribuição de moléculas de solvente em interfaces sólido-líquido. Inicialmente aplicado para estudar situações em que o solvente é a água, o método agora está sendo utilizado também para outras moléculas. Um desenvolvimento recente é usar 3D-AFM para resolver a organização de biopolímeros, como cromossomos ou proteínas dentro das células. Devido à complexidade de tais sistemas, no entanto, são necessárias simulações do processo de imagem 3D-AFM para auxiliar na sua interpretação. Os métodos de simulação desenvolvidos até agora assumiram que o sistema testado está em equilíbrio durante o ciclo de varredura do AFM. Isso limita sua validade a situações em que as moléculas do solvente se movem muito mais rápido do que a sonda de varredura. Agora, Takeshi Fukuma da Universidade de Kanazawa e colegas desenvolveram uma abordagem de simulação 3D-AFM que funciona para sistemas não em equilíbrio, aplicável a situações em que o movimento molecular acontece em escalas de tempo comparáveis ou maiores que a do ciclo de sondagem AFM.
O princípio básico do AFM é fazer uma ponta muito pequena, presa a um cantilever, para escanear a superfície de uma amostra. A resposta da ponta às diferenças de altura na superfície digitalizada fornece informações estruturais da amostra. No 3D-AFM, a ponta é feita para penetrar na amostra e a força experimentada pela ponta é o resultado de interações com moléculas próximas (partes de). Para uma dada posição horizontal (xy) da ponta, a dependência da força F na posição vertical (z) da ponta conforme ela penetra na amostra é capturada em uma curva força-distância (F versus z). A combinação de todas as curvas força-distância obtidas durante a varredura xy fornece a imagem 3D-AFM.
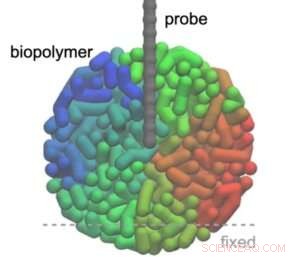
Crédito:Universidade de Kanazawa Fukuma e colegas consideraram a situação em que uma ponta AFM sonda um biopolímero globular e modelaram tanto a ponta quanto a molécula como contas conectadas por molas (2.000 contas para a molécula, 50 contas para a ponta). Eles calcularam as curvas força-distância usando a chamada igualdade de Jarzynski, uma equação que relaciona a diferença de energia livre entre dois estados de um sistema com o trabalho (proporcional à força) necessário para ir de um estado para o outro. É importante ressaltar que a igualdade vale para situações de não equilíbrio.
Os pesquisadores conseguiram mostrar que as simulações reproduziam a estrutura interna do biopolímero, com algumas características da fibra sendo claramente observáveis. Eles também analisaram como a velocidade de varredura afeta os resultados da simulação e descobriram que existe uma faixa de velocidade ideal para a varredura vertical (z). Finalmente, Fukuma e colegas simularam imagens 3D-AFM de fibras de citoesqueleto para as quais existem imagens 3D-AFM obtidas experimentalmente e descobriram que as simulações concordam bem com o experimento. Os cientistas concluíram, portanto, que seu método "é aplicável a várias fibras em células, como DNA e assim por diante, alterando parâmetros como rigidez, fornecendo uma base teórica importante para essas medições experimentais".
+ Explorar mais Atribuindo recursos móveis em microscopia de força atômica de alta velocidade
