Supercomputadores simulam novos caminhos para o potencial tratamento de vírus de RNA
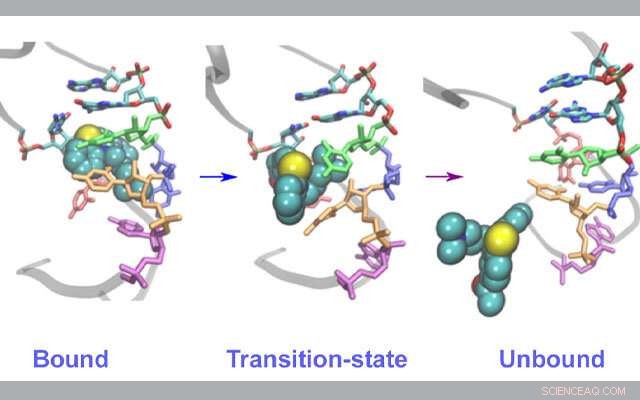 p Alterações estruturais no RNA durante a ligação / desacoplamento do fármaco. Crédito:Lev Levintov, Universidade de New Hampshire
p Alterações estruturais no RNA durante a ligação / desacoplamento do fármaco. Crédito:Lev Levintov, Universidade de New Hampshire
p Pesquisadores da University of New Hampshire (UNH) recentemente usaram o Comet no San Diego Supercomputer Center na UC San Diego e Stampede2 no Texas Advanced Computing Center para identificar novas vias de ligação / desacoplamento de inibidor em um vírus baseado em RNA. As descobertas podem ser benéficas para a compreensão de como esses inibidores reagem e, potencialmente, ajudar a desenvolver uma nova geração de drogas para combater vírus com altas taxas de mortalidade, como HIV-1, Zika, Ebola, e SARS-CoV2, o vírus que causa COVID-19. p "Quando começamos esta pesquisa, nunca previmos que estaríamos no meio de uma pandemia causada por um vírus de RNA, "disse Harish Vashisth, professor associado de engenharia química da UNH. "À medida que esses tipos de vírus surgem, esperamos que nossas descobertas ofereçam uma compreensão aprimorada de como os RNAs virais interagem com os inibidores e sejam usados para projetar tratamentos melhores. "
p Semelhante a como os humanos codificam seu genoma usando DNA, muitos vírus têm uma composição genética de moléculas de RNA. Esses genomas baseados em RNA contêm locais potenciais onde os inibidores podem anexar e desativar o vírus. Parte do desafio no desenvolvimento de drogas são as variações ou mutações no genoma viral que podem impedir que os inibidores se liguem.
p Em seu estudo, publicado recentemente no
Journal of Physical Chemistry Letters , Vashisth e sua equipe criaram simulações de dinâmica molecular usando os supercomputadores Comet e Stampede2 para observar especificamente um fragmento de RNA do vírus HIV-1 e sua interação com a acetilpromazina, uma pequena molécula que interfere no processo de replicação do vírus.
p Os cientistas se concentraram nos elementos estruturais do genoma do RNA do HIV-1 porque eles são considerados um bom modelo para estudar os mesmos processos em uma ampla gama de vírus de RNA. Essas simulações permitiram que eles descobrissem as vias de desvinculação do inibidor do RNA viral em vários eventos raros - que muitas vezes são difíceis de observar experimentalmente - que inesperadamente mostraram um movimento coordenado em muitas partes da bolsa de ligação que são os blocos de construção do RNA.
p Graças às alocações de Extreme Science and Engineering Environment (XSEDE) da National Science Foundation (NSF) no Comet e Stampede2, os pesquisadores conseguiram executar centenas de simulações ao mesmo tempo para observar o que é chamado de eventos raros de inversão de base envolvidos no processo de ligação / desacoplamento do inibidor que forneceu os novos detalhes do mecanismo subjacente a esse processo.
p "Nossa esperança é que isso agregue novas possibilidades a um campo tradicionalmente focado em estruturas biomoleculares estáticas e leve a novos medicamentos, "Disse Vashisth.
