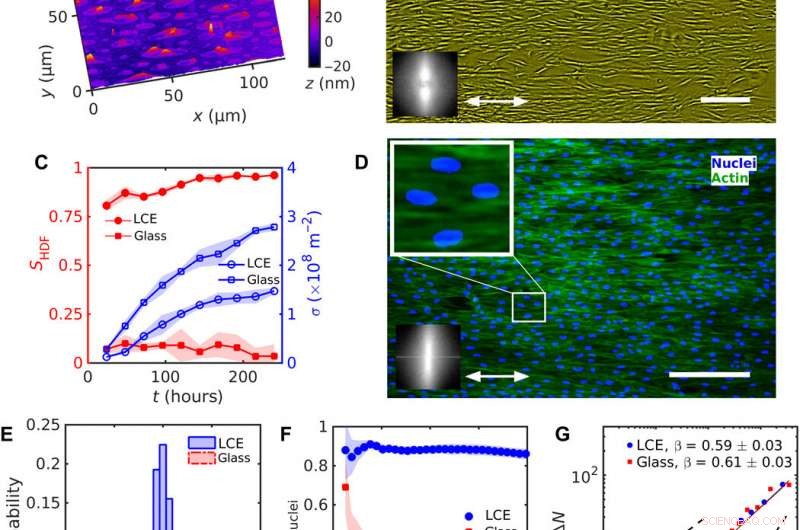
Alinhamento uniforme de células HDF em LCE com um nˆLCE uniforme =const. (A) Textura de microscopia holográfica digital (DHM) da superfície do LCE após o contato com o meio de crescimento aquoso. (B) Textura de microscopia de contraste de fase (PCM) de células HDF crescendo em substratos LCE 120 horas após a semeadura. A seta de duas pontas representa nˆLCE. (C) Evolução do parâmetro de ordem SHDF de corpos celulares (símbolos vermelhos preenchidos) e densidade celular σ (símbolos azuis vazios). (D) Texturas microscópicas fluorescentes de células HDF em LCE; núcleos marcados com fluorescência (azul) e proteínas F-actina do citoesqueleto (verde). A textura ampliada mostra núcleos alongados orientados na mesma direção que os corpos das células. Inserções em (B) e (D) mostram rápida transformação de Fourier de (B) PCM e (D) texturas fluorescentes de F-actina indicando ordem de orientação ao longo do nˆLCE uniforme. (E) Distribuição da orientação dos núcleos. (F) Dependência do parâmetro de ordem Snuclei de núcleos do tamanho de uma subjanela quadrada. (G) Flutuações de densidade numérica ΔN calculadas para o número médio de núcleos celulares 〈N〉. Barras de escala, 300 μm. Crédito:Science Advances, doi:10.1126 / sciadv.aaz6485
As células eucarióticas nos tecidos vivos podem afetar processos fisiológicos importantes, como apoptose e migração celular com base na formação de padrões dinâmicos com orientações espacialmente variáveis. Contudo, ainda é um desafio projetar um mapa predefinido de ordem orientacional em um tecido em crescimento no laboratório. Em um novo estudo agora publicado em Avanços da Ciência , Taras Turiv e uma equipe de pesquisa em física química, materiais avançados e ciências biomédicas na Kent State University, Ohio, NÓS., detalhou uma nova abordagem para produzir monocamadas de células de fibroblastos dérmicos humanos. Eles predefiniram os padrões de orientação e defeitos topológicos usando um elastômero de cristal líquido fotoalinhado (LCE) que inchou anisotropicamente em um meio aquoso. A equipe inscreveu os padrões no LCE, e a monocamada de tecido replicou os padrões para causar fortes variações nos fenótipos celulares (tamanho e forma), sua densidade superficial e flutuações de densidade numérica. A nova abordagem pode controlar o comportamento coletivo de células em tecidos vivos durante a diferenciação celular e morfogênese de tecidos para amplas aplicações em bioengenharia e medicina regenerativa.
Células que constituem tecidos vivos freqüentemente exibem ordem de orientação quando em contato próximo devido ao alinhamento mútuo de células anisométricas. A direção da orientação média pode variar no espaço e no tempo para produzir defeitos topológicos conhecidos como disclinações. Esses defeitos podem se mover dentro do tecido para desempenhar um papel importante durante os processos e tensões compressivo-dilativas, incluindo extração de células mortas. A capacidade de projetar um arcabouço de tecido de células vivas com ordem e controle orientacional é importante para pesquisadores biomédicos, a fim de investigar e manipular matéria viva. Os cientistas já produziram conjuntos de células ordenadas em superfícies fabricadas litograficamente, incluindo as bordas dos microcanais, em microgrooves e superfícies com gradientes de rigidez do material. Nesse trabalho, Turiv et al. tecidos projetados com um alto grau de ordem de orientação e direção espacialmente variável predeterminada, com base em um modelo de padrões de diretor em substratos LCE. A equipe usou células de fibroblasto dérmico humano (HDF) como unidades de construção do tecido modelado.
Os grãos de material servem como guia para as células HDF. Crédito:Science Advances, doi:10.1126 / sciadv.aaz6485
Os fibroblastos são as células do tecido conjuntivo mais comuns em mamíferos e geralmente mantêm uma forma plana alongada com papéis importantes durante a reparação e reestruturação do tecido, bem como cicatrização de feridas. Os cientistas podem reprogramar essas células em células-tronco pluripotentes para aplicações promissoras em diagnósticos e terapia. Nesse trabalho, os efeitos combinados de semeadura celular e divisão de tecidos HDF padronizados em substratos LCE predefinidos produziram tecidos confluentes. O LCE estruturado teve um impacto marcante no tecido, onde controlavam o padrão de alinhamento e distribuição espacial das células, a densidade deles, flutuações, e fenótipo. O LCE padronizado mostrou localizações de defeitos topológicos em tecidos por meio de interações superficiais anisotrópicas em locais predeterminados. Uma vez que o alinhamento celular e defeitos topológicos podem controlar processos bioquímicos em microescala, este trabalho abre a possibilidade de projetar superfícies para padronização controlada de tecidos, a fim de projetá-las para funções específicas.
Durante os experimentos, Turiv et al. apoiou o substrato LCE por uma placa de vidro e cobriu-o com óxido de índio e estanho (ITO) para reduzir a rugosidade da superfície, seguido pelo revestimento de uma camada de corante azo fotossensível e, finalmente, cobriu o substrato com um meio aquoso de cultura de células. Os grãos da superfície do material serviram como guia para as células HDF. Quando as células HDF foram suspensas em cultura de células, eles pareciam redondos, mas após a fixação no substrato, eles desenvolveram uma aparência alongada. Os cientistas registraram resultados de confluência (crescimento) de efeitos combinados de semeadura de células. Os resultados mostraram que a ordem de orientação ocorreu devido às interações diretas entre as células e o substrato LCE. Os substratos ajudaram a alinhar os corpos e núcleos das células HDF como um recurso importante para muitas funções celulares, incluindo a expressão de proteínas, motilidade, metabolismo e diferenciação.

Alinhamento padronizado de células HDF em LCE com um (-1/2, +1/2) par de defeitos. (A) Textura PolScope mostrando nˆLCE e retardo óptico de LCE em contato com o meio de crescimento celular. (B e C) texturas DHM da superfície LCE em contato com o meio de crescimento celular com defeitos (B) -1/2 e (C) +1/2. (D) células HDF coradas com fluorescência; 4 ′, Núcleos de células marcadas com 6-diamidino-2-fenilindol (azul) e filamentos de citoesqueleto de actina marcados com faloidina (verde). (E) A densidade superficial dos núcleos celulares σ como a função da distância r dos núcleos defeituosos +1/2 (azul) e -1/2 (vermelho). (F) Grandes flutuações de densidade numérica ΔN dos núcleos na vizinhança dos núcleos defeituosos. (G) Imagens de PCM de células HDF em substrato LCE 240 horas após a semeadura. Os pontos azuis e vermelhos denotam a localização dos núcleos com defeito +1/2 e -1/2, respectivamente, obtido da textura de microscopia óptica polarizada (POM) de LCE. (H) Campo orientacional codificado por cores e (I) o esquema correspondente do diretor de tecido HDF padronizado nˆHDF com imagem com PCM. Barras vermelhas em (I) denotam a orientação local dos eixos longos das células. (J) Separação entre defeitos de meia resistência para diretor horizontal e vertical entre eles (ver fig. S6). Barras de escala, 300 μm. Crédito:Science Advances, doi:10.1126 / sciadv.aaz6485
The HDF cells on LCE self-organized into aligned assemblies following pre-imposed directions. The team noted the behavior of cells and cell density to vary as they approached defect cores and other topological inconsistencies (bent type defects or splay type defects) on the LCE substrates. The substrates markedly impacted the HDF cells that were in contact with each other, resulting in collectively strong differences in the size and shape of cells. The marked differences indicated the influence of the predesigned patterns on the HDF cell phenotype (size and shape). Based on additional results, Turiv et al. credited the number density fluctuations in tissues to be influenced by the surface charge of director patterns and studied the issue in detail in a larger surface area.
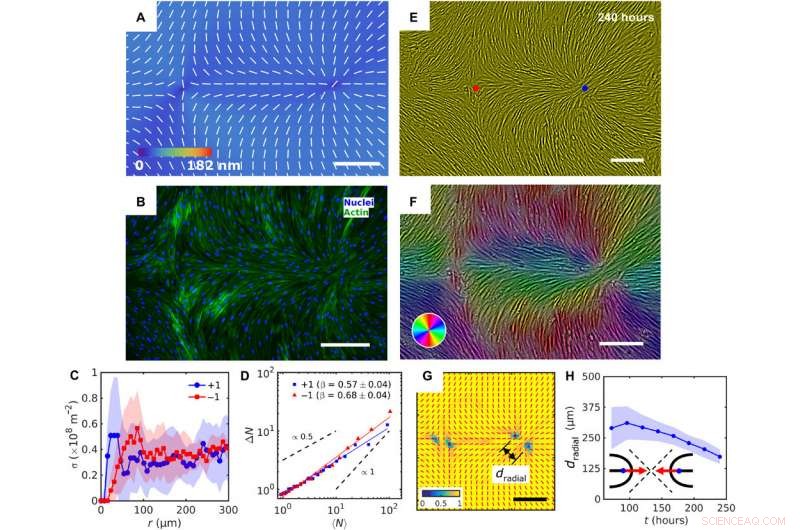
Patterned alignment of HDF cells on LCE predesigned with a pair of −1 and radial +1 (splay type) defects. (A) PolScope image of nˆLCE pattern of LCE in contact with the cell growth medium. (B) Fluorescently labeled HDF cells. (C) The surface density of cell nuclei σ as the function of distance from defect cores. (D) Large number density fluctuations ΔN with mean number of nuclei 〈N〉 showing a larger slope near −1 cores as compared to +1. (E) PCM textures of HDF cells on LCE layer at 240 hours after cell seeding. Red and blue dots denote location of −1 and +1 defects in patterned LCE obtained from crossed polarized textures. (F) Color-coded orientational field and (G) of the corresponding scheme of patterned HDF tissue director nˆHDF obtained from local anisotropy of PCM texture in (E). Red bars in (G) denote local orientation of cells’ long axes. (H) Time dependence of separation between two +1/2 defects near the +1 radial core. Barras de escala, 300 μm. Crédito:Science Advances, doi:10.1126/sciadv.aaz6485
Desta maneira, Taras Turiv and colleagues showed the dynamics and propagation of defects in patterned tissues and how they could be halted through surface anchoring forces. The scientists used LCE substrates with photopatterned structures of varying molecular orientations to grow biological tissues with predesigned cell alignment. The substrates affected cell alignment as well as cell surface density and cell phenotypes. The team noted higher density of cells in defect cores with positive topological charge, while cell density was lower near negative defects. The cells mechanistically aligned to the substrates by swelling upon contact with the aqueous cell culture medium, followed by aligning to predesigned photopatterned direction. This approach will allow materials scientists and bioengineers to design biological tissues with predetermined cell alignment and precise location of orientational defects. The outcomes can facilitate controlled cell migration, diferenciação. and apoptosis. The work can be further optimized to advance the understanding of fundamental mechanisms underlying tissue development and regeneration.

The orientational order of the assemblies in +1/2 and −1/2 disclinations. Crédito:Science Advances, doi:10.1126/sciadv.aaz6485
© 2020 Science X Network