
Uma nova técnica permitirá detectar vírus no campo. Nesse caso, um lote de inhame em Guadalupe. Crédito:D. Filloux, Cirad
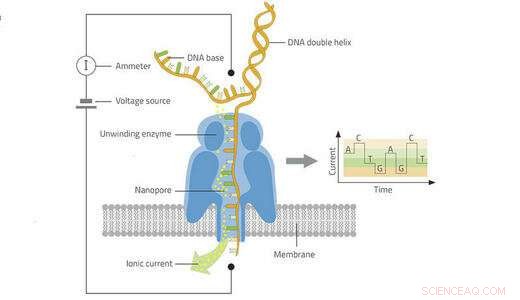
Em uma primeira vez para virologia de plantas, uma equipe do CIRAD recentemente usou a tecnologia nanopore para sequenciar os genomas inteiros de dois vírus de RNA do inhame. Esta técnica de biologia molecular pouco usada, mas promissora, abre caminho para novas ferramentas para o diagnóstico de plantas em campo, doenças animais e humanas.
Um novo, alto rendimento, A técnica de sequenciamento portátil foi desenvolvida nos últimos anos para fins de saúde humana e animal. Ele usa laboratórios móveis para diagnosticar vírus como Ebola ou Zika quase que instantaneamente, no campo. O diagnóstico é rápido e precoce, o que evita a necessidade de transferência de amostras contaminadas.
“A tecnologia é caracterizada pela produção de longas sequências de nucleotídeos, o que torna possível sequenciar todo o genoma viral, "Philippe Roumagnac, um virologista com CIRAD, explica. O CIRAD foi um dos primeiros laboratórios do mundo a testar e validar seu uso em virologia vegetal. "Usando uma planta doente de inhame, levou apenas algumas horas para sequenciar todo o genoma de dois vírus de RNA de fita simples, um macluravírus e um potivírus, "acrescenta seu colega Denis Filloux.
Tal como acontece com a virologia humana, o fato de que a técnica agora foi validada em um laboratório de virologia de plantas abre caminho para o tempo real, detecção móvel de crônica, vírus sazonais ou emergentes de plantas, mesmo em áreas isoladas. Ao encurtar o tempo que decorre entre a amostragem e o diagnóstico, a tecnologia ajudará as redes de vigilância epidemiológica a detectar organismos prejudiciais em um estágio anterior.
Crédito:Kerstin Göpfrich