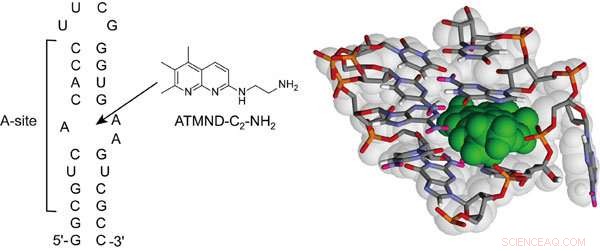
Estrutura química do ATMND-C 2 -NH 2 e sequência do modelo de RNA contendo local A bacteriano (Escherichia coli) usado neste estudo. Mostra também a possível estrutura do complexo entre ATMND-C 2 -NH 2 (cor verde) e loop interno do site A. Crédito:Seiichi Nishizawa
Um grupo de pesquisa da Universidade de Tohoku fez uma descoberta significativa com implicações positivas para o desenvolvimento de drogas de combate a bactérias. O local do aminoacil-tRNA (local A) da região de decodificação do RNA 16S no ribossomo bacteriano parece promissor para uma nova era de desenvolvimento de antibióticos.
Os antibióticos aminoglicosídeos tradicionais são problemáticos devido à sua alta toxicidade e ao potencial de desenvolvimento de resistência. A pesquisa na Universidade de Tohoku se concentrou em pequenos ligantes de ligação bacteriana ao sítio A, cujas estruturas são distintas da família dos aminoglicosídeos, que oferecem potencial para o desenvolvimento de novos medicamentos que tratam as infecções bacterianas com uma redução dos problemas associados aos antibióticos tradicionais.
O grupo de pesquisa liderado pelo Dr. Seiichi Nishizawa e Dr. Yusuke Sato (Departamento de Química, Graduate School of Science) relatou um novo pequeno ligante, ATMND-C 2 -NH 2 que tem a afinidade de ligação mais forte para o sítio A bacteriano entre os ligantes não aminoglicosídeos.
ATMND-C 2 -NH 2 mostra uma resposta de extinção de fluorescência significativa após a ligação seletiva à alça interna do RNA modelo contendo o local A bacteriano (Escherichia coli).
ATMND-C 2 -NH 2 também se mostrou útil como um indicador para avaliar as interações ligante / local A.
Os resultados obtidos pelo grupo de pesquisa oferecem uma base racional para a geração de novos ligantes de ligação ao local A com vista a novos antibióticos com menos toxicidade e desenvolvimento mínimo de resistência.