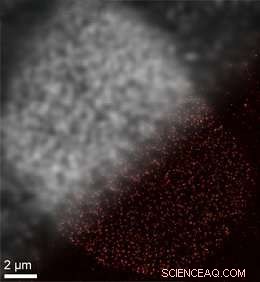
A microscopia de super-resolução baseada em SOMAmer DNA-PAINT permite uma resolução espacial aprimorada. Superior esquerdo:imagem tradicional limitada por difração de proteínas do complexo de poros nulcear em uma membrana de célula nuclear. Inferior direito:imagem de super-resolução DNA-PAINT obtida com ligantes SOMAmer. Crédito:Jungmann / LMU
Ralf Jungmann está interessado em processos que ocorrem em dimensões espaciais incrivelmente minúsculas. Jungmann é professor de física experimental na Ludwig-Maximilians-Universitaet (LMU) em Munique, e chefia um grupo de pesquisa em imagem molecular e bionanotecnologia no Instituto Max Planck de Bioquímica (Martinsried), e se concentra em estender os recursos da microscopia óptica. Seu objetivo é visualizar as interações moleculares que ocorrem dentro das células individuais. A fim de monitorar as redes de proteínas envolvidas em tais processos, ele usa fitas curtas de DNA covalentemente ligadas a vários marcadores de fluorescência como sondas para localizar proteínas-alvo com marcadores de DNA complementares. Ao explorar a especificidade de sequência e versatilidade da hibridização de DNA, é possível visualizar as distribuições de um grande número de moléculas em células individuais em super-resolução. A combinação de sequências de DNA com diferentes compostos fluorescentes explica porque a técnica leva o nome de DNA-PAINT.
Uma das principais restrições ao potencial da microscopia de fluorescência de super-resolução tem a ver com os marcadores empregados para detectar alvos de interesse biológico - eles são simplesmente grandes demais. “Estamos trabalhando com uma resolução instrumental de menos de 10 nanômetros. Mas os rótulos fluorescentes convencionalmente usados para marcar proteínas são muito maiores do que isso. E esse fator tem dificultado o progresso de todo esse campo de pesquisa, "Jungmann explica. Isso é o que motivou o trabalho descrito em um novo estudo, que aparece no jornal Métodos da Natureza . Nesse artigo, Jungmann e seus colegas exploram o uso dos chamados SOMAmers, uma classe especial de aptâmeros de DNA, como meio de reduzir o tamanho dos marcadores usados no DNA-PAINT. O termo 'aptâmero' foi originalmente cunhado para se referir a moléculas de RNA de fita simples que se dobram em formas tridimensionais definidas e são capazes de detectar especificamente espécies de proteínas únicas. Os aptâmeros de Jungmann são moléculas de DNA de fita simples, que se dobram em formas tridimensionais definidas que podem se ligar diretamente a proteínas especificamente direcionadas.
"O rótulo ideal usado para marcar proteínas de forma eficiente e específica deve atender a vários critérios, "diz Sebastian Strauss, membro do grupo de Jungmann e primeiro autor do novo estudo. "Deve ser o menor possível, e deve ligar-se a alvos estequiometricamente para permitir a quantificação precisa. Além disso, seria ideal sintetizar bibliotecas inteiras desses compostos e identificar rapidamente marcadores adequados para as proteínas de interesse. A fim de avaliar o potencial de aptâmeros de DNA, a equipe LMU colaborou com a empresa americana SomaLogic, que já havia desenhado, para outros propósitos, uma grande variedade de aptâmeros modificados (SOMAmers) que podem ligar especificamente milhares de proteínas diferentes. No novo estudo, os pesquisadores de Munique modificaram uma seleção desses aptâmeros para DNA-PAINT e desenvolveram protocolos de rotulagem eficientes para células fixas e de vida. O estudo atual mostra que é realmente possível melhorar a resolução atingível com marcadores de fluorescência convencionais usando esses novos reagentes de marcação em combinação com microscopia de super-resolução DNA-PAINT.
"Esperamos que o novo método forneça um impulso significativo para a microscopia de super-resolução, particularmente no que diz respeito à sua gama de aplicação em biologia, "diz Ralf Jungmann. Seu objetivo é usar o DNA-PAINT para visualizar e monitorar simultaneamente o maior número possível de proteínas e suas interações. Em experimentos futuros, ele e seus colegas planejam usar o novo método de marcação para criar imagens de redes de proteínas inteiras em alta resolução. "Seremos capazes de abordar questões biológicas e biomédicas que até agora eram experimentalmente inacessíveis."