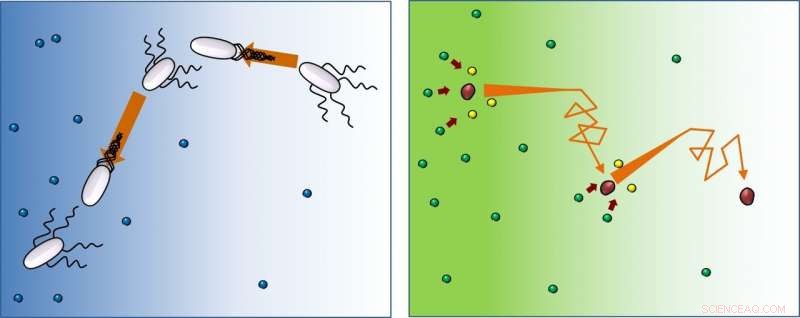
Na presença de comida (azul), bactérias (brancas) nadam em linha reta, em seguida, mude a orientação (queda), e siga esse padrão indefinidamente. Este estudo mostra que as enzimas (vermelhas) se movem da mesma maneira, mas depois de reagir com substratos (de verde para amarelo), eles continuam a correr e cair para longe da maior concentração de substratos. Crédito:IBS
Embora juntar as palavras "aleatório" e "tendencioso" possa parecer um conflito, esses são os atributos que descrevem como as bactérias navegam e buscam seus alimentos. Eles seguem um caminho aleatório, mas inclinado para a direção da fonte de nutrientes. Cientistas do Center for Soft and Living Matter, dentro do Institute for Basic Science (IBS, Coreia do Sul), observou que as enzimas, as moléculas que ajudam as reações biológicas a acontecerem mais rapidamente, mova-se com um padrão semelhante, mas na rota oposta - viajando aleatoriamente em direção à área com menos substratos. Esses resultados, obtido com novas tecnologias ópticas e publicado como um artigo inaugural em Proceedings of the National Academy of Sciences (PNAS) , são particularmente intrigantes, pois constituem um afastamento da visão tradicional.
Milhares de enzimas promovem uma infinidade de reações em nossos corpos, cada um dos quais sustenta nossas vidas. As enzimas agem sobre outros produtos químicos chamados substratos e aceleram as reações químicas que convertem substratos em produtos. Fazer isso, enzimas e substratos devem se encontrar. Originalmente, acreditava-se que as enzimas e seus substratos correspondentes casualmente se chocam:"Os livros-texto padrão mencionam que as enzimas e os substratos devem se aproximar 'de alguma forma, "interagir, e é assim que as reações acontecem. Tentamos nos aprofundar na parte "de alguma forma" desse processo. Depois de coletar milhares de eventos, concluímos que a caminhada das enzimas é enviesada ao acaso, o que significa que eles são microscopicamente direcionais, como bactérias, "explica Ah-Young Jee, primeiro autor deste estudo.
O movimento das bactérias nadadoras é conhecido como "correr e cair":correr, vire e repita. Eles seguem em uma direção, então mude de direção aleatoriamente, e repita. Contudo, enquanto as bactérias se orientam para o suprimento de alimentos, as enzimas se movem na direção de menor concentração de substrato. "As moléculas não têm capacidade de tomada de decisão, mas surpreendentemente mova em direção a áreas com menos substrato. De fato, o substrato introduz o viés de direção, e quanto maior a concentração de substrato que usamos no experimento, mais forte é essa tendência. Nossa hipótese é que pode ser uma maneira de tornar a concentração do produto uniforme em torno do meio, mesmo quando a concentração de substrato não é, "diz o professor Tsvi Tlusty, que ofereceu a compreensão teórica desta pesquisa.
Como a passada de cada enzima tem apenas cerca de 50 nanômetros de comprimento, dispositivos extremamente precisos são necessários para medir sua velocidade e orientação. A equipe de pesquisa, liderado por Steve Granick, olhou para o movimento enzimático com uma tecnologia de microscopia de super-resolução, conhecido como espectroscopia de correlação de fluorescência de depleção de emissão estimulada, ou STED-FCS para breve.

A) Enzima-substrato-tampão (E + S + B) entra na entrada esquerda, e o tampão enzimático (E + B) é injetado na entrada direita. Desta maneira, a concentração da enzima é uniforme em todo o chip, enquanto a concentração de substrato é maior no lado esquerdo (azul) diminuindo gradualmente para a direita (branco). O retângulo de traços indica a área onde o movimento da enzima é estudado em grande detalhe usando a tecnologia de microscopia STED-FCS. B) A concentração da enzima urease (preto) e a concentração de ureia (seu substrato, cinza) são plotados contra a posição dentro da zona FCS, entre 0 e 2,5 milímetros do feixe de laser. A escala em cada ponto é uma barra de erro, mostrando o desvio padrão em cinco medições repetidas. C) O coeficiente de difusão (Da), que indica o quão rápido a enzima está se movendo, não muda na ausência de substrato, mas aumenta quando o substrato está presente, o que significa que a enzima está se movendo mais rapidamente para a direita. Crédito:IBS, publica em PNAS
No método FCS, as enzimas são decoradas com moléculas fluorescentes e sua passagem por um pequeno ponto é detectada por um feixe de laser. As informações estatísticas são coletadas, como o número de enzimas que passam pelo ponto e sua velocidade (taxa de difusão). A equipe complementou o FCS com a tecnologia STED, um dos pontos fortes deste centro de pesquisa. STED concentra o feixe de laser em uma área extremamente pequena, permitindo uma medição mais refinada da posição das enzimas. O feixe de laser FCS cobre uma área de cerca de 250 nanômetros de diâmetro, que é aproximadamente 25 vezes o diâmetro da enzima (10 nanômetros). STED-FCS é mais preciso, pois reduz o diâmetro do feixe para 50 nanômetros, que é comparável à distância da etapa de uma enzima.
Os pesquisadores também projetaram um novo chip microfluídico, formada por canais de espessura micrométrica por onde os líquidos podem passar. Estudos anteriores, usando FCS sem STED e um chip microfluídico diferente, sugeriu que as enzimas se movem em direção à área com a maior concentração de substrato. Contudo, esta nova tecnologia permite observações mais profundas e mostrou o contrário. Os autores acham que os resultados diferem porque as informações detalhadas sobre o movimento da enzima foram canceladas usando um feixe de laser maior. Seria semelhante a localizar alguém dentro de 50 quilômetros quadrados ou dentro de 2 quilômetros quadrados, o último é mais preciso. Eles criaram condições em que a enzima é uniformemente distribuída dentro do chip, mas a concentração de substrato varia da esquerda para a direita:de uma concentração mínima no lado direito do chip, para uma concentração máxima à esquerda. Então, eles usaram FCS para observar a concentração da enzima e a velocidade dentro do chip, e descobriram que a enzima tende a acelerar em direção à área com menor concentração de substrato (ou seja, em direção ao lado direito do chip). Em vez de, quando nenhum substrato está presente, as enzimas não mudam sua velocidade. Além disso, os recursos de super-resolução do STED-FCS mostraram dinâmica de corrida e queda.
A equipe observou o mesmo comportamento com dois pares enzima-substrato separadamente:urease-ureia e acetilcolinesterase (AChE) -acetilcolina. A primeira enzima é usada por alguns microrganismos para transformar a ureia em amônia e ácido carbônico, o último desempenha um papel essencial nas junções neuromusculares.
No futuro, a equipe de pesquisa visa expandir a complexidade do estudo e das condições de teste mais semelhantes à vida real:"Este estudo analisou o comportamento de uma enzima em resposta a um substrato. Em seguida, planejamos examinar várias enzimas ao mesmo tempo. Nosso corpo funciona com uma cascata de reações, o que significa que uma reação desencadeia a seguinte:a enzima A desencadeia a enzima B, então B aciona C, e assim por diante. A análise STED-FCS pode ser uma ferramenta chave para o avanço da pesquisa metabólica, revelando como esses múltiplos sistemas enzimáticos estão organicamente relacionados, "conclui Jee.