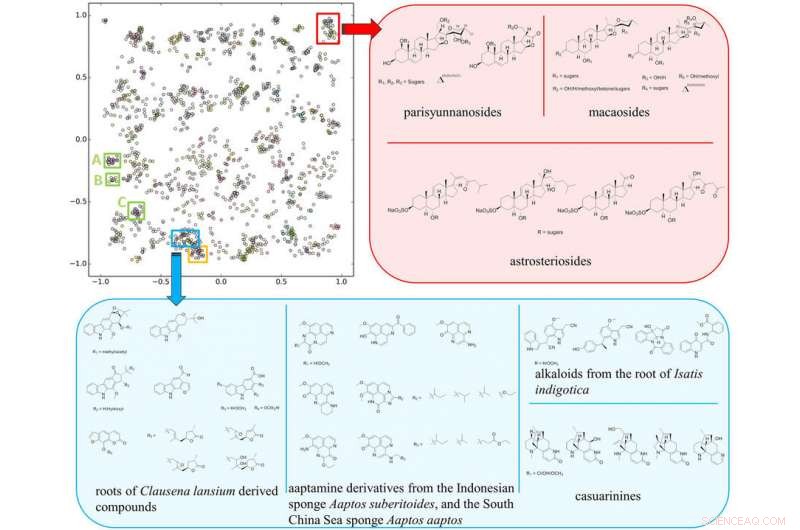
O mapa de cluster SMART com base no resultado do treinamento de 2, 054 espectros HSQC acima de 83, 000 iterações, com caixas inseridas representando diferentes classes compostas discutidas no texto. Crédito:Universidade da Califórnia - San Diego
Uma equipe interdisciplinar de pesquisadores da Universidade da Califórnia em San Diego desenvolveu um método para identificar as estruturas moleculares de produtos naturais que é significativamente mais rápido e preciso do que os métodos existentes. O método funciona como o reconhecimento facial de estruturas moleculares:ele usa um pedaço de dados espectrais exclusivos para cada molécula e, em seguida, o executa por meio de uma rede neural de aprendizado profundo para colocar a molécula desconhecida em um agrupamento de moléculas com estruturas semelhantes.
O novo sistema é denominado "SMART, "que significa tecnologia de reconhecimento preciso de moléculas pequenas, e tem o potencial de acelerar dez vezes o processo de identificação da estrutura molecular. Este desenvolvimento pode representar uma mudança de paradigma na análise química, campos farmacêuticos e de descoberta de medicamentos, uma vez que 70 por cento de todos os medicamentos aprovados pela Food and Drug Administration (FDA) são baseados em produtos naturais, como microrganismos do solo, plantas terrestres e, cada vez mais, formas de vida marinha, como algas.
"A estrutura de uma molécula é a informação capacitadora, "disse Bill Gerwick, professor de oceanografia e ciências farmacêuticas na Scripps Institution of Oceanography da UC San Diego. "Você tem que ter a estrutura para qualquer aprovação do FDA. Se você quiser ter propriedade intelectual, você tem que patentear essa estrutura. Se você quiser fazer análogos dessa molécula, você precisa saber qual é a molécula inicial. É uma informação crítica. "
Chen Zhang, um Ph.D. em nanoengenharia estudante da UC San Diego colaborando com Gerwick e o primeiro autor do artigo publicado em Nature Scientific Reports , disse que determinar a estrutura de uma molécula pode ser um gargalo no processo de pesquisa de produto natural, levando meses e até anos para os especialistas determinarem com precisão a estrutura correta e completa. Embora cada molécula e sua linha de tempo de identificação sejam diferentes, a abordagem SMART dá aos pesquisadores uma pista inicial sobre em qual família uma nova molécula se enquadra, reduzindo drasticamente o tempo que leva para caracterizar um novo produto natural.
"A forma como fomos capazes de acelerar o processo é essencialmente usando um software de reconhecimento facial para observar a informação-chave que obtemos sobre as moléculas, "Gerwick disse. A peça-chave de informação que a equipe usa é chamada de ressonância magnética nuclear de coerência quântica singular heteronuclear, ou HSQC NMR, espectro. Ele produz um mapa topológico de pontos que revelam quais prótons na molécula estão ligados diretamente a quais átomos de carbono, um arranjo único para cada molécula.
Zhang e Gerwick se juntaram a Gary Cottrell, professor de ciência da computação e engenharia na Escola de Engenharia da UC San Diego Jacobs, para desenvolver um sistema de aprendizado profundo treinado com milhares de espectros HSQC extraídos de pesquisas anteriores. Esta rede neural convolucional pega uma imagem 2-D do espectro HSQC NMR de uma molécula desconhecida e mapeia em um espaço de 10 dimensões agrupado perto de moléculas semelhantes, tornando mais fácil para os pesquisadores elucidar a estrutura de uma molécula desconhecida.
"Chen adotou essa abordagem para obter espectros de NMR de mais de 4, 000 compostos da literatura, cortando literalmente as imagens dos PDFs dos documentos, "Cottrell disse." Foi um esforço incrível! Mesmo assim, normalmente não são dados suficientes para treinar uma rede profunda, mas usamos uma tecnologia chamada rede siamesa, em que você treina em pares de imagens. Isso amplifica seu treinamento definido por aproximadamente o quadrado do número de compostos em uma família, e é o que tornou este projeto viável. "
Esta colaboração é a primeira vez que Gerwick orientou um estudante de engenharia, e a troca de ideias foi frutífera.
"Tem sido uma interação maravilhosa. UC San Diego tem algo realmente mágico sobre isso, e essa é a profundidade da colaboração que ocorre entre os departamentos - é fenomenal, "Gerwick disse." Quando você tenta e cuidadosamente tira de outra disciplina algo que talvez seja até comum naquela disciplina e aplica de uma maneira nova e única em nossa disciplina, é uma oportunidade de realmente ter esse tipo de mudança de paradigma. E eu acho que essa tecnologia, com algum avanço, pode ser uma verdadeira mudança de paradigma na maneira como fazemos todos os tipos de química e análise química. "