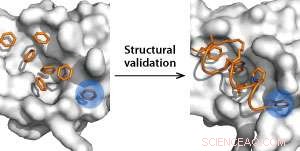
Detecção de bolso de ligação usando moléculas de benzeno como sondas (esquerda) e validação estrutural usando peptídeos grampeados (direita). Crédito:Y. S. Tan et al.
Na busca por novas terapias contra o câncer, Os pesquisadores do A * STAR desenvolveram uma estratégia computacional que desenterra quaisquer sítios de ligação previamente desconhecidos ou 'bolsões' em alvos de drogas.
Tratamentos de câncer mais eficazes provavelmente surgirão a partir do desenvolvimento de medicamentos. A descoberta de drogas contra o câncer depende da identificação e caracterização de bolsas de ligação em proteínas-alvo. Tipicamente, esta avaliação usa técnicas computacionais que dependem de estruturas de proteínas estáticas. Contudo, as proteínas têm uma flexibilidade inerente que causa uma tendência a mudar de forma ao entrar em contato com as drogas. Certos bolsos de ligação permanecem indetectáveis, a menos que interajam com uma substância apropriada e, Portanto, são perdidos por simulações convencionais. Esses bolsos escondidos, Contudo, são geralmente locais hidrofóbicos ou repelentes de água que só se abrem quando há substâncias de baixa polaridade.
Para resolver isso, Yaw Sing Tan e Chandra Verma do Instituto de Bioinformática desenvolveram um método baseado em sonda chamado dinâmica molecular de mapeamento de ligante (LMMD). Eles usaram essa técnica para procurar bolsos de ligação ocultos na proteína alvo anticâncer MDM2. As previsões resultantes foram validadas experimentalmente por colaboradores de longa data do A * STAR's p53 Laboratory e Institute of Chemical and Engineering Sciences, bem como biólogos estruturais da Newcastle University, REINO UNIDO.
Tan explica que inicialmente ele projetou este método baseado em sonda para outra proteína alvo e o usou com sucesso para encontrar uma bolsa de ligação oculta que permanecia fechada nas simulações convencionais. "Decidimos então aplicar esta abordagem ao MDM2 para ver se poderíamos descobrir quaisquer locais de ligação anteriormente desconhecidos que pudessem aumentar a potência dos inibidores de MDM2 existentes, " ele adiciona.
Usando moléculas de benzeno como sondas hidrofóbicas de detecção de bolso, os pesquisadores identificaram computacionalmente dois novos locais de ligação no MDM2. "Ficamos entusiasmados em ver que esses locais ficam muito próximos da bolsa de ligação da proteína supressora de tumor p53, "diz Tan.
Além disso, os pesquisadores esperam que os locais recém-descobertos levem a peptídeos grampeados mais potentes - são hélices de aminoácidos quimicamente estabilizadas por uma cadeia de hidrocarbonetos que recentemente emergiram como poderosos ativadores de p53. Consequentemente, eles criaram peptídeos grampeados de análogos conhecidos por se ligarem fortemente ao MDM2 e reativar o p53, e determinou a afinidade desses peptídeos para MDM2. Suas simulações mostraram que os peptídeos se ligaram ao MDM2 mais fortemente do que o p53 nos bolsos e combinaram experimentos de cristalografia biofísica e de raios-X.
"Este método pode ser usado para interrogar outros alvos proteicos anticâncer para descobrir novos locais de ligação que podem ser alvo de inibição, "diz Tan. A equipe agora está trabalhando para expandir o alcance das sondas LMMD para outros tipos de ligantes.