Durante a replicação, os dois fios são separados. Cada fita da molécula de DNA original serve então como um modelo para a produção de sua contraparte complementar. Crédito:Wikimedia Commons
A análise do DNA é útil para várias aplicações vitais. Isso inclui diagnóstico e monitoramento de doenças, identificação de criminosos, e estudar a função de um segmento-alvo de DNA. Contudo, os métodos usados para análises geralmente requerem mais DNA do que pode estar disponível em uma amostra típica. 'Portanto, a amplificação é necessária, mas nem sempre direto. O método de amplificação ou fotocópia mais utilizado é a reação em cadeia da polimerase (PCR). Um novo método de PCR pode ajudar no processo de amplificação, e, assim, desenvolver ensaios robustos que anteriormente não seriam possíveis.
O ácido desoxirribonucléico (DNA) é uma molécula encontrada no núcleo das células e carrega as 'instruções' para o desenvolvimento e funcionamento dos organismos vivos. Muitas vezes, é comparado a um conjunto de projetos, pois contém as instruções necessárias para construir células. Essas instruções são divididas em segmentos ao longo de uma fita de DNA.
Replicação
O DNA é composto por uma dupla hélice de duas fitas complementares feitas de nucleotídeos, uma estrutura frequentemente comparada a uma escada. Quando é hora de replicar, as duas fitas de DNA - ou "lados" da escada - se desenrolam e se separam. Uma enzima chamada DNA polimerase lê as fitas individuais e combina as bases complementares - os 'degraus' da escada - à fita original. Novas fitas são produzidas em ambos os lados do DNA original, fazendo duas hélices duplas de DNA idênticas compostas por uma fita original e uma nova. Este processo ocorre em todos os organismos vivos e é a base para a herança biológica.
PCR
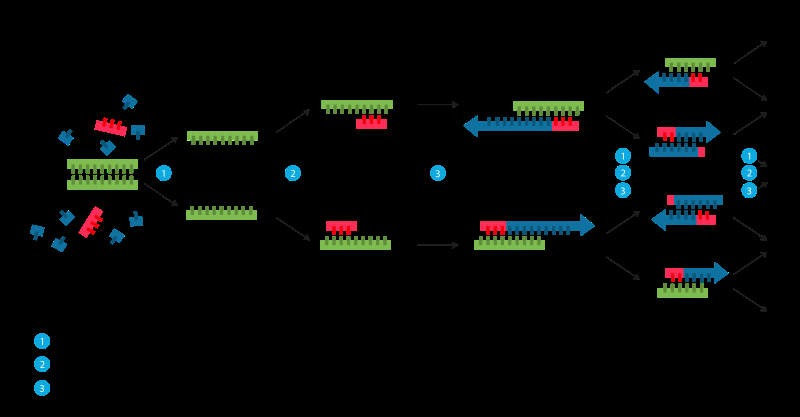
A replicação também pode ser processada artificialmente. Às vezes chamado de "fotocópia molecular, "a reação em cadeia da polimerase (PCR) é uma técnica rápida e relativamente barata usada para amplificar, ou fazer muitas cópias de, pequenos segmentos de DNA. Isso é necessário porque os métodos usados para analisar o DNA ou determinar a sequência do par de bases do DNA requerem mais DNA do que pode haver em uma amostra típica. Portanto, o objetivo do PCR é amplificar uma região específica de uma fita de DNA.
Para realizar PCR, as duas fitas da dupla hélice do DNA são fisicamente separadas pela aplicação de alta temperatura (93-98 ° C) em um processo chamado fusão do DNA. Na segunda etapa, a temperatura é reduzida e as duas fitas de DNA, agora separado, tornam-se modelos para os chamados primers. Os primers são moléculas curtas de DNA de fita simples, complementar à região de DNA alvo para amplificação. Depois de baixar a temperatura, os primers adicionados ao processo de PCR encontram e se ligam ao seu alvo específico. Uma enzima chamada DNA polimerase alonga - ou constrói - uma nova fita de DNA a partir do primer, usando a molécula de DNA de fita simples subjacente como molde.
Construindo uma escada
COMPAS-PCR usando primers altamente complementares aplicados a genes de repetição direta de DNA ribossomal 5S, conforme mostrado neste exemplo. Crédito:Instituto Norueguês de Pesquisa Hídrica (NIVA)
Como acima mencionado, os primers são moléculas curtas de DNA de fita simples. Eles geralmente têm cerca de 20 nucleotídeos de comprimento. Esta cadeia de nucleotídeos, anexa-se especificamente ao início da fita do molde por emparelhamento de bases - encontrando as bases complementares. A DNA polimerase é então capaz de adicionar o próximo nucleotídeo complementar. A polimerase continua adicionando mais nucleotídeos complementares ao DNA molde até que uma nova fita dupla de DNA seja concluída, ou para usar a metáfora acima; uma nova escada é construída, com um lado original e um novo lado.
Os primers duplos permitem definir com precisão a área de uma molécula de DNA a ser amplificada pela PCR. Esses dois primers de flanqueamento especificam onde a nova cadeia deve começar e terminar.
Uma pequena mudança de paradigma
Uma regra central fortemente ancorada na mente do cientista molecular é que os primers devem ser complementares apenas à sequência alvo, e não uns com os outros. Isso evita que os primers usem uns aos outros como modelos e, assim, desabilitem um possível envolvimento adicional na amplificação do alvo tradicional.
Em um estudo recente, publicado na revista científica PLOS One , O cientista pesquisador sênior Marc Anglès d´Auriac do Instituto Norueguês de Pesquisa sobre a Água (NIVA) mostra que é possível usar primers altamente complementares e ainda evitar as conseqüências infelizes mencionadas acima. O novo método foi denominado COMPAS-PCR, abreviação de COMplementary Primer Asymmetric PCR.
Resumidamente, Anglès d'Auriac observou que primers complementares entre si e um alvo (overlay triplo) ainda podiam definir um produto de amplificação quando parte de um motivo ou estrutura de DNA repetido. Isso é mostrado na figura abaixo. Avançar, o obstáculo físico da amplificação do alvo devido à complementaridade do iniciador foi aliviado pela introdução de concentrações de iniciador assimétricas. Assimétrico a este respeito significa um número ímpar de moléculas entre os dois primers. Um primer consiste em um baixo número de moléculas, o outro com um número alto.
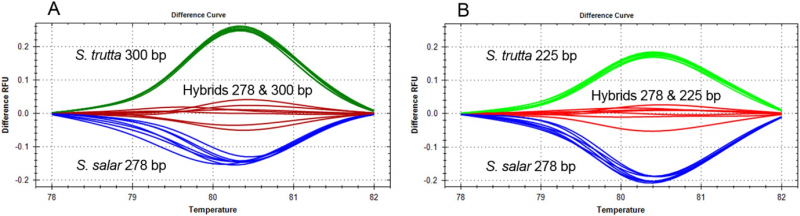
Análise de fusão de alta resolução de dois COMPAS-PCR duplex de três iniciadores para a diferenciação de S. salar, S. trutta e híbridos. Crédito:Instituto Norueguês de Pesquisa Hídrica (NIVA)
"Se estiver usando concentrações iguais, como você costuma fazer para PCR, os primers, presentes em quantidades iguais, ficarão um no outro, "Anglès d'Auriac diz.
"Com concentrações assimétricas, o primer em excesso ou altamente concentrado tem um número de moléculas não presas ao primer limitante, e, portanto, está disponível para amplificação de alvo. "
Trabalhando sozinho
"Esta abordagem contra-intuitiva irá isolar, mas também protege, o primer de baixa concentração, "Anglès d'Auriac diz.
Com o primer limitador preso, e assim protegido, é possível que o primer de alta concentração funcione sozinho, isso é, faça uma cópia de fita única do DNA-alvo. Quando amplicons de fita simples suficientes são produzidos, eles podem servir como modelo para o primer de baixa concentração. O primer limitador é liberado, e a reação muda para amplificação por PCR exponencial clássica.
"Isso amplia as possibilidades de aplicação de PCR para o cientista, Anglès d'Auriac elucida. "
Estruturas repetitivas
Reação em cadeia da polimerase (PCR). Crédito:Wikimedia
Em muitos organismos, uma fração significativa do DNA genômico é altamente repetitiva, com mais da metade da sequência consistindo de elementos repetitivos em humanos.
Foi realmente uma estrutura de DNA repetitiva que fez Anglès d'Auriac pensar fora da caixa e propor o princípio COMPAS-PCR, uma pequena mudança de paradigma no campo da fotocópia molecular.
No processo de realização de diagnóstico baseado em DNA para salmonídeos, em particular a identificação da truta marrom intimamente relacionada (Salmo trutta) e do salmão do Atlântico (Salmo salar), Anglès d'Auriac lutou para separar essas espécies - incluindo híbridos entre elas. A identificação rápida e precisa ajudaria a melhorar o monitoramento e os estudos do ecossistema do rio, como a identificação de híbridos é importante para estimar a saúde ecológica das bacias hidrográficas. Depois de usar COMPAS-PCR, Anglès d'Auriac foi capaz de aplicar um método de análise de produto de PCR chamado análise de fusão de alta resolução na identificação de truta marrom, Salmão do Atlântico e seus híbridos em um teste.
Anglès d'Auriac enfatiza, no entanto, que o princípio COMPAS-PCR não se restringe à identificação de espécies de peixes. O uso de primers quase totalmente complementares direcionados à mesma sequência pode se aplicar a quaisquer cópias adjacentes umas às outras, em motivos de DNA de interesse como sequências alvo.
"As sequências de repetição de DNA são relatadas como constituindo mais de 50% do genoma humano e estão presentes em muitas famílias de genes" domésticos ", como o gene ribossômico 5S usado neste estudo. os princípios gerais do COMPAS-PCR ajudarão a desenvolver novas estratégias de amplificação de DNA aproveitando essas estruturas de DNA repetidas, "Marc Anglès d'Auriac conclui.