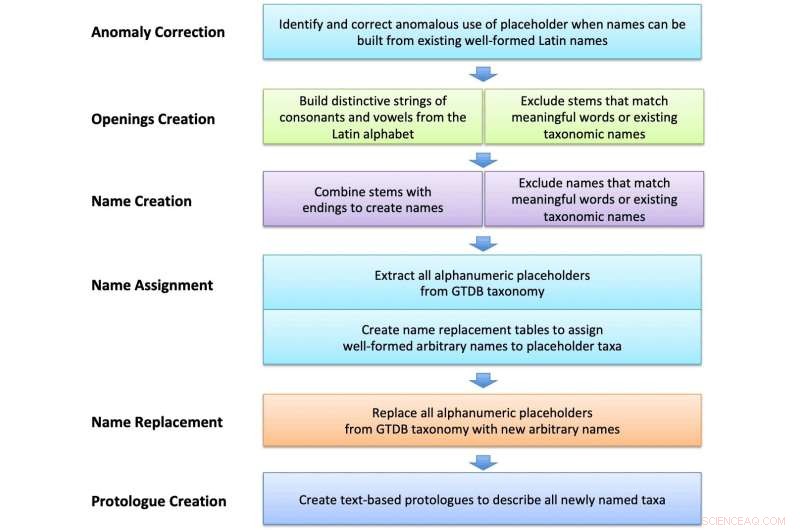
Fluxo de trabalho esquemático para criação e atribuição de nomes latinos arbitrários. Strings construídas a partir de letras latinas foram selecionadas e selecionadas para excluir componentes indesejados e palavras significativas. Estes foram combinados com sufixos latinos ou palavras para criar nomes, que foram então usados para nomear taxa placeholder em arquivos GTDB e criar protólogos baseados em texto. Crédito:Jornal Internacional de Microbiologia Sistemática e Evolutiva (2022). DOI:10.1099/ijsem.0.005482
Em um artigo publicado recentemente no
International Journal of Systematic and Evolutionary Microbiology , pesquisadores do Reino Unido e da Áustria nomearam mais de 65.000 tipos diferentes de micróbios. O estudo, liderado pelo professor Mark Pallen, do Quadram Institute, em Norwich, se baseia em uma longa tradição de criar nomes latinos bem formados, mas arbitrários, para novas espécies, mas aplica essa abordagem em uma escala sem precedentes na história da taxonomia.
O Prof. Pallen argumenta que a microbiologia é vítima de seu próprio sucesso, com dezenas de milhares de novas espécies descobertas nos últimos anos, mas a maioria permanece sem nome. No passado, as bactérias receberam nomes descritivos ou receberam nomes de pessoas ou lugares. Atualmente, essa abordagem está fornecendo cerca de mil novos nomes de espécies por ano. No entanto, com um acúmulo de mais de 50.000 espécies bem classificadas, mas sem nome, nesse ritmo de progresso, levaria pelo menos meio século para nomear todas essas bactérias sem nome - quando os cientistas enfrentariam o problema de nomear os milhões de espécies a mais. descoberto nesse meio tempo.
Pallen e seus colegas adotaram uma abordagem de big data eficiente, de alto rendimento, usando um programa de computador para gerar dezenas de milhares de nomes distintos, mas fáceis de usar, que não têm nenhuma semelhança com nenhuma palavra existente. Para cumprir a exigência de que os nomes das bactérias sejam em latim, a equipe combinou sequências arbitrárias de letras do alfabeto latino com sufixos femininos gramaticalmente bem formados. O resultado é um conjunto de nomes que lembram a familiaridade e a seriedade do latim, embora careçam de um pedigree significativo. Exemplos incluem Dupisella tifacia para uma bactéria do intestino de ovelha, Hopelia gocarosa para uma bactéria que vive em águas subterrâneas suecas ou Saxicetta apufaria de um lago salgado russo.
Embora isso possa parecer radical, na verdade a formação de nomes de forma arbitrária tem uma longa tradição, que remonta ao primeiro código de nomenclatura taxonômica em 1869 e até mesmo ao pai da taxonomia, o naturalista sueco Linnaeus. O que é diferente aqui é o surpreendente senso de escala, com um catálogo de nomes que se estende por mais de dez mil páginas no que representa a maior nomeação de espécies em uma única publicação.
Como eles foram aplicados a espécies descobertas por sequenciamento de DNA, mas ainda não cultivadas em laboratório, por enquanto os novos nomes permanecem provisórios, e não permanentes. No entanto, como o código de nomenclatura insiste que os bacteriologistas visam a estabilidade nos nomes e não há abordagens concorrentes de nomenclatura em escala, parece provável que a grande maioria dos novos nomes seja usada nos próximos anos ou mesmo séculos.
Para encerrar, Pallen diz:"Fui membro do grupo de trabalho que nos deu letras gregas para variantes de COVID, que foram rapidamente adotadas pela comunidade científica. Espero que os nomes propostos aqui também sejam rapidamente adotados e amplamente utilizados. Isso é apenas o primeiro passo. A era da descoberta microbiana está longe de terminar, mas será fácil criar nomes futuros em massa usando os princípios que estabelecemos aqui."
+ Explorar mais Um sistema automatizado para gerar um milhão de novos nomes para bactérias
