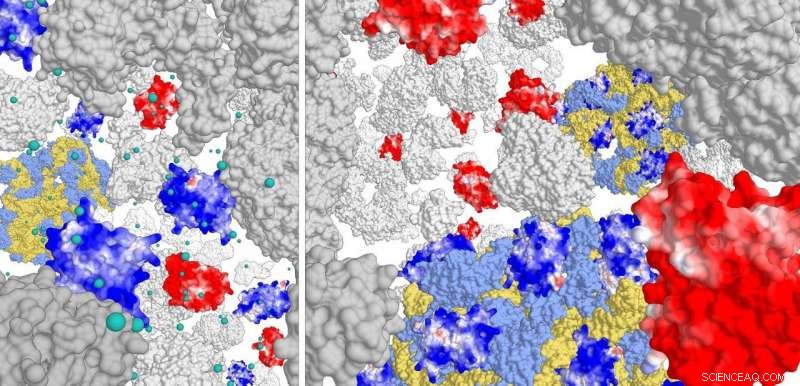
Painel direito:interação de proteínas carregadas positivamente (azul escuro) com o complexo ribossômico (azul claro / amarelo). Proteínas carregadas negativamente não interagem. Em alta força iônica (painel esquerdo), as proteínas positivas dificilmente interagem com o ribossomo. Crédito:Poolman lab, Universidade de Groningen
As células estão repletas de macromoléculas, que limita a difusão de proteínas, especialmente em células procarióticas sem transporte ativo no citoplasma. Ao investigar a relação entre aglomeração, força iônica e difusão de proteínas, Os bioquímicos da Universidade de Groningen fizeram uma descoberta fascinante:proteínas com carga positiva aderem à superfície dos complexos de ribossomos. Isso explica por que a maioria das proteínas solúveis em água carregam uma carga geral negativa. A descoberta aparecerá em breve no jornal eLife .
A velocidade de movimento das proteínas dentro das células é importante:muitos processos em células biológicas dependem de interações entre macromoléculas (proteínas e ácidos nucléicos) e, portanto, de sua capacidade de se encontrarem. 'Mas o citoplasma da célula é um lugar movimentado e isso afetará a difusão de proteínas e RNA', observa o professor de bioquímica da Universidade de Groningen, Bert Poolman.
Cobranças
Seu grupo estudou os efeitos da aglomeração na difusão, e encontraram uma correlação entre o tamanho da proteína e a velocidade de difusão. 'Mas para algumas proteínas não encontramos esta correlação, então começamos a investigar o porquê. ' A equipe usou três procariontes diferentes com força iônica crescente:a bactéria Gram-negativa Escherichia coli, o Lactococcus lactis Gram-positivo e o extremófilo Haloferax volcanii, que vive em concentrações de sal muito altas.
Para este estudo, os pesquisadores construíram diferentes variantes de Green Fluorescent Protein (GFP), com cargas de superfície variando de -30 a +25. Eles então estudaram o movimento dessas variantes GFP nos três tipos de células. 'Vimos que proteínas carregadas positivamente se difundem muito lentamente. Eles ficaram presos na cela ', explica Poolman. Uma análise posterior mostrou que as proteínas positivas não se ligavam ao DNA ou à membrana celular, mas ao complexo ribossômico.
Interessante
Uma análise de bioinformática dos proteomas de microrganismos e células eucarióticas mostrou que, na maioria dos casos, cerca de 70 por cento das proteínas têm carga negativa. 'Interessantemente, os 30% restantes são proteínas de membrana ou proteínas envolvidas no funcionamento ou dobramento do ribossomo ou mRNA. '
As proteínas da membrana são protegidas por chaperones durante a biogênese, para que eles não grudem nos ribossomos. Portanto, não há proteínas citoplasmáticas "livres" com uma carga positiva alta o suficiente para fazê-las se estabelecer nos ribossomos. A carga negativa do complexo ribossômico e a força iônica ambiente do citoplasma parecem ter moldado a evolução das cargas no proteoma celular.
Inesperado
A nova e inesperada descoberta de que a mobilidade da proteína é uma função da carga da proteína pode explicar por que é difícil expressar algumas proteínas em sistemas bacterianos com baixa força iônica. 'Observamos que uma força iônica mais alta reduz a viscosidade de proteínas carregadas positivamente. Isso poderia ser um insight valioso para a construção de plataformas de expressão de proteínas. '
Uma observação final no artigo eLife é que os genomas de vários endossimbiontes mostram uma abundância de proteínas carregadas positivamente. 'Esta descoberta realmente nos confunde', admite Poolman. “Você esperaria que todas essas proteínas fossem atraídas pelos ribossomos dos endossimbiontes. Até aqui, não temos nenhuma explicação de como esses organismos são capazes de lidar com a difusão lenta e os ribossomos sendo engolfados por proteínas positivas. '