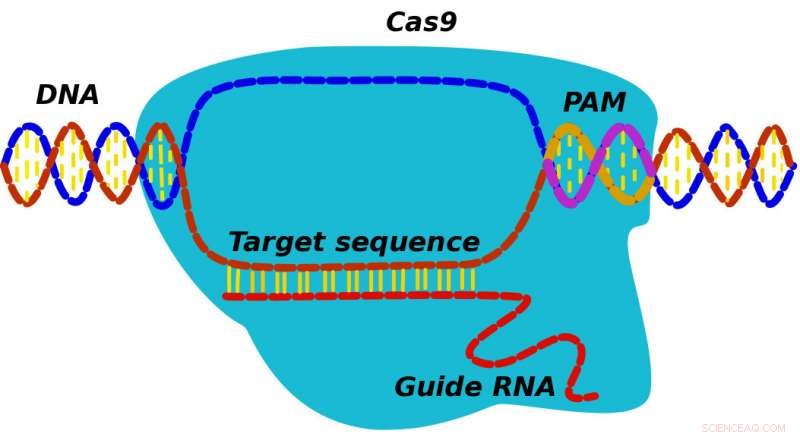
Quando uma proteína CRISPR-Cas9 encontra seu alvo, primeiro encontra uma sequência espaçadora conhecida como PAM e, em seguida, pesquisa DNA adjacente para ver se corresponde ao RNA guia de Cas9. Um novo modelo desenvolvido por pesquisadores da Rice University pode ajudar a descobrir detalhes sobre o mecanismo pelo qual CRISPR-Cas9 pode substituir mutações por DNA correto. Crédito:Alexey Shvets / Rice University
Os pesquisadores da Rice University desenvolveram um modelo computacional para quantificar o mecanismo pelo qual as proteínas CRISPR-Cas9 encontram seus alvos de edição de genoma.
Anatoly Kolomeisky, um professor de química e engenharia química e biomolecular de Rice, e o ex-aluno Alexey Shvets adaptou um sistema que desenvolveram anteriormente para mostrar como as proteínas geralmente encontram seus alvos biológicos. Eles esperam que o modelo revisado ajude a desvendar os mistérios restantes do CRISPR.
Em seu estado natural, CRISPR, que significa "repetições palindrômicas curtas com espaçamento regular agrupado, "é o mecanismo biológico pelo qual as bactérias se protegem das infecções virais. As bactérias incorporam uma cópia do DNA estranho e constroem um registro de todos aqueles que invadem. Elas se referem a esse registro quando novos invasores são detectados e o usam para destruí-los.
Nos últimos anos, pesquisadores começaram a adaptar o mecanismo para uso na edição do genoma, que tem o potencial de curar doenças e melhorar os organismos, incluindo humanos. Mas um obstáculo tem sido o risco de que as proteínas CRISPR-Cas9, um dos sistemas que utilizam a abordagem CRISPR, cortará e substituirá as sequências de destino erradas, introdução de mutações.
O modelo de arroz descrito no Biophysical Journal descobriram que é provável que CRISPR-Cas9 localize bons alvos de forma mais eficiente quando essas edições fora do alvo podem acontecer, porque as proteínas não perdem tempo se desassociando dos alvos fora do alvo para continuar procurando.
Isso pode ou não ser uma coisa boa, mas certamente vale a pena estudar, Kolomeisky disse.
"A taxa de erro (corte fora do alvo) às vezes é de 10-20 por cento, ", disse ele." Temos duas ideias sobre isso:uma é que os vírus sofrem mutação muito rapidamente e talvez as bactérias estejam tentando cortar alvos que sofreram mutação apenas leve, como forma de serem mais flexíveis. A outra é que existem proteínas que podem corrigir erros, então, se não houver muitos cortes errados, o sistema pode tolerá-los.
Kolomeisky disse que seu modelo é um passo simples para descobrir a dinâmica da edição do CRISPR. "CRISPR-Cas9 é a variação mais popular porque tem apenas uma proteína e é mais fácil, biologicamente, trabalhar com, " ele disse.
O laboratório Rice desenvolveu seu modelo original para aprender como as proteínas deslizam ao longo do DNA para encontrar alvos e desencadear processos como a transcrição de genes. Kolomeisky observou que a pioneira do CRISPR, Jennifer Doudna, descobriu que o CRISPR-Cas9 não busca da mesma forma. "Ela descobriu que não desliza em nenhum lugar do DNA, " ele disse.
Em vez de, de acordo com Doudna e sua equipe, a proteína reconhece inicialmente as sequências PAM de três nucleotídeos (para o motivo adjacente do protoespaçador) que marcam a localização de alvos potenciais. "CRISPR encontra e se liga a PAM e, em seguida, seu RNA associado explora o DNA adjacente para ver se este é o alvo, "Kolomeisky disse." Se for, a proteína começa a cortar. Se não, ele se desassocia e olha para outro lugar. "
Nos experimentos subsequentes de Doudna com sequências PAM removidas, As proteínas CRISPR-Cas9 não conseguiram encontrar seus alvos. Portanto, os PAMs têm um papel importante e não são apenas um espaçador genérico, ele disse. "Assim que li isto, Eu entendi que poderíamos usar nosso modelo aqui também. "
O modelo teórico analisa os processos de primeira passagem - aqueles que acontecem quando um sistema cruza um limite físico ou químico, como encontrar um PAM relevante - para rastrear proteínas CRISPR-Cas9 inseridas em uma célula à medida que pesquisam as sequências de PAM e, em seguida, enquanto ligado a PAMs, procure o alvo de DNA que corresponda ao RNA do Cas9.
Eles descobriram que os CRISPRs que evitam cortes fora do alvo ao se desassociar do DNA "errado" levam mais tempo para se estabilizar do que aquele que simplesmente corta os alvos. "Indo para o PAM errado leva tempo, "Kolomeisky disse." Nosso cálculo mostra que o CRISPR pode encontrar alvos reais mais rápido quando às vezes corta nos lugares errados. A fração que vai para os alvos certos pode ser menor, mas você irá cortá-los eventualmente.
"É um modelo simples e perfeitamente solucionável, "Disse Kolomeisky." Se alguém quiser testar, o modelo pode fornecer previsões específicas e, em alguns casos, oferecer tendências para o que deve ser observado. "O que falta no modelo é a capacidade de ver se a chave de RNA reconhece seu alvo simultaneamente - ligando-se ao DNA de uma só vez - ou sequencialmente, nucleotídeo por nucleotídeo.
“O mais impressionante sobre o CRISPR não é a descoberta de um sistema imunológico nas bactérias, mas o fato de que isso criou uma revolução na biotecnologia, porque significa que em qualquer célula podemos cortar qualquer DNA em um local específico, muito precisamente, "Kolomeisky disse." Espero que nosso trabalho estimule estudos mais fundamentais, porque gosto muito do método CRISPR. Mas não fico feliz quando as pessoas o aplicam sem entender como funciona no nível molecular. "
Shvets é agora um pesquisador de pós-doutorado no Instituto de Tecnologia de Massachusetts. Kolomeisky é professor de química e de engenharia química e biomolecular.