Um modelo de uma miniproteína projetada computacionalmente de um estudo em grande escala do UW Medicine Institute for Protein Design. Crédito:UW Medicine Institute for Protein Design
O cenário está armado para uma nova era de engenharia molecular de proteínas orientada por dados, à medida que os avanços na tecnologia de síntese de DNA se fundem com as melhorias no design computacional de novas proteínas.
Desta semana Ciência relata o teste em maior escala de estabilidade de dobramento para proteínas projetadas computacionalmente, possibilitado por uma nova abordagem de alto rendimento.
Os cientistas são do UW Medicine Institute for Protein Design da Universidade de Washington em Seattle e da Universidade de Toronto em Ontário.
O principal autor do artigo é Gabriel Rocklin, um pós-doutorado em bioquímica na University of Washington School of Medicine. Os autores sênior são Cheryl Arrowsmith, do Princess Margaret Cancer Center, o Structural Genomics Consortium e o Departamento de Biofísica Médica da Universidade de Toronto, e David Baker, Professor de bioquímica da UW e investigador do Howard Hughes Medical Institute.
As proteínas são burros de carga biológicos. Os pesquisadores querem construir novas moléculas, não encontrado naturalmente, que pode realizar tarefas na prevenção ou tratamento de doenças, em aplicações industriais, na produção de energia, e em limpezas ambientais.
"Contudo, proteínas projetadas computacionalmente muitas vezes falham em formar as estruturas dobradas que foram projetadas para ter quando são realmente testadas em laboratório, "Rocklin disse.
No último estudo, os pesquisadores testaram mais de 15, 000 mini-proteínas recém-projetadas que não existem na natureza para ver se formam estruturas dobradas. Mesmo os principais estudos de design de proteínas nos últimos anos examinaram geralmente apenas 50 a 100 designs.
"Aprendemos muito nesta nova escala, mas o sabor nos deu um apetite ainda maior, "disse Rocklin." Estamos ansiosos para testar centenas de milhares de projetos nos próximos anos. "
O teste mais recente levou ao design de 2, 788 estruturas de proteínas estáveis e podem ter muitas aplicações de bioengenharia e biologia sintética. Seu tamanho pequeno pode ser vantajoso para o tratamento de doenças quando a droga precisa atingir o interior de uma célula.
As proteínas são feitas de cadeias de aminoácidos com sequências específicas, e as sequências de proteínas naturais são codificadas no DNA celular. Essas cadeias se dobram em conformações tridimensionais. A sequência de aminoácidos na guia da cadeia onde ela se dobrará e se torcerá, e como as partes irão interagir para manter a estrutura unida.
Por décadas, os pesquisadores estudaram essas interações examinando as estruturas das proteínas que ocorrem naturalmente. Contudo, estruturas de proteínas naturais são tipicamente grandes e complexas, com milhares de interações que coletivamente mantêm a proteína em sua forma dobrada. Medir a contribuição de cada interação se torna muito difícil.
Os cientistas resolveram esse problema projetando computacionalmente os seus próprios, proteínas muito mais simples. Essas proteínas mais simples tornaram mais fácil analisar os diferentes tipos de interações que mantêm todas as proteínas em suas estruturas dobradas.
"Ainda, até mesmo proteínas simples são tão complicadas que era importante estudar milhares delas para aprender por que se dobram, "Rocklin disse." Isso tinha sido impossível até recentemente, devido ao custo do DNA. Cada proteína projetada requer seu próprio pedaço de DNA personalizado para que possa ser feito dentro de uma célula. Isso limitou os estudos anteriores a testar apenas dezenas de designs. "
Para codificar seus designs de proteínas curtas neste projeto, os pesquisadores usaram o que é chamado de tecnologia de síntese de biblioteca de oligo de DNA. Foi originalmente desenvolvido para outros protocolos de laboratório, como a grande montagem de genes. Uma das empresas que forneceram seu DNA é a CustomArray em Bothell, Wash. Eles também usaram bibliotecas de DNA feitas pela Agilent em Santa Clara, Califórnia, e Twist Bioscience em San Francisco.
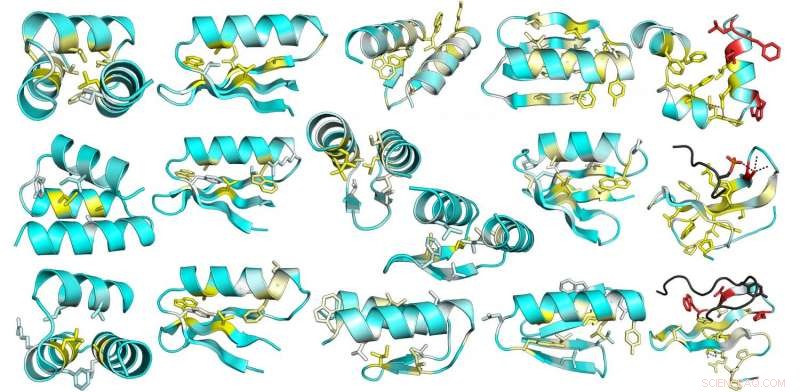
Esta imagem é de uma análise mutacional abrangente de estabilidade em proteínas naturais e projetadas. A mudança média na estabilidade devido à mutação de cada posição em 13 proteínas projetadas é representada nas estruturas do modelo de design. Amarelo indica posições onde as mutações são mais desestabilizadoras; as posições onde há pouco efeito são azuis. Crédito:UW Medicine Institute for Protein Design
Ao repetir o ciclo de computação e teste experimental em várias iterações, os pesquisadores aprenderam com suas falhas de projeto e aprimoraram progressivamente sua modelagem. A taxa de sucesso do projeto aumentou de 6% para 47%. Eles também produziram proteínas estáveis em formatos em que todos os seus primeiros designs falharam.
Seu grande conjunto de mini-proteínas estáveis e instáveis permitiu que analisassem quantitativamente quais características de proteínas se correlacionavam com o enovelamento. Eles também compararam a estabilidade de suas proteínas projetadas com tamanhos semelhantes, proteínas de ocorrência natural.
A proteína natural mais estável que os pesquisadores identificaram foi uma proteína muito estudada da bactéria Bacillus stearothermophilus. Este organismo se aquece em altas temperaturas, como aqueles em fontes termais e fontes termais oceânicas. A maioria das proteínas perde suas estruturas dobradas sob tais condições de alta temperatura. Os organismos que prosperam lá desenvolveram proteínas altamente estáveis que permanecem dobradas mesmo quando quentes.
"Um total de 774 proteínas projetadas tinham índices de estabilidade mais elevados do que esta proteína monomérica mais resistente à protease, "observaram os pesquisadores. As proteases são enzimas que quebram as proteínas, e foram ferramentas essenciais que os pesquisadores usaram para medir a estabilidade de seus milhares de proteínas.
Os pesquisadores prevêem que, à medida que a tecnologia de síntese de DNA continua a melhorar, projeto de proteína de alto rendimento se tornará possível para maiores, estruturas de proteínas mais complexas.
"Estamos nos afastando do velho estilo de design de proteínas, que era uma mistura de modelagem de computador, intuição humana, e pequenas evidências sobre o que funcionava antes. "Rocklin disse." Os designers de proteínas eram como mestres artesãos que usavam sua experiência para esculpir à mão cada peça em sua oficina. Às vezes, as coisas funcionavam, mas quando eles falharam, era difícil dizer por quê. Nossa nova abordagem nos permite coletar uma enorme quantidade de dados sobre o que torna as proteínas estáveis. Esses dados agora podem orientar o processo de design. "