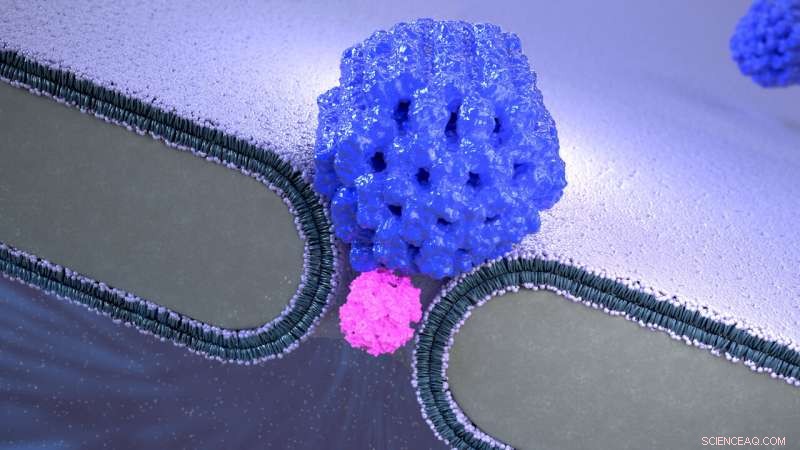
 p Impressão artística da armadilha eletro-osmótica Nanopore (NEOtrap) - um novo método para analisar moléculas individuais. Uma nanobola de origami de DNA cobre um nanofuro sólido, permitindo que ele capture moléculas de proteína individuais não modificadas, permitindo análises sensíveis à conformação de uma hora. Crédito:Cees Dekker Lab / SciXel
p Impressão artística da armadilha eletro-osmótica Nanopore (NEOtrap) - um novo método para analisar moléculas individuais. Uma nanobola de origami de DNA cobre um nanofuro sólido, permitindo que ele capture moléculas de proteína individuais não modificadas, permitindo análises sensíveis à conformação de uma hora. Crédito:Cees Dekker Lab / SciXel
p Pesquisadores das universidades técnicas de Delft e Munique inventaram um novo tipo de armadilha molecular que pode reter uma única proteína por horas para estudar seu comportamento natural - um milhão de vezes mais do que antes. A nova técnica NEOtrap permite aos cientistas usar correntes elétricas para estudar a natureza vibrante das proteínas, que pode desencadear inovações em biomedicina, biotecnologia, e mais. p Mesmo que as proteínas sejam cruciais para a vida, fornecendo a você visão e conexões neurais para ler este texto, por exemplo - a forma como mudam de forma ainda é mal compreendida. Conforme publicado na segunda-feira, 30 de agosto, em
Nature Nanotechnology , uma equipe liderada por Cees Dekker na Delft University of Technology desenvolveu uma nova técnica, chamada de armadilha eletro-osmótica Nanopore (NEOtrap), para estudar moléculas de proteína individuais por muito mais tempo do que era possível antes. O NEOtrap permite aos pesquisadores medir como proteínas individuais mudam de forma ao longo do tempo.
p
Como uma rolha em uma garrafa
p O NEOtrap combina duas nanotecnologias:nanoporos de estado sólido e origami de DNA. Nanoporos são pequenos orifícios que os cientistas usam como sensores para moléculas únicas, como proteínas. Uma vez que as proteínas normalmente passam pelo pequeno orifício em microssegundos, eles podem ser registrados apenas brevemente. Ao lacrar o nanofuro com uma bola em nanoescala construída inteiramente de DNA (!), os pesquisadores podem bloquear a proteína no local por horas, muito parecido com uma rolha fecha uma garrafa de vinho. Hendrik Dietz e seu grupo na Universidade Técnica de Munique construíram esta nano-bola usando uma abordagem chamada "origami de DNA" - uma técnica que imita a dobra de origami em macroescala - usando fios de DNA em nanoescala em vez de papel.
p Autora principal do artigo Sonja Schmid, que desenvolveu o NEOtrap como pós-doutorado no laboratório de Dekker, explica:"Esta nanobola de origami de DNA atua como uma esponja que suga água através do nanopore, atrair uma única proteína para o nanopore e prendê-la lá. Isso significa que podemos estudar essa proteína por longos períodos de tempo. Neste trabalho já demonstramos que podemos distinguir entre diferentes tipos de proteínas, e até mesmo diferentes formas funcionais de uma e da mesma proteína. "
p
Avanço radical no campo
p Cees Dekker acrescenta:"Esta nova técnica é realmente um grande passo à frente - um revisor anônimo de nosso artigo a chamou de" um dos avanços mais radicais no campo de detecção de nanoporos ". o NEOtrap nos permite capturar uma única proteína nativa sem a necessidade de modificar a molécula de interesse, ao contrário das técnicas anteriores. Esta técnica pode, por exemplo, ajudam os pesquisadores a descobrir o mecanismo subjacente de enzimas e outras proteínas importantes que mudam de forma para facilitar as reações químicas. "
p O NEOtrap permite que cientistas de todo o mundo realizem experimentos inteiramente novos, com o potencial de revelar características funcionais anteriormente negligenciadas de proteínas e, portanto, desencadear inovação na biomedicina, biotecnologia, e mais. Schmid (que agora começou seu próprio laboratório em Wageningen) e Dekker estão planejando muitos estudos de acompanhamento da dinâmica de proteínas individuais nos próximos anos.
