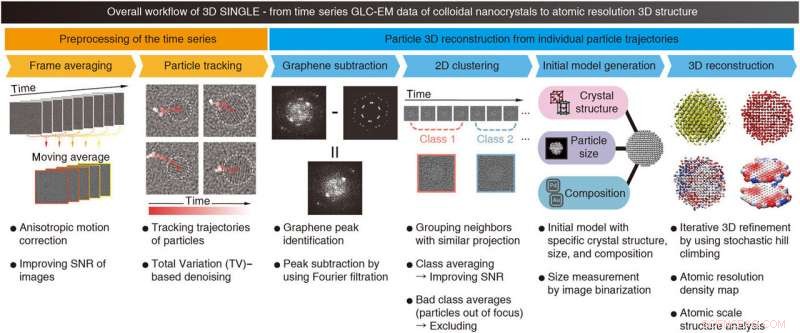
Fluxo de trabalho geral do SINGLE. O fluxo de trabalho geral e as descrições de cada etapa de SINGLE são mostrados. SINGLE consiste em duas etapas principais:Pré-processamento da série temporal (laranja), incluindo (i) cálculo da média de janela de tempo com correção de movimento anisotrópico e (ii) trajetória de partículas de rastreamento com o uso de redução de ruído à base de variação total (TV), e reconstrução 3D de partículas a partir de trajetórias de partículas individuais (azul), incluindo (i) identificação e subtração de fundo de grafeno, (ii) agrupamento 2D com restrição de tempo, com exclusão de imagens fora de foco, (iii) geração inicial do modelo, e (iv) reconstrução 3D e análise de estrutura em escala atômica. Crédito:Science Advances, doi:10.1126 / sciadv.abe6679
Os cientistas de materiais normalmente usam microscopia eletrônica de transmissão de fase de solução (TEM) para revelar as propriedades físico-químicas únicas das estruturas tridimensionais (3-D) dos nanocristais. Em um novo relatório sobre Avanços da Ciência , Cyril F. Reboul e uma equipe de pesquisa da Monash University, Austrália, Universidade Nacional de Seul, Coreia do Sul, e o Laboratório Nacional Lawrence Berkeley, nos Estados Unidos, desenvolveu um método de reconstrução 3-D browniano de partícula única. Para conseguir isso, eles obtiveram imagens de conjuntos de nanocristais coloidais usando microscopia eletrônica de transmissão de células líquidas de grafeno. A equipe obteve imagens de projeção de nanocristais girados de forma diferente usando um detector de elétrons direto para obter um conjunto de reconstruções 3-D. Nesse trabalho, eles introduziram métodos computacionais para reconstruir com sucesso nanocristais 3-D em resolução atômica e conseguiram isso rastreando partículas individuais ao longo do tempo, ao subtrair o fundo interferente. O método também pode identificar / rejeitar imagens de baixa qualidade para facilitar estratégias personalizadas para o alinhamento 2-D / 3-D que diferem daquelas na microscopia crioeletrônica biológica. A equipe disponibilizou os desenvolvimentos por meio de um pacote de software de código aberto conhecido como SINGLE. O software gratuito está disponível no GitHub.
Usando SINGLE para Cristalografia
Os pesquisadores sustentaram avanços na cristalografia nos últimos 50 anos para transformar a compreensão existente da química e da biologia. No entanto, alguns alvos, incluindo nanocristais solubilizados, permanecem intratáveis para os métodos cristalográficos tradicionais. Por exemplo, nanocristais coloidais contêm dezenas a centenas de átomos e mantêm uma variedade de aplicações em campos multidisciplinares, incluindo eletrônica, catálise e sensores biológicos. As versatilidades surgem da alta sensibilidade das propriedades nanocristais ao tamanho, composição química e outras variáveis durante a síntese. Tipicamente, cientistas usam uma única partícula, Reconstrução 3-D em biologia estrutural para determinar a estrutura das proteínas. A técnica é relativamente nova para reconstrução 3-D in-situ de nanocristais individuais solubilizados. Nesse trabalho, Reboul et al. desenvolvido SINGLE; um método que se baseou na reconstrução 3-D independente de nanocristais individuais solubilizados, incluindo o movimento browniano. A técnica é um avanço pioneiro no estudo para resolver estruturas atômicas 3-D de nanocristais diretamente da fase de solução.
Rastreamento de trajetórias nanocristais individuais. Acompanhando os resultados da partícula 1 (A e B) e 2 (C e D) ao longo do filme (azul para vermelho). Médias de tempo representativas de imagens de partículas não alinhadas brutas (50 quadros) são mostradas (B e D). Barras de escala, 1 nm. Crédito:Science Advances, doi:10.1126 / sciadv.abe6679 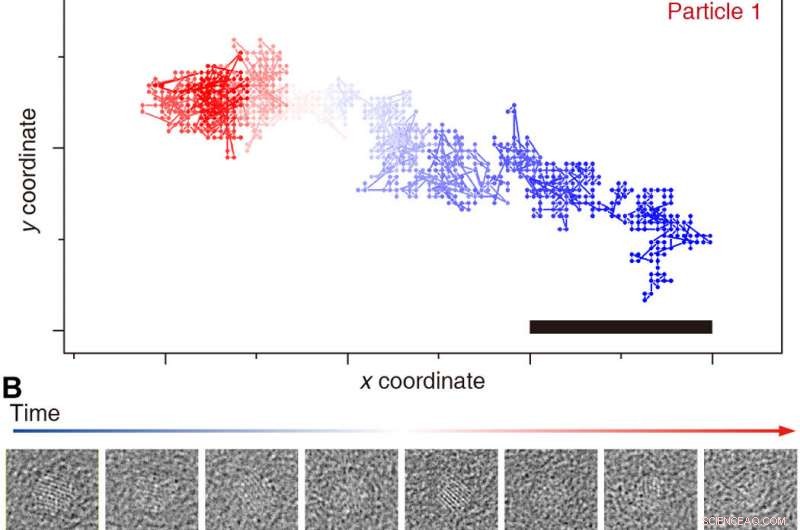
Os cientistas introduziram novos métodos de pré-processamento para melhorar a relação sinal-ruído (SNR) para rastrear as trajetórias das partículas ao remover o sinal de fundo induzido pelo grafeno. Métodos computacionais avançados podem reconstruir com sucesso em 3-D os dados de microscopia eletrônica de transmissão de célula líquida de grafeno (GLC) in-situ. Em comparação com as técnicas existentes, o trabalho apresentou a aplicabilidade de um método computacional inédito para a obtenção de reconstruções 3-D em resolução atômica para nanocristais dispersos em solução. Eles dividiram o fluxo de trabalho ÚNICO em duas etapas principais (1) pré-processamento e (2) reconstrução 3-D das partículas. Os cientistas tiveram como objetivo fornecer o melhor desempenho e eficiência possíveis em qualquer hardware de CPU, incluindo supercomputadores para estações de trabalho ou até mesmo laptops.
Inicialmente, a equipe calculou a média da janela de tempo em vários quadros com movimento anisotrópico para melhorar a relação sinal-ruído, levando a partículas visíveis e um sinal de grafeno aprimorado. A equipe então identificou as posições das partículas manualmente na média da primeira janela de tempo. Depois disso, a equipe desenvolveu um modelo inicial com base na estrutura cristalográfica esperada, diâmetro de partícula e elementos constituintes e reconstruções 3-D produzidas com coordenadas atômicas ajustadas para análise de estrutura em escala atômica.
Clustering 2D com restrição de tempo. (A) Fração de mudança angular ao longo da série temporal. A linha tracejada vermelha é a linha de tendência. As direções de projeção estão mudando rapidamente nas regiões entre os quadros 1500 a 1600 (laranja), quadros 3800 a 3900 (verde), e quadros 5600 a 5700 (azul). As inserções são plotagens que representam a diferença angular na direção da projeção nessas regiões. (B) Representação esquemática da média das classes 2D com restrição de tempo. (C) Gráfico mostrando classes alocadas para quadros individuais na região de 1 a 400. A inserção é o gráfico que mostra as classes alocadas em todos os quadros. (D) Médias de classe obtidas com agrupamento e alinhamento 2D com restrição de tempo. Crédito:Science Advances, doi:10.1126 / sciadv.abe6679 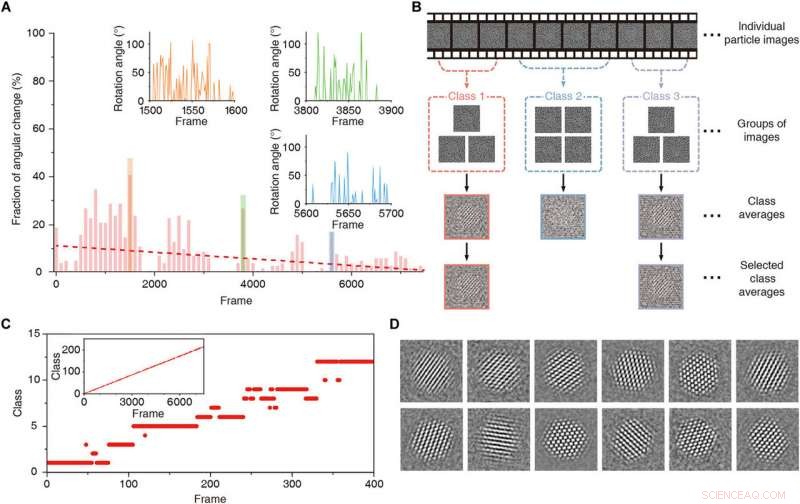
Reboul et al. introduziu um novo método de rastreamento usando transformadas rápidas de Fourier e a correlação de fase para identificar um máximo de correlação com a precisão de subpixel. A equipe reduziu a ruído da janela de tempo de extração usando redução de ruído à base de variação total (TV) e redução de ruído combinada e média de tempo para fornecer um método robusto para rastrear o movimento de nanocristais individuais em toda a amostra. O método permitiu que eles discernissem a forma geral dos nanocristais e / ou suas características cristalinas - o que atestou a robustez do algoritmo de rastreamento. Usando o método, eles também recuperaram trajetórias anteriormente desafiadoras para obter reconstruções 3-D e empregaram uma trajetória de partícula subtraída de fundo em todas as etapas de processamento de imagem para subtração de grafeno do GLC (célula líquida de grafeno). A equipe ainda caracterizou a natureza das rotações de nanocristais no espaço altamente confinado da célula líquida de grafeno. O método não era trivial devido à natureza probabilística do algoritmo de reconstrução 3-D. A equipe, portanto, incorporou uma abordagem determinística para melhorar a precisão do cluster, enquanto melhora a relação sinal-ruído em relação aos quadros individuais.
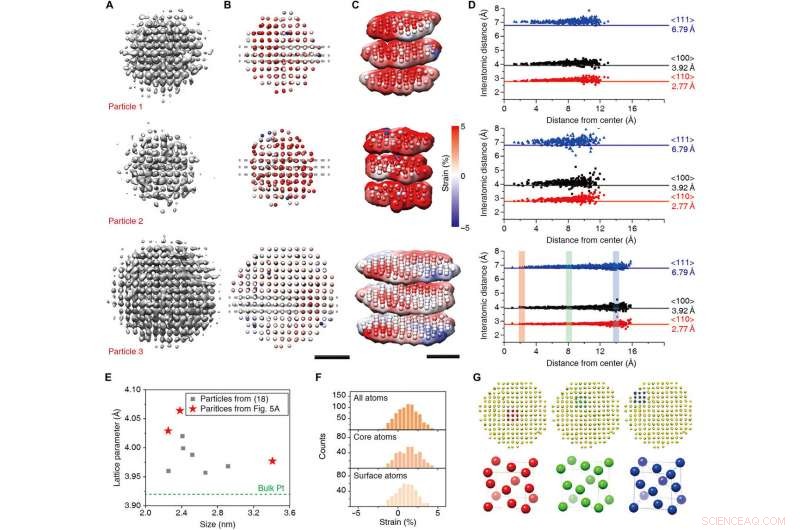
Resultados da reconstrução 3D e análise da estrutura em nível atômico. (A a C) Mapas de densidade 3D (A), mapas de deformação radial de coordenadas atômicas ajustadas (B), e sua representação de fatia (C). (D) Distâncias interatômicas nas direções de <110> (vermelho), <100> (Preto), e <111> (azul) para três nanocristais. (E) Parâmetros de rede ajustados de nanocristais relatados anteriormente (18) (quadrados cinza) e novos resultados (estrelas vermelhas). (F) Histograma de deformação radial de todos os átomos (topo), átomos centrais (meio), átomos de superfície (parte inferior) da partícula 3. (G) Estrutura da célula unitária do núcleo (vermelho), meio (verde), e a superfície (azul) da partícula 3. Barras de escala, 1 nm. Crédito:Science Advances, doi:10.1126 / sciadv.abe6679
Gerando modelos
Os pesquisadores desenvolveram um modelo inicial usando o conhecimento de que as partículas têm um arranjo de posição atômica aproximadamente cúbica. Eles simularam as densidades atômicas usando fatores de espalhamento atômico 5-Gaussiano. As projeções 2-D da densidade 3-D simulada representaram o caráter das projeções no núcleo do nanocristal, para superar problemas relacionados à simetria translacional e um sinal de fundo interferente. O método de refinamento 3-D em uso para microscopia crioeletrônica biológica não poderia ser diretamente aplicado a dados de séries temporais de nanocristais; Portanto, Reboul et al. introduziu modificações críticas. Eles usaram um esquema de refinamento de dois estágios para estabelecer a forma correta do nanocristal para permitir que os átomos e suas formas conduzam o alinhamento 3-D. Os pesquisadores escolheram três nanocristais de tamanhos variados que não foram reconstruídos anteriormente para benchmarking, em seguida, usando mapas atômicos produzidos com o método, Reboul et al. obteve detalhes estruturais microscópicos no nível atômico. O trabalho também facilitou mapas atômicos detalhando análise de deformação e análise de estrutura de célula unitária.
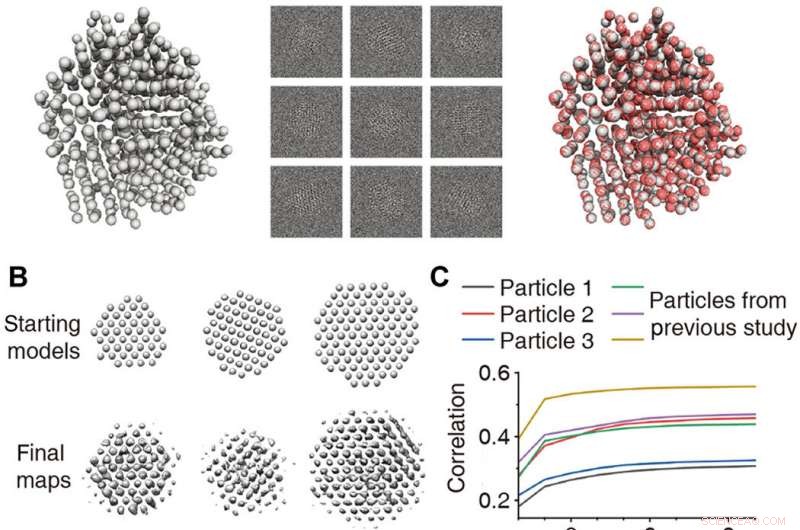
Validação de reconstruções 3D. (A) reconstrução 3D de partícula desordenada simulada com estrutura atômica conhecida. Esquerda:Modelo de um nanocristal desordenado obtido por simulação de dinâmica molecular. Médio:5000 imagens simuladas multislice com ruído adicionado para dar um SNR =0,1. À direita:Mapa atômico (vermelho) como resultado da reconstrução 3D sobreposto com o modelo de realidade terrestre (cinza). (B) Modelos 3D iniciais e mapas de densidade 3D finais obtidos a partir de dados experimentais. (C) Correlação entre as reprojeções do mapa de densidade 3D refinado e as visualizações de partículas experimentais plotadas como uma função de iteração para o primeiro estágio de refinamento 3D. Partículas apresentadas neste artigo (preto, vermelho, e azul) e apresentado em um estudo anterior (18) (verde, roxa, e ocre) são plotados. (D) Comparação das médias das turmas (indicadas como projeção) com reprojeções para validação das três estruturas. (E) Representação atômica dependente do tempo das direções de projeção para as três estruturas:branco (início) a rosa (meio) a vermelho (final). Vermelho, amarelo, e as setas azuis indicam x, y, e eixos z, respectivamente. Crédito:Science Advances, doi:10.1126 / sciadv.abe6679
Validando as reconstruções 3-D
Os pesquisadores ainda geraram um modelo de um nanocristal desordenado usando simulações de dinâmica molecular para entender a aplicabilidade do SINGLE em nanocristais altamente desordenados. Usando simulações multi-slice, eles aplicaram movimento translacional e variações aleatórias de desfocagem para representar o movimento realista das partículas. Eles então obtiveram um mapa de densidade 3-D do nanocristal desordenado de 500 imagens simuladas com uma relação sinal-ruído de 0,1 e um modelo inicial com ordem cristalina perfeita para concordar perfeitamente com as partículas originais. A equipe obteve a distribuição das direções de projeção dos nanocristais rotativos para validar a qualidade da reconstrução 3-D e exigirá mais estudos para entender como as estruturas atômicas reais dos nanocristais afetam a dinâmica rotacional.
Desta maneira, Cyril F. Reboul e colegas demonstraram métodos computacionais em SINGLE para obter mapas de densidade nanocristal com resolução atômica. Usando uma configuração de célula líquida avançada, como células líquidas de grafeno com nanocâmaras ordenadas, a equipe permitiu o controle da espessura do líquido para estender a aplicabilidade do SINGLE para aquisição de dados eficiente. O pacote SINGLE forneceu a primeira plataforma analítica eficiente no estudo para entender a origem estrutural das propriedades físicas e químicas exclusivas dos nanocristais em sua fase de solução nativa.
© 2021 Science X Network