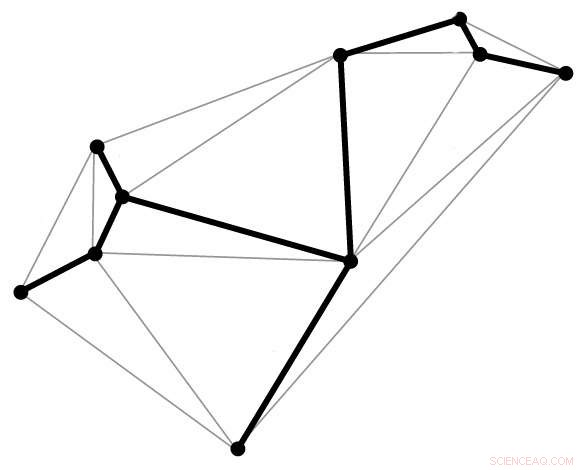
 p A linha em negrito, conhecido como uma árvore de abrangência, segue a forma geométrica desejada, tocando cada vértice apenas uma vez. Um algoritmo de árvore geradora é usado no novo método de origami de DNA para mapear o caminho de roteamento adequado para a fita de DNA. Crédito:Domínio Público
p A linha em negrito, conhecido como uma árvore de abrangência, segue a forma geométrica desejada, tocando cada vértice apenas uma vez. Um algoritmo de árvore geradora é usado no novo método de origami de DNA para mapear o caminho de roteamento adequado para a fita de DNA. Crédito:Domínio Público
p Entre os valiosos acervos da Biblioteca Wellcome de Londres, está um esboço a lápis feito em 1953 por Francis Crick. O desenho é um dos primeiros a mostrar a estrutura de dupla hélice do DNA - o projeto da Natureza para o design de caracóis marinhos, seres humanos, e todas as outras formas de vida na terra. p Poucos poderiam ter previsto, no entanto, as propriedades simples de auto-montagem do DNA, e sua capacidade versátil de transporte de informações, poderia ter muitos usos nunca imaginados por Watson e Crick, (ou de fato, pela própria Natureza).
p Em uma nova pesquisa que aparece na edição online avançada da revista
Ciência , Mark Bathe, junto com os colegas Hao Yan da ASU e Wah Chiu do Baylor College of Medicine descrevem um novo método para projetar formas geométricas construídas a partir do DNA. Eles apresentam uma nova variante de uma técnica conhecida como origami de DNA, em que as propriedades de emparelhamento de base do DNA são exploradas para a construção de estruturas minúsculas em 2 e 3 dimensões.
p "Um desafio importante no campo da nanotecnologia de DNA é projetar quaisquer estruturas desejáveis de uma maneira de cima para baixo, sem muita contribuição humana sobre os detalhes das trajetórias de dobra da fita de DNA, "Yan diz.
p Seus colaboradores no MIT, liderado por Mark Bathe, desenvolveu um algoritmo de computador para projetar nanoestruturas de DNA inserindo apenas uma forma de destino. Eles desenvolveram uma plataforma de software que pode computar e produzir os filamentos de DNA necessários para formar arquiteturas de designer. A formação dessas estruturas foi sistematicamente caracterizada e confirmada experimentalmente nos três institutos. "Isso realmente mostra a ciência colaborativa interdisciplinar em todo o país, "Yan diz
p Yan dirige o Centro de Biodesign para Design Molecular e Biomimética na Arizona State University.
p
Outros mundos
p A equipe projeta estruturas úteis em uma escala surpreendentemente minuciosa. (Um nanômetro é um bilionésimo de um metro ou aproximadamente o tamanho de uma molécula de açúcar.) Técnicas de imagem especializadas, incluindo força atômica - e microscopia crioeletrônica são usados para visualizar as formas resultantes.
p A técnica simplificada descrita promete estender significativamente o uso de origami de DNA além da comunidade de especialistas e expandir a gama de possíveis aplicações em ciência biomolecular e nanotecnologia. Estes incluem o uso de nanopartículas para entrega de drogas e direcionamento de células, construção de robôs em nanoescala capazes de realizar diversas atividades na medicina e na indústria e o projeto de dispositivos ópticos sob medida.
p Uma das inovações mais interessantes no horizonte envolve o uso de DNA como um meio de armazenamento - um meio de retenção que ostenta milhões de anos. (Um único grama de DNA pode armazenar cerca de 700 terabytes de informações, uma quantidade equivalente a 14, 000 discos Blu-ray de 50 gigabytes. Avançar, essa memória de ácido nucleico pode potencialmente ser operada com uma fração da energia necessária para outras opções de armazenamento de informações.)
p
Mudança de forma
p O novo método de design, que pode produzir virtualmente qualquer forma poliédrica, depende de uma estratégia de cima para baixo, que começa com um esboço da forma desejada e retrocede em etapas para definir a sequência de DNA necessária que se dobrará adequadamente para formar o produto acabado.
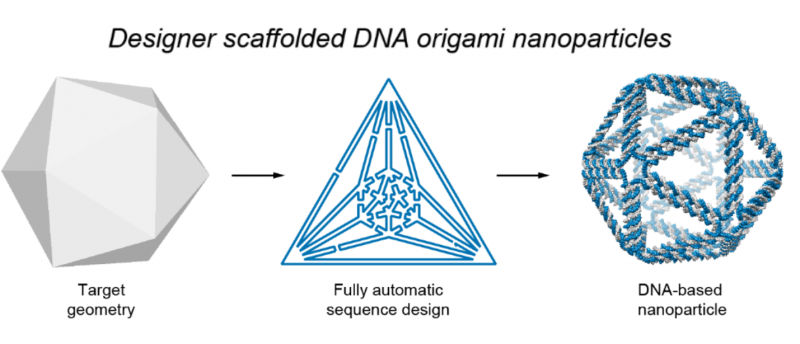 p A ilustração mostra o processo básico usado para projetar nanoestruturas de origami de DNA. Primeiro, um wireframe do design de destino pretendido é feito. O software então traduz isso em um plano para o encaminhamento de andaimes de DNA e fitas básicas, que se montam para formar a forma desejada. Crédito:Biodesign Institute
p A ilustração mostra o processo básico usado para projetar nanoestruturas de origami de DNA. Primeiro, um wireframe do design de destino pretendido é feito. O software então traduz isso em um plano para o encaminhamento de andaimes de DNA e fitas básicas, que se montam para formar a forma desejada. Crédito:Biodesign Institute
p O processo autônomo é realizado por meio de um programa de software desenvolvido pelos autores. Conhecido como DAEDALUS (para DNA Origami Sequence Design Algorithm para estruturas definidas pelo usuário), o programa realiza projeto inverso de nanoformas de origami de DNA arbitrárias, com base em uma malha de wireframe de entrada, (uma representação visual do fechado, Superfície geométrica tridimensional).
p O programa não é apenas amigável, mas altamente versátil, produzir formas não limitadas à topologia esférica, (ou seja, fechado, estruturas de dois lados sem limites ou orifícios). Uma vez que a forma alvo foi descrita como uma rede de nós e arestas, As fitas de andaime de DNA de comprimento e sequência customizados são geradas usando tecnologia conhecida como reação em cadeia da polimerase assimétrica.
p O novo estudo descreve a fabricação de uma variedade de objetos geométricos de DNA, incluindo 35 formas poliédricas (platônicas, Arquimedeano, Sólidos Johnson e Catalan) 6 estruturas assimétricas, e quatro poliedros com topologia não esférica, usando princípios de design inversos. O método pode produzir nanoformas com alta fidelidade e estabilidade sem o laborioso processo normal de projetar manualmente pares de bases para formar a estrutura alvo pretendida.
p
Entrando na dobra
p O origami de DNA traz o antigo método japonês de dobrar papel para a escala molecular. O básico é simples:pegue um pedaço de DNA de fita simples e guie-o na forma desejada, prender a estrutura em conjunto usando os chamados fios básicos mais curtos, que se ligam em locais estratégicos ao longo do comprimento mais longo do DNA. O método se baseia no fato de que as quatro letras de nucleotídeos do DNA - A, T, C, &G se unem de maneira consistente; Como sempre, emparelhar com Ts e Cs com Gs.
p A molécula de DNA em sua forma característica de fita dupla é bastante rígida, em comparação com o DNA de fita simples, que é flexível. Por esta razão, o DNA de fita simples é um material de andaime semelhante a uma renda. Avançar, suas propriedades de emparelhamento são previsíveis e consistentes, (ao contrário do RNA, que é considerado promíscuo, devido a pares de bases que podem ser inesperados).
p A técnica tem se mostrado extremamente bem-sucedida na criação de uma miríade de formas em 2 e 3 dimensões, que convenientemente se auto-montam quando as sequências de DNA projetadas são misturadas. A parte complicada é preparar a sequência de DNA adequada e o projeto de roteamento para o andaime e os filamentos básicos a fim de atingir a estrutura alvo desejada. Tipicamente, trata-se de um trabalho árduo que deve ser executado manualmente.
p Com a nova técnica, a estrutura alvo é descrita pela primeira vez em termos de uma malha de arame feita de poliedros. A partir disso, um algoritmo de spanning tree é gerado. Este é basicamente um mapa que guiará automaticamente o encaminhamento da fita de andaime de DNA por toda a estrutura do origami, tocando cada vértice na forma geométrica, uma vez. Fios complementares básicos são então atribuídos e a forma final se auto-monta.
p Para testar o método, formas mais simples conhecidas como sólidos platônicos foram fabricadas pela primeira vez, seguido por estruturas cada vez mais complexas. Isso incluía objetos com topologias não esféricas e detalhes internos incomuns, que nunca havia sido realizado experimentalmente antes.
p Os projetos concluídos demonstraram a capacidade da técnica de cima para baixo de gerar automaticamente andaimes e roteamentos básicos para uma ampla gama de nanoformas, baseado unicamente na geometria da superfície. Cryo-EM foi usado para confirmar a fidelidade estrutural e a estabilidade das estruturas de origami montadas.
p Outras experiências confirmaram que as estruturas de DNA produzidas eram potencialmente adequadas para aplicações biológicas, uma vez que exibiam estabilidade a longo prazo no soro e em condições de baixo teor de sal.
p A pesquisa abre caminho para o desenvolvimento de sistemas projetados em nanoescala que imitam as propriedades dos vírus, organismos fotossintéticos e outros produtos sofisticados da evolução natural.
p Além de sua nomeação no Instituto Biodesign, Hao Yan é o distinto professor Milton D. Glick, Faculdade de Artes e Ciências Liberais, Escola de Ciências Moleculares da ASU.

