Imagens inéditas podem ajudar no uso de DNA para construir dispositivos em nanoescala
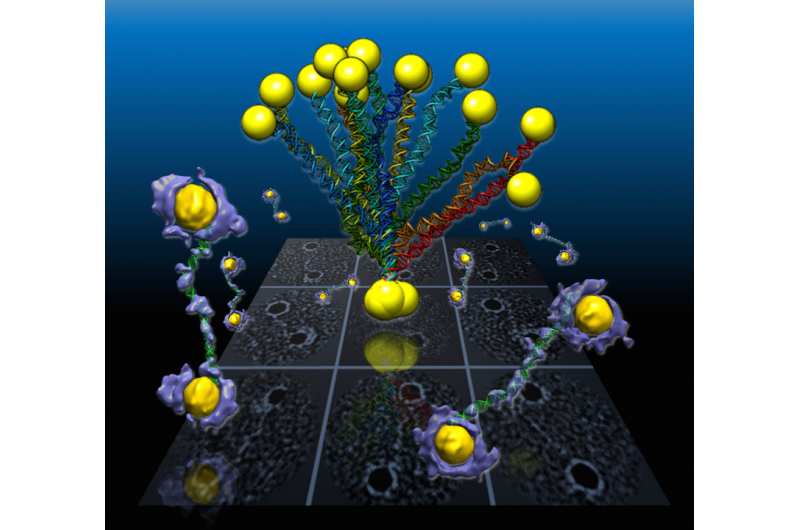 p Em um estudo conduzido pelo Berkeley Lab, segmentos flexíveis de dupla hélice de DNA conectados a nanopartículas de ouro são revelados a partir dos mapas de densidade 3-D (roxo e amarelo) reconstruídos a partir de amostras individuais usando uma técnica desenvolvida pelo Berkeley Lab chamada tomografia eletrônica de partícula individual ou IPET. As projeções das estruturas são mostradas na grade de fundo. Crédito:Berkeley Lab
p Em um estudo conduzido pelo Berkeley Lab, segmentos flexíveis de dupla hélice de DNA conectados a nanopartículas de ouro são revelados a partir dos mapas de densidade 3-D (roxo e amarelo) reconstruídos a partir de amostras individuais usando uma técnica desenvolvida pelo Berkeley Lab chamada tomografia eletrônica de partícula individual ou IPET. As projeções das estruturas são mostradas na grade de fundo. Crédito:Berkeley Lab
p Uma equipe internacional que trabalha no Laboratório Nacional Lawrence Berkeley do Departamento de Energia (Berkeley Lab) capturou as primeiras imagens 3-D de alta resolução de segmentos individuais de dupla hélice de DNA anexados em cada extremidade a nanopartículas de ouro. As imagens detalham a estrutura flexível dos segmentos de DNA, que aparecem como cordas de salto em nanoescala. p Esta capacidade de imagem única, pioneiro por cientistas do Berkeley Lab, poderia ajudar no uso de segmentos de DNA como blocos de construção para dispositivos moleculares que funcionam como sistemas de entrega de drogas em nanoescala, marcadores para pesquisa biológica, e componentes para memória de computador e dispositivos eletrônicos. Também pode levar a imagens de proteínas importantes para doenças relevantes que se mostraram ilusórias para outras técnicas de imagem, e do processo de montagem que forma o DNA separado, fios individuais.
p As formas das fitas enroladas de DNA, que foram imprensadas entre nanopartículas de ouro em forma de polígono, foram reconstruídos em 3-D usando uma técnica de microscópio eletrônico de ponta juntamente com um processo de coloração de proteína e software sofisticado que forneceu detalhes estruturais na escala de cerca de 2 nanômetros, ou dois bilionésimos de um metro.
p "Não tínhamos ideia sobre como seria o DNA de fita dupla entre as partículas nanogold, "disse o Gang" Gary "Ren, um cientista do Berkeley Lab que liderou a pesquisa. "Esta é a primeira vez para visualizar diretamente um segmento individual de DNA de fita dupla em 3-D, "disse. Os resultados foram publicados na edição de 30 de março da
Nature Communications .
 p Gang Ren (em pé) e Lei Zhang participaram de um estudo no Berkeley Lab's Molecular Foundry que produziu reproduções 3-D de amostras individuais de segmentos de DNA de dupla hélice anexados a nanopartículas de ouro. Crédito:Roy Kaltschmidt / Berkeley Lab
p Gang Ren (em pé) e Lei Zhang participaram de um estudo no Berkeley Lab's Molecular Foundry que produziu reproduções 3-D de amostras individuais de segmentos de DNA de dupla hélice anexados a nanopartículas de ouro. Crédito:Roy Kaltschmidt / Berkeley Lab
p O método desenvolvido por esta equipe, chamada de tomografia eletrônica de partículas individuais (IPET), já havia capturado a estrutura 3-D de uma única proteína que desempenha um papel fundamental no metabolismo do colesterol humano. Ao capturar imagens 2-D do mesmo objeto de diferentes ângulos, a técnica permite que os pesquisadores montem uma imagem 3-D desse objeto. A equipe também usou a técnica para descobrir a flutuação de outra proteína flexível bem conhecida, imunoglobulina 1 humana, que desempenha um papel no nosso sistema imunológico.
p Para este último estudo de nanoestruturas de DNA, Ren usou uma técnica de estudo de feixe de elétrons chamada microscopia crioeletrônica (cryo-EM) para examinar amostras de nanogold de DNA congeladas, e usou IPET para reconstruir imagens 3-D de amostras coradas com sais de metais pesados. A equipe também usou ferramentas de simulação molecular para testar as variações naturais da forma, chamado de "conformações, "nas amostras, e comparou essas formas simuladas com observações.
p Ren explicou que a dinâmica naturalmente flexível das amostras, como um homem agitando os braços, não pode ser totalmente detalhado por nenhum método que use uma média de muitas observações.
p Uma maneira popular de visualizar os detalhes estruturais em nanoescala de delicadas amostras biológicas é transformá-los em cristais e dispará-los com raios-X, embora isso não preserve sua forma natural e as amostras de nanogold de DNA neste estudo sejam incrivelmente desafiadoras para cristalizar. Outras técnicas de pesquisa comuns podem exigir uma coleção de milhares de objetos quase idênticos, visto com um microscópio eletrônico, para compilar um único, estrutura 3-D média. Mas esta imagem 3-D pode não mostrar adequadamente as flutuações da forma natural de um determinado objeto.
Este vídeo mostra técnicas que os cientistas usaram para produzir reconstruções 3-D de flutuações de forma em segmentos de DNA de dupla hélice anexados a nanopartículas de ouro. Crédito:Lei Zhang, Dongsheng Lei, Jessica M. Smith, Meng Zhang, Huimin Tong, Xing Zhang, Zhuoyang Lu, Jiankang Liu, A. Paul Alivisatos e Gang "Gary" Ren p As amostras no último experimento foram formadas a partir de nanoestruturas de ouro poligonal individuais, medindo cerca de 5 nanômetros de diâmetro, conectado a fitas de segmento único de DNA com 84 pares de bases. Os pares de bases são blocos de construção químicos básicos que dão ao DNA sua estrutura. Cada segmento de DNA individual e nanopartícula de ouro naturalmente compactados com um parceiro para formar o segmento de DNA de fita dupla com uma partícula de ouro em cada extremidade.
p As amostras foram congeladas rapidamente para preservar sua estrutura para estudo com imagem crio-EM, e a distância entre as duas partículas de ouro em amostras individuais variou de 20-30 nanômetros com base nas diferentes formas observadas nos segmentos de DNA. Os pesquisadores usaram um microscópio crioeletrônico na Fundição Molecular do Berkeley Lab para este estudo.
p Eles coletaram uma série de imagens inclinadas dos objetos manchados, e reconstruiu 14 mapas de densidade de elétrons que detalhavam a estrutura de amostras individuais usando a técnica IPET. Eles reuniram uma dúzia de conformações para as amostras e descobriram que as variações da forma do DNA eram consistentes com as medidas nas amostras crio-EM congeladas por flash. As formas também eram consistentes com as amostras estudadas usando outras imagens baseadas em elétrons e métodos de espalhamento de raios-X, e com simulações de computador.
p Enquanto as reconstruções 3-D mostram a estrutura básica em nanoescala das amostras, Ren disse que o próximo passo será trabalhar para melhorar a resolução para a escala sub-nanométrica.
p "Mesmo neste estado atual, começamos a ver estruturas 3-D com resolução de 1 a 2 nanômetros, "disse ele." Por meio de melhor instrumentação e algoritmos computacionais aprimorados, seria promissor empurrar a resolução para a visualização de uma única hélice de DNA dentro de uma proteína individual. "
p A tecnica, ele disse, já despertou o interesse de algumas empresas farmacêuticas proeminentes e pesquisadores de nanotecnologia, e sua equipe científica já tem dezenas de projetos de pesquisa relacionados em andamento.
p Em estudos futuros, pesquisadores poderiam tentar melhorar a resolução de imagem para estruturas complexas que incorporam mais segmentos de DNA como uma espécie de "origami de DNA, "Ren disse. Os pesquisadores esperam construir e melhor caracterizar dispositivos moleculares em nanoescala usando segmentos de DNA que podem, por exemplo, armazenar e distribuir medicamentos em áreas específicas do corpo.
p "O DNA é fácil de programar, sintetizar e replicar, portanto, pode ser usado como um material especial para se automontar rapidamente em nanoestruturas e para guiar a operação de dispositivos em escala molecular, ", disse ele." Nosso estudo atual é apenas uma prova de conceito para imagens desses tipos de estruturas de dispositivos moleculares. "

