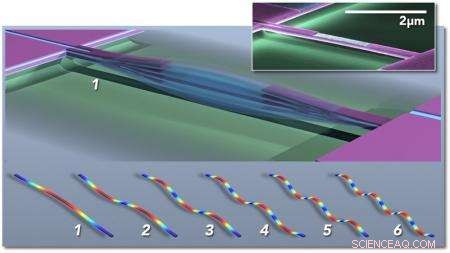
 p Sensor de massa baseado em sistemas nanoeletromecânicos multimodo (NEMS); a figura principal representa esquematicamente uma viga duplamente fixada vibrando no modo fundamental (1). "Instantâneos" conceituais dos primeiros seis modos vibracionais são mostrados abaixo (1-6), as cores indicam tensão alta (vermelha) a baixa (azul). A inserção mostra uma micrografia eletrônica colorida de um ressonador piezoelétrico NEMS fabricado no Instituto de Nanociência Kavli da Caltech. Crédito:M. Matheny, L.G. Villanueva, P. Hung, J. Li e M. Roukes / Caltech
p Sensor de massa baseado em sistemas nanoeletromecânicos multimodo (NEMS); a figura principal representa esquematicamente uma viga duplamente fixada vibrando no modo fundamental (1). "Instantâneos" conceituais dos primeiros seis modos vibracionais são mostrados abaixo (1-6), as cores indicam tensão alta (vermelha) a baixa (azul). A inserção mostra uma micrografia eletrônica colorida de um ressonador piezoelétrico NEMS fabricado no Instituto de Nanociência Kavli da Caltech. Crédito:M. Matheny, L.G. Villanueva, P. Hung, J. Li e M. Roukes / Caltech
p Com base na criação do primeiro dispositivo mecânico que pode medir a massa de moléculas individuais, um por vez, uma equipe de cientistas do Caltech e seus colegas criaram nanodispositivos que também podem revelar sua forma. Essas informações são cruciais ao tentar identificar grandes moléculas de proteínas ou conjuntos complexos de moléculas de proteínas. p "Você pode imaginar que, com grandes complexos de proteínas feitos de muitos diferentes, subunidades menores, há muitas maneiras de montá-las. Eles podem acabar tendo massas bastante semelhantes, embora sejam, na verdade, espécies diferentes com funções biológicas diferentes. Isso é especialmente verdadeiro com enzimas, proteínas que medeiam reações químicas no corpo, e proteínas de membrana que controlam as interações de uma célula com seu ambiente, "explica Michael Roukes, o professor de física Robert M. Abbey, Física aplicada, e Bioengineering at Caltech e o co-autor correspondente de um artigo descrevendo a tecnologia que apareceu em 30 de março na edição online da revista
Nature Nanotechnology .
p Um dos alicerces da revolução genômica tem sido a capacidade de replicar moléculas de DNA ou RNA em massa usando a reação em cadeia da polimerase para criar os muitos milhões de cópias necessárias para o sequenciamento e análise típicos. Contudo, a mesma tecnologia de produção em massa não funciona para copiar proteínas. Agora mesmo, se você quiser identificar adequadamente uma proteína em particular, você precisa muito dele - normalmente milhões de cópias apenas da proteína de interesse, com muito poucas outras proteínas estranhas como contaminantes. A massa média desta população molecular é então avaliada com uma técnica chamada espectrometria de massa, em que as moléculas são ionizadas - para que atinjam uma carga elétrica - e então podem interagir com um campo eletromagnético. Ao analisar essa interação, os cientistas podem deduzir a relação massa-carga molecular.
p Mas a espectrometria de massa muitas vezes não consegue discriminar diferenças sutis, mas cruciais, em moléculas com proporções de massa para carga semelhantes. "Com a espectrometria de massa hoje, "explica Roukes, "grandes moléculas e complexos moleculares são primeiro cortados em muitos pedaços menores, isso é, em fragmentos de moléculas menores que os instrumentos existentes podem manipular. Esses diferentes fragmentos são analisados separadamente, e então a bioinformática - envolvendo simulações de computador - é usada para montar o quebra-cabeça. Mas esse processo de remontagem pode ser frustrado se as peças de diferentes complexos forem misturadas. "
p Com seus dispositivos, Roukes e seus colegas podem medir a massa de uma molécula intacta individual. Cada dispositivo - que tem apenas alguns milionésimos de metro de tamanho ou menor - consiste em uma estrutura vibratória chamada ressonador de sistema nanoeletromecânico (NEMS). Quando uma partícula ou molécula pousa no nanodispositivo, a massa adicionada muda a frequência com que a estrutura vibra, da mesma forma que colocar gotas de solda em uma corda de violão mudaria a frequência de sua vibração e o tom resultante. As mudanças induzidas na frequência fornecem informações sobre a massa da partícula. Mas eles também, conforme descrito no novo artigo, pode ser usado para determinar a distribuição espacial tridimensional da massa:ou seja, a forma da partícula.
p "Uma corda de violão não vibra apenas em uma frequência, "Roukes diz." Existem harmônicos em seu tom fundamental, ou os chamados modos vibracionais. O que distingue uma corda de violino de uma corda de violão são, na verdade, as diferentes misturas desses diferentes harmônicos do tom fundamental. O mesmo se aplica aqui. Temos um monte de tons diferentes que podem ser ativados simultaneamente em cada um de nossos nanodispositivos, e rastreamos muitos tons diferentes em tempo real. Acontece que quando a molécula pousa em orientações diferentes, esses harmônicos são deslocados de forma diferente. Podemos então usar a teoria da imagem inercial que desenvolvemos para reconstruir uma imagem no espaço da forma da molécula. "
p "A nova técnica revela uma capacidade anteriormente não realizada de sensores mecânicos, "diz o professor Mehmet Selim Hanay, da Universidade Bilkent em Ancara, Turquia, um ex-pesquisador de pós-doutorado no laboratório de Roukes e co-autor do artigo. "Anteriormente, identificamos moléculas, como o anticorpo IgM, com base apenas em seus pesos moleculares. Agora, ao permitir que as informações de peso molecular e forma sejam deduzidas para a mesma molécula simultaneamente, a nova técnica pode melhorar muito o processo de identificação, e isso é importante tanto para a pesquisa básica quanto para a indústria farmacêutica. "
p Atualmente, estruturas moleculares são decifradas usando cristalografia de raios-X, uma técnica frequentemente trabalhosa que envolve isolar, purificando, e então moléculas de cristalização, e então avaliando sua forma com base nos padrões de difração produzidos quando os raios X interagem com os átomos que juntos formam os cristais. Contudo, muitas moléculas biológicas complexas são difíceis, senão impossíveis, de cristalizar. E, mesmo quando podem ser cristalizados, a estrutura molecular obtida representa a molécula no estado cristalino, que pode ser muito diferente da estrutura da molécula em sua forma biologicamente ativa.
p "Você pode imaginar situações em que não sabe exatamente o que está procurando - em que está no modo de descoberta, e você está tentando descobrir a resposta imunológica do corpo a um patógeno específico, por exemplo, "Roukes diz. Nesses casos, a capacidade de realizar a detecção de uma única molécula e de obter o máximo possível de bits separados de informação sobre aquela molécula individual aumenta muito as chances de fazer uma identificação única.
p "Dizemos que o câncer começa muitas vezes com uma única célula aberrante, e o que isso significa é que, embora possa ser uma de uma multiplicidade de células semelhantes, há algo único na composição molecular daquela célula. Com esta técnica, temos potencialmente uma nova ferramenta para descobrir o que é único nisso, " ele adiciona.
p Até aqui, a nova técnica foi validada usando partículas de tamanhos e formas conhecidas, como nanogotículas de polímero. Roukes e colegas mostram que, com os nanodispositivos modernos de hoje, a abordagem pode fornecer resolução em escala molecular, ou seja, fornecem a capacidade de ver os subcomponentes moleculares do indivíduo, conjuntos de proteínas intactas. Os esforços atuais do grupo agora estão focados em tais explorações.
p Scott Kelber, um ex-aluno de pós-graduação no laboratório de Roukes, é o outro co-primeiro autor do artigo, intitulado "Imagem inercial com sistemas nanoeletromecânicos". Professor John Sader da Universidade de Melbourne, Austrália, e um associado visitante em física na Caltech, é o autor co-correspondente. Outros co-autores são Cathal D. O'Connell e Paul Mulvaney, da University of Melbourne. O trabalho foi financiado por um prêmio Pioneer do National Institutes of Health Director, um distinto professor visitante do Caltech Kavli Nanoscience Institute, a Fundação para la Recherche et l'Enseignement Superieur em Paris, e o esquema de bolsas do Australian Research Council.
