
Uma captura de tela do site DIVE. Crédito:Gupta et al.
Os artigos acadêmicos geralmente contêm relatos de novos avanços e teorias interessantes relacionadas a uma variedade de campos. Contudo, a maioria desses artigos é escrita usando jargão e linguagem técnica que só podem ser entendidos por leitores familiarizados com aquela área específica de estudo.
Leitores não especialistas são, portanto, normalmente incapazes de compreender artigos científicos, a menos que sejam selecionados e tornados mais acessíveis por terceiros que entendam os conceitos e ideias contidos neles. Com isso em mente, uma equipe de pesquisadores do Texas Advanced Computing Center da University of Texas at Austin (TACC), A Oregon State University (OSU) e a American Society of Plant Biologists (ASPB) se propuseram a desenvolver uma ferramenta que pode extrair automaticamente frases importantes e terminologia de documentos de pesquisa para fornecer definições úteis e melhorar sua legibilidade.
“Nosso projeto é motivado pela necessidade de melhorar a legibilidade dos artigos de periódicos, "Weijia Xu, que lideram a equipe da TACC, disse TechXplore. “É um esforço conjunto de curadores biológicos, editores de periódicos e cientistas da computação com o objetivo de desenvolver um serviço da Web que possa reconhecer e permitir a curadoria do autor de terminologia importante usada em publicações de periódicos. A terminologia e as palavras são então anexadas ao final do artigo da revista, a fim de aumentar sua acessibilidade para os leitores. "
Xu e seus colegas desenvolveram uma estrutura extensível que pode ser usada para extrair informações de documentos. Eles então implementaram essa estrutura dentro de um serviço da web chamado DIVE (Domain Information Vocabulary Extraction), integrando-o com o pipeline de publicação de periódicos do ASPB. Ao contrário das ferramentas existentes para extrair informações de domínio, sua estrutura combina várias abordagens, incluindo extração guiada por ontologia, extração baseada em regras, processamento de linguagem natural (PNL) e técnicas de aprendizagem profunda.

A visão geral da arquitetura do sistema proposta pelos pesquisadores. Crédito:Gupta et al.
“Os resultados obtidos por diferentes modelos são armazenados em um banco de dados centralizado, "Xu explicou." Nós também projetamos um serviço da web que permite aos usuários selecionar os resultados da extração. O serviço da web é integrado com o pipeline de publicação de produção na ASPB. "
Uma vez que a versão prévia de um artigo de jornal é enviada e entra no pipeline do ASPB, o manuscrito é alimentado automaticamente para DIVE, que o processa e produz uma URL com a qual o autor poderá acessar os resultados do processamento de DIVE. O autor do artigo é convidado a visitar o link fornecido e revisar as informações extraídas antes de poder enviar o artigo oficialmente.
“O autor precisa visitar o site do DIVE para revisar os resultados da extração e fazer a aprovação final da lista de informações a serem incluídas no final de seu artigo, "Disse Xu." O DIVE também rastreia as correções do autor para melhorar as tarefas de extração futuras. Atualmente, nenhum outro editor de periódicos adotou uma abordagem semelhante e a integrou ao seu pipeline de publicações. "
Durante suas análises e ao extrair dados importantes de documentos, o framework desenvolvido pelos pesquisadores utiliza diversas técnicas. Isso permite que ele capture mais informações do que outros métodos, como ABNER (A Biomedical Named Entity Recognizer), que é uma ferramenta de software de código aberto para mineração de texto de biologia molecular que só pode extrair termos gerais (por exemplo, genes e proteínas). Ao contrário de DIVE, ABNER é baseado apenas em campos aleatórios condicionais (CRFs), um método de modelagem estatística comumente usado em aplicativos de reconhecimento de padrões e aprendizado de máquina.
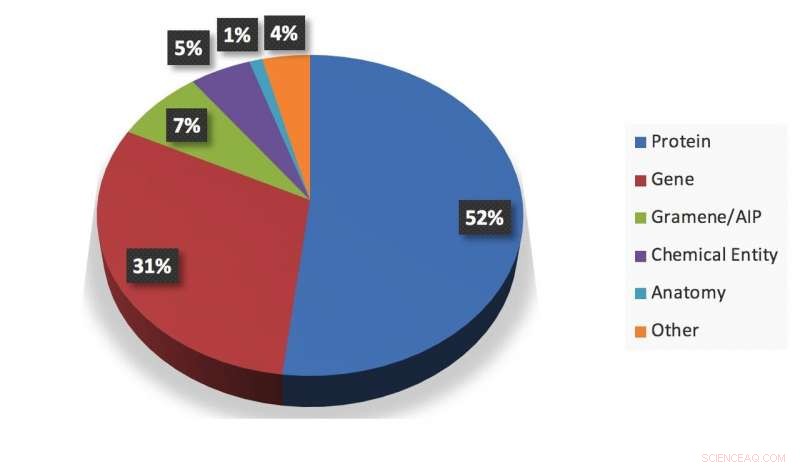
Um resumo visual de um instantâneo das informações extraídas pelo sistema. Crédito:Gupta et al.
"Uma grande contribuição do nosso projeto é que ele ajuda a construir conjuntos de dados e modelos que podem inferir os interesses de pesquisa dos autores a partir de suas publicações, "Xu disse." Nosso projeto pode beneficiar comunidades mais amplas de pesquisadores biológicos. Para autores, as extrações e inclusão das principais informações podem aumentar a acessibilidade de seus artigos. "
Xu e seu colega Amit Gupta avaliaram sua estrutura e compararam seu desempenho com o de outras ferramentas de extração de informações, incluindo ABNER. Suas descobertas revelaram que, usando várias abordagens, incluindo aprendizagem profunda, O DIVE atinge pontuações de maior precisão do que outros modelos pré-treinados baseados exclusivamente em CRFs. Interessantemente, a estrutura DIVE também pode ser atualizada continuamente, já que modelos de extração adicionais podem ser adicionados a ele a qualquer momento.
O aplicativo da web DIVE não permite apenas que leitores não especialistas entendam melhor os trabalhos acadêmicos, também pode ajudá-los a identificar trabalhos alinhados aos seus interesses. Pesquisadores, por outro lado, pode usar o DIVE para se manter informado sobre áreas de pesquisa específicas, bem como para aprender sobre novas terminologias e tendências relacionadas ao seu campo de interesse. Finalmente, as informações geradas pelo aplicativo também podem orientar curadores de biologia em suas decisões e processos de coleta de dados.
"Continuamos nosso projeto explorando duas direções, "Xu disse." Por um lado, estamos investigando novos métodos para incorporar aos nossos modelos de extração de informações para melhorar o desempenho. Por outro lado, também estamos tentando expandir nosso serviço, oferecendo-o a outras comunidades de usuários e editores de periódicos. "
© 2019 Science X Network