Simulações computadorizadas de proteínas ajudam a desvendar por que ocorre a resistência à quimioterapia
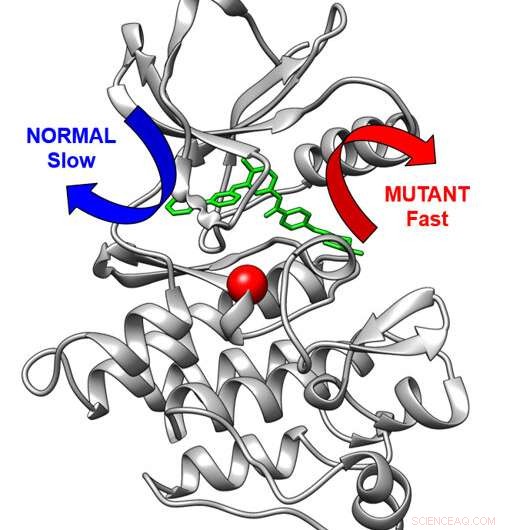
Estrutura tridimensional do alvo da droga contra o câncer Abl quinase (cinza) ligada à droga anticâncer imatinib. Normalmente, a droga sai lentamente pela seta azul. Uma modificação na quinase (esfera vermelha) faz com que a droga saia por uma via rápida (seta vermelha). Crédito:Aziz M. Rangwala
Compreender por que e como a resistência à quimioterapia ocorre é um passo importante para otimizar os tratamentos para o câncer. Uma equipe de cientistas, incluindo Markus Seeliger, Ph.D., do Stony Brook Cancer Center e da Renaissance School of Medicine da Stony Brook University, acredita ter encontrado um novo processo pelo qual a resistência às drogas acontece. Eles estão usando um modelo de simulação de computador que os ajuda a entender exatamente como as moléculas interagem com o medicamento contra o câncer Imatinib (conhecido como Glivec) no processo de resistência à quimioterapia. O imatinib trata a leucemia mieloide crônica (LMC) de forma altamente eficaz, mas muitos pacientes em estágio avançado apresentam resistência ao medicamento, o que torna o medicamento minimamente eficaz nesse estágio.
A pesquisa é destacada em um artigo publicado em
Angewandte Chemie e se baseia em pesquisas anteriores detalhadas em 2021 no
PNAS .
O imatinib inibe a proteína quinase BCR-Abl, uma maquinaria de sinalização celular excessivamente ativa na LMC. No
PNAS No estudo, os pesquisadores mostraram que variações no plano de construção da quinase podem dificultar a ligação do Imatinib à quinase e também acelerar a liberação da droga da quinase. Na
Angewandte Chemie No artigo, a equipe de pesquisa usou a metodologia computacional - desenvolvida pelo co-autor Pratyush Tiwary, da Universidade de Maryland - que lhes permitiu estudar a liberação muito lenta de Imatinib da quinase.
"Este método em si é uma grande conquista técnica que amplia as habilidades computacionais para a pesquisa de resistência a medicamentos e nos levou a ser capaz de prever a rapidez com que proteínas saudáveis e mutantes liberariam esse medicamento", diz Seeliger, Professor Associado do Departamento de Farmacologia Ciências. "Pela primeira vez, pudemos ver a liberação de uma droga de uma proteína com tanto detalhe e precisão. Além disso, pudemos mostrar que a mutação muda fundamentalmente dentro da rota de saída da droga da proteína.
"Isso é importante, pois a velocidade de liberação do medicamento pode ser tão importante para o efeito terapêutico de um medicamento quanto a força com que um medicamento se liga à proteína".
Seeliger explica ainda que o método pode fornecer uma base para a compreensão dos mecanismos moleculares por trás da resistência à quimioterapia.
Mais amplamente, as implicações do que eles descobriram são que, se os cientistas puderem entender como os medicamentos são liberados de suas proteínas, eles poderão projetar medicamentos com liberação mais lenta e maior impacto terapêutico. Além disso, se a liberação rápida do medicamento puder causar resistência ao medicamento, e os médicos puderem mostrar que isso está acontecendo, eles poderão reativar a eficácia do medicamento solicitando ao paciente que tome o medicamento com mais frequência.
A base para o teste de mutação através do método computacional foi delineada no
PNAS papel. Seeliger e colegas testaram como o imatinib se liga a mutações em pacientes com LMC resistente ao imatinib. Eles descobriram que a maioria das mutações se liga prontamente ao imatinib, de modo que colocou a questão de como essas mutações causam resistência nos pacientes? Os pesquisadores então identificaram vários mutantes que se ligaram prontamente ao imatinib, mas liberaram a droga muito mais rapidamente.
Depois de identificar esses mutantes com uma liberação mais rápida da droga, a equipe usou ressonância magnética nuclear (RMN) e dinâmica molecular para vincular a proteína à dissociação da droga – subjacente à importância da cinética de dissociação da droga para a eficácia da droga. Isso permitiu que eles identificassem um novo mecanismo de resistência ao imatinibe.
O trabalho que resultou no artigo publicado no
PNAS envolveu os esforços colaborativos de Seeliger e seus colegas em Stony Brook, e pesquisadores do Memorial Sloan Kettering Cancer Center e da Goethe University of Frankfurt, Alemanha.
A pesquisa que resultou no artigo mais recente foi liderada por Tiwary e colegas da Universidade de Maryland, em colaboração com Seeliger e cientistas do Broad Institute do MIT e da Universidade de Harvard.
+ Explorar mais Nova simulação de computador desvenda o mistério da resistência aos medicamentos contra o câncer
