Pesquisadores investigam mudanças estruturais em proteínas congeladas rapidamente
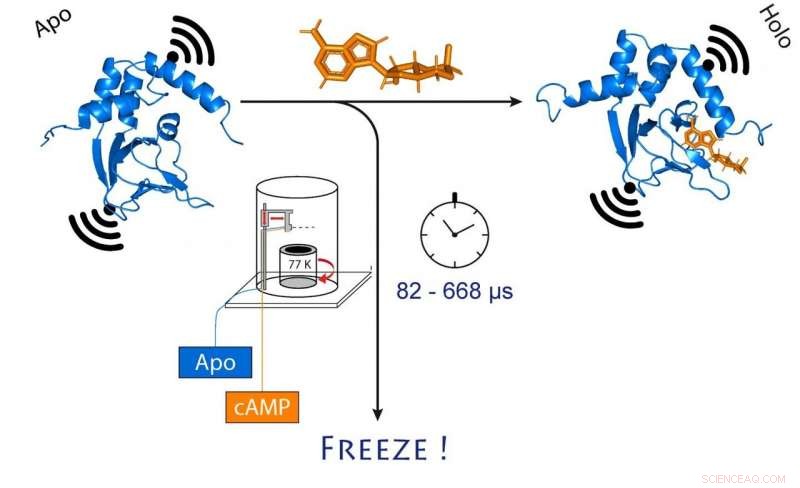 p quando o cAMP (laranja) se liga. Isso também altera a distância entre os dois ímãs moleculares presos ao canal (preto). Crédito:(c) Tobias Hett / Universität Bonn und Forschungszentrum caesar
p quando o cAMP (laranja) se liga. Isso também altera a distância entre os dois ímãs moleculares presos ao canal (preto). Crédito:(c) Tobias Hett / Universität Bonn und Forschungszentrum caesar
p Pesquisadores da Universidade de Bonn e do Centro de Pesquisa caesar conseguiram congelar proteínas ultrarrápidas após um período de tempo precisamente definido. Eles foram capazes de acompanhar as mudanças estruturais na escala de tempo de microssegundos e com precisão sub-nanométrica. Devido à sua alta resolução espacial e temporal, o método permite rastrear mudanças estruturais rápidas em enzimas e ácidos nucléicos. Os resultados são publicados no
Journal of the American Chemical Society. p Se você quiser saber como é a estrutura espacial de uma biomolécula, você tem um formidável arsenal de ferramentas à sua disposição. Os mais populares são microscopia eletrônica e difração de raios-X, que pode revelar até os menores detalhes de uma proteína. Contudo, uma limitação significativa desses métodos é que eles geralmente fornecem imagens estáticas, que muitas vezes são insuficientes para entender os processos biomoleculares em termos mecanísticos precisos. Portanto, um objetivo de longo prazo de muitos grupos de pesquisa em todo o mundo tem sido rastrear os movimentos dentro de uma macromolécula, como uma proteína, ao longo do tempo enquanto ela realiza seu trabalho, exatamente como em um filme. Os grupos de pesquisa liderados pelo Prof. Dr. Olav Schiemann do Instituto de Química Física e Teórica da Universidade de Bonn e o Prof. Dr. Benjamin Kaupp do Centro de Pesquisa caesar da Max Planck Society estão agora um passo mais perto de alcançar isso meta.
p Eles escolheram um canal iônico para sua investigação. Esta é uma proteína que forma poros minúsculos na membrana celular que são permeáveis a partículas carregadas chamadas íons. "Este canal está normalmente fechado, "Schiemann explica." Ele só abre quando um mensageiro celular, chamado cAMP, se liga a ele. Queríamos saber exatamente como esse processo funciona. "
p
Mini ímãs para medir distâncias
p Para fazer isso, os pesquisadores primeiro misturaram a proteína do canal e o cAMP e, em seguida, congelaram rapidamente a solução. No estado congelado, a estrutura da proteína agora pode ser analisada. Para que seu método funcione, eles fixaram eletroímãs moleculares em dois pontos do canal. A distância entre esses ímãs pode ser determinada com uma precisão de alguns Angstrom (dez bilionésimos de milímetro) usando um método sofisticado chamado PELDOR, que funciona como uma régua molecular. Nos últimos anos, o método foi significativamente refinado e melhorado no grupo de Schiemann.
p "Contudo, isso apenas nos dá uma imagem estática da ligação do cAMP ao canal iônico, "Schiemann diz." Portanto, repetimos o processo de congelamento em momentos diferentes depois de misturar as duas moléculas. Isso permitiu reconstruir os movimentos na proteína após a ligação do cAMP - assim como um filme, que também é composta por uma sequência de imagens. "
p No centro deste procedimento está um método sofisticado que permite que as amostras sejam misturadas e congeladas muito rapidamente em um momento preciso. A tecnica, chamado de "microssegundo congelamento hiperquenching" (abreviado MHQ), foi originalmente desenvolvido na Delft University, mas depois caiu em desuso. Foi redescoberto e definitivamente refinado pelo grupo de Kaupp.
p "No dispositivo MHQ, a molécula de cAMP e o canal de íons são misturados em velocidade ultrarrápida, "Kaupp explica." Em seguida, a mistura é injetada como um fio de cabelo em um cilindro de metal muito frio a -190 ° C, que gira 7, 000 vezes por minuto. Foi particularmente desafiador transferir as amostras congeladas para a medição PELDOR da placa de metal para tubos de vidro finos, e mantê-los congelados enquanto isso. Tivemos que projetar e construir ferramentas especiais para isso. "
p
Congelamento em 82 milionésimos de segundo
p Todo o processo de mistura e congelamento leva apenas 82 microssegundos (um microssegundo é igual a um milionésimo de segundo). "Isso nos permite visualizar mudanças muito rápidas na estrutura espacial das proteínas, "explica Tobias Hett, um dos dois alunos de doutorado que contribuíram significativamente para o sucesso. A vantagem do método é a combinação de alta resolução espacial e temporal. "Isso representa um grande passo no estudo de processos dinâmicos em biomoléculas, "Kaupp enfatiza.
p Os pesquisadores agora planejam usar seu método para examinar mais de perto outras biomoléculas. Eles esperam obter novos insights, por exemplo, no funcionamento de enzimas e ácidos nucleicos. A importância de tais percepções é mais bem ilustrada pelo recente surto mundial de pesquisas estruturais sobre o coronavírus-2 da SARS:a chamada proteína de pico do vírus também sofre uma mudança estrutural quando as células humanas são infectadas. O esclarecimento desse mecanismo fornecerá informações valiosas sobre como direcionar o mecanismo de infecção com novos medicamentos.
p A preparação das amostras, a execução experimental, e a análise dos dados é muito complexa. Os resultados do estudo, portanto, também refletem uma cooperação científica bem-sucedida com pesquisadores liderados pelo Prof. Dr. Helmut Grubmüller do Instituto Max Planck de Química Biofísica em Göttingen e o Prof. Dr. Heinz-Jürgen Steinhoff da Universidade de Osnabrück.
