Fotocageamento enzimático para o estudo da regulação gênica por meio da metilação do DNA
 p Crédito:Wiley
p Crédito:Wiley
p A adição e remoção de grupos metil no DNA desempenha um papel importante na regulação do gene. Para estudar esses mecanismos com mais precisão, uma equipe alemã desenvolveu um novo método pelo qual locais específicos de metilação podem ser bloqueados e, em seguida, desbloqueados em um momento preciso por meio de irradiação com luz (fotocageamento). Conforme relatado no jornal
Angewandte Chemie , o regente necessário é produzido enzimaticamente, no local. p Embora pareçam muito diferentes e tenham funções completamente diferentes, todas as células do nosso corpo têm DNA idêntico. Contudo, eles não usam os mesmos genes. Certos genes estão ativados e outros desativados, dependendo do tipo de célula e do momento. Os "interruptores" são mudanças químicas nos blocos de construção do DNA. Essas mudanças são chamadas de modificações epigenéticas. Um mecanismo de regulação significativo é a metilação e desmetilação, significando a ligação e remoção de um grupo metil (-CH
3 ) Os padrões de metilação das células cancerosas, por exemplo, diferem das células saudáveis. Durante uma metilação, enzimas conhecidas como metil transferases (MTases) transferem um grupo metil de S-adenosil-
eu -metionina (AdoMet) para a molécula alvo.
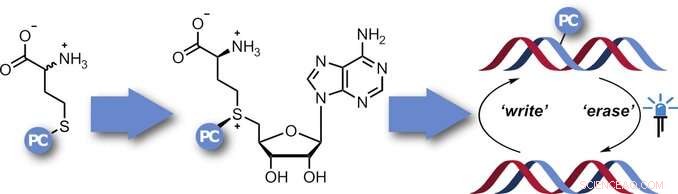
p A fim de estudar a finalidade e a função desta regulação mais de perto e determinar os padrões de metilação, seria útil ter "ferramentas" para inibir especificamente a metilação em locais-alvo e, em seguida, levantar a inibição em um momento definido. Para este fim, uma equipe liderada por Andrea Rentmeister optou por usar um método conhecido como fotocaging. Neste método, um "fotocage" é uma molécula que se desfaz com a irradiação, tal como um grupo 2-nitrobenzil. A gaiola primeiro bloqueia o local alvo, então, a irradiação direcionada com luz atua como um 'interruptor' para remover o bloqueio.
p A ideia era equipar os análogos AdoMet com uma fotocagem que é então transferida para os locais de metilação. Contudo, Os análogos de AdoMet se decompõem em soluções aquosas e não podem entrar nas células. Portanto, a equipe da Universidade de Münster queria produzi-los no local. No corpo, O AdoMet é produzido a partir do aminoácido metionina por meio da ação da enzima, metionina adenosil transferase (MAT). A síntese dos análogos AdoMet requer metionina com uma fotocagem de nitrobenzil anexada e um MAT que pode usar tal substrato alterado. Começando com uma enzima MAT de um organismo unicelular (Cryptosporidium hominis), os pesquisadores foram capazes de alterar cuidadosamente os aminoácidos específicos na enzima para aumentar o tamanho de sua cavidade de ligação hidrofóbica para que pudesse conter o grupo nitrobenzil. Uma análise da estrutura cristalina mostrou que o análogo ADoMet está ligado à cavidade deste MAT de fotocadeamento (PC-MAT). Com base nessas informações, a equipe também produziu um segundo PC-MAT baseado em uma enzima MAT termoestável do archaeon Methanocaldococcus jannaschii.
p Ambos os PC-MATs são compatíveis com DNA e RNA MTases e tornaram possível anexar fotocágulos a todos os sítios naturais de metilação de um DNA plasmídeo. A irradiação com luz removeu o bloqueio.
