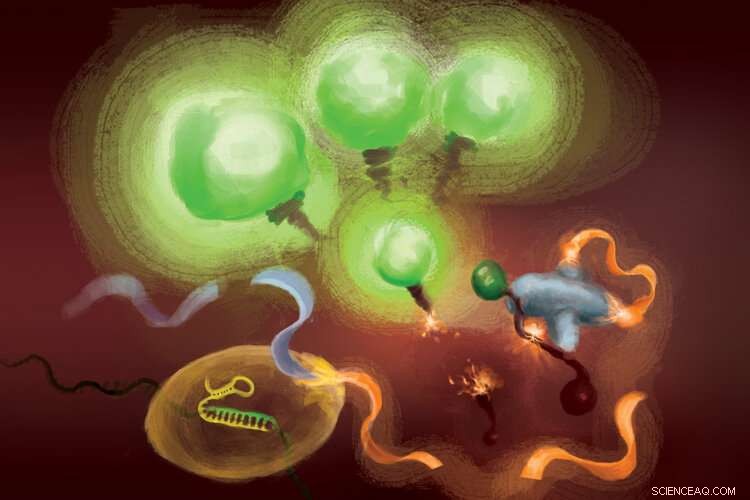
As reações envolvidas no ensaio FIND-IT para detectar a infecção com o vírus SARS-CoV-2. Quando a enzima Cas13 (canto inferior esquerdo) se liga ao seu RNA alvo, ele corta uma molécula (fita laranja e azul claro) para liberar um ativador (laranja) que sobrecarrega a nuclease Csm6 (direita) para clivar e liberar moléculas fluorescentes que acendem (verde) e sinalizam a presença de RNA viral. Crédito:Margaret L. Liu, Escola de Medicina Pritzker da Universidade de Chicago.
Freqüente, o teste rápido para COVID-19 é fundamental para controlar a propagação de surtos, especialmente como novo, mais variantes transmissíveis surgem.
Embora o teste de diagnóstico padrão ouro COVID-19 de hoje, que usa qRT-PCR - reação em cadeia da polimerase reversa quantitativa (PCR) - é extremamente sensível, detectando até uma cópia de RNA por microlitro, requer equipamento especializado, um tempo de execução de várias horas e uma instalação de laboratório centralizada. Como resultado, o teste normalmente leva pelo menos um a dois dias.
Uma equipe de pesquisa liderada por cientistas nos laboratórios de Jennifer Doudna, David Savage e Patrick Hsu da Universidade da Califórnia, Berkeley, tem como objetivo desenvolver um teste de diagnóstico muito mais rápido e fácil de implantar do que o qRT-PCR. Agora ele combinou dois tipos diferentes de enzimas CRISPR para criar um ensaio que pode detectar pequenas quantidades de RNA viral em menos de uma hora. Doudna compartilhou o Prêmio Nobel de Química de 2020 pela invenção da edição do genoma CRISPR-Cas9.
Embora a nova técnica ainda não esteja no estágio em que rivaliza com a sensibilidade do qRT-PCR, que pode detectar apenas algumas cópias do vírus por microlitro de líquido, já é capaz de captar níveis de RNA viral - cerca de 30 cópias por microlitro - suficientes para serem usados para vigiar a população e limitar a propagação de infecções.
"Você não precisa da sensibilidade do PCR para basicamente capturar e diagnosticar COVID-19 na comunidade, se o teste for conveniente e rápido o suficiente, "disse o co-autor David Savage, professor de biologia molecular e celular. "Nossa esperança era conduzir a bioquímica o mais longe possível ao ponto onde você pudesse imaginar um formato muito conveniente em um ambiente onde você pode fazer o teste todos os dias, dizer, na entrada do trabalho. "
Os pesquisadores reportarão seus resultados online no dia 5 de agosto na revista Nature Chemical Biology .
Vários ensaios baseados em CRISPR foram autorizados para uso de emergência pela Food and Drug Administration, mas todos requerem uma etapa inicial em que o RNA viral é amplificado para que o sinal de detecção - que envolve a liberação de uma molécula fluorescente que brilha sob luz azul - seja brilhante o suficiente para ser visto. Embora esta amplificação inicial aumente a sensibilidade do teste para um nível semelhante ao qRT-PCR, também apresenta etapas que tornam o teste mais difícil de ser realizado fora do laboratório.
A equipe liderada pela UC Berkeley procurou alcançar uma sensibilidade e velocidade úteis sem sacrificar a simplicidade do ensaio.
"Para aplicações em pontos de atendimento, você deseja ter uma resposta rápida para que as pessoas possam saber rapidamente se estão infectadas ou não, antes de embarcar em um vôo, por exemplo, ou visite parentes, "disse a líder da equipe Tina Liu, um cientista pesquisador no laboratório de Doudna no Innovative Genomics Institute (IGI), um centro focado no CRISPR envolvendo cientistas da UC Berkeley e da UC San Francisco.
Além de ter uma etapa adicional, outra desvantagem da amplificação inicial é que, porque faz bilhões de cópias de RNA viral, há uma maior chance de contaminação cruzada nas amostras de pacientes. A nova técnica desenvolvida pela equipe inverte isso e, em vez disso, aumenta o sinal fluorescente, eliminando uma importante fonte de contaminação cruzada.
A técnica livre de amplificação, que eles denominam Detecção de nuclease integrada rápida em tandem (FIND-IT), poderia permitir testes de diagnóstico rápidos e baratos para muitas outras doenças infecciosas.
"Embora tenhamos iniciado este projeto com o propósito expresso de impactar o COVID-19, Acho que esta técnica específica pode ser aplicável a mais do que apenas esta pandemia porque, em última análise, CRISPR é programável, "Liu disse." Então, você pode carregar a enzima CRISPR com uma sequência direcionada ao vírus da gripe ou HIV ou qualquer tipo de vírus de RNA, e o sistema tem potencial para funcionar da mesma maneira. Este artigo realmente estabelece que esta bioquímica é uma maneira mais simples de detectar RNA e tem a capacidade de detectar esse RNA em um período de tempo sensível e rápido que pode ser passível de futuras aplicações em diagnósticos de ponto de atendimento. "
Os pesquisadores estão atualmente no processo de construção de tal diagnóstico usando FIND-IT, que incluiria etapas para coletar e processar amostras e executar o ensaio em um dispositivo microfluídico compacto.
Empregando proteínas Cas em tandem
Para remover a amplificação do alvo da equação, a equipe empregou uma enzima CRISPR - Cas13 - para primeiro detectar o RNA viral, e outro tipo de proteína Cas, chamado Csm6, para amplificar o sinal de fluorescência.
Cas13 é uma tesoura de uso geral para cortar RNA; uma vez que se liga à sua sequência alvo, especificado por um RNA guia, ele é preparado para cortar uma ampla gama de outras moléculas de RNA. Esta atividade de corte desencadeada por alvo pode ser aproveitada para acoplar a detecção de uma sequência de RNA específica para a liberação de uma molécula repórter fluorescente. Contudo, sozinho, Cas13 pode levar horas para gerar um sinal detectável quando quantidades muito baixas de RNA alvo estão presentes.
O insight de Liu foi usar Csm6 para amplificar o efeito de Cas13. Csm6 é uma enzima CRISPR que detecta a presença de pequenos anéis de RNA e se torna ativada para cortar uma ampla gama de moléculas de RNA nas células.
Para aumentar a detecção de Cas13, ela e seus colegas projetaram uma molécula ativadora especialmente projetada que é cortada quando o Cas13 detecta o RNA viral. Um fragmento desta molécula pode se ligar e acionar o Csm6 para cortar e liberar uma molécula fluorescente brilhante de um pedaço de RNA. Normalmente, a molécula ativadora é rapidamente quebrada por Csm6, limitando assim a quantidade de sinal fluorescente que pode gerar. Liu e seus colegas desenvolveram uma maneira de modificar quimicamente o ativador para que ele seja protegido da degradação e possa sobrecarregar o Csm6 para cortar e liberar repetidamente moléculas fluorescentes ligadas ao RNA. Isso resulta em uma sensibilidade 100 vezes melhor do que o ativador original.
"Quando Cas13 é ativado, ele cliva este pequeno ativador, removendo um segmento que o protege, "Liu disse." Agora que está liberado, pode ativar muitas moléculas diferentes dessa segunda enzima, Csm6. E entao, um alvo reconhecido pelo Cas13 não leva apenas à ativação de sua própria capacidade de corte de RNA; leva à geração de muito mais enzimas ativas que podem, cada uma, clivar ainda mais repórteres fluorescentes. "
A equipe de pesquisadores também incorporou uma combinação otimizada de RNAs guia que permite um reconhecimento mais sensível do RNA viral por Cas13. Quando isso foi combinado com Csm6 e seu ativador, a equipe foi capaz de detectar até 31 cópias por microlitro de RNA SARS-CoV-2 em apenas 20 minutos.
Os pesquisadores também adicionaram RNA extraído de amostras de pacientes ao ensaio FIND-IT em um cartucho microfluídico, para ver se este ensaio pode ser adaptado para ser executado em um dispositivo portátil. Usando um pequeno dispositivo com uma câmera, eles poderiam detectar o RNA SARS-CoV-2 extraído de amostras de pacientes com uma sensibilidade que capturaria infecções por COVID-19 em seu pico.
"Esta abordagem de nuclease em tandem - Cas13 mais Csm6 - combina tudo em uma única reação a uma única temperatura, 37 graus Celsius, portanto, não requer aquecimento de alta temperatura ou várias etapas, como é necessário para outras técnicas de diagnóstico, "Liu disse." Eu acho que isso abre oportunidades para mais rapidez, testes mais simples que podem atingir uma sensibilidade comparável a outras técnicas atuais e podem potencialmente atingir sensibilidades ainda maiores no futuro. "
O desenvolvimento deste método livre de amplificação para detecção de RNA resultou de uma reorientação da pesquisa dentro do IGI quando a pandemia começou em direção a problemas de diagnóstico e tratamento de COVID-19. Em última análise, cinco laboratórios na UC Berkeley e dois laboratórios na UCSF se envolveram neste projeto de pesquisa, um de muitos dentro do IGI.
"Quando começamos isso, tínhamos esperança de criar algo que atingisse a paridade com o PCR, mas não exigia amplificação - esse seria o sonho, "disse Savage, que foi o investigador principal do projeto. "E de uma perspectiva de sensibilidade, tínhamos uma lacuna de cerca de dez mil vezes para pular. Fizemos cerca de mil vezes; baixamos cerca de três ordens de magnitude. Então, estamos quase lá. Abril passado, quando estávamos realmente começando a mapear, isso parecia quase impossível. "