Evidência estrutural de um metalocofator dinâmico durante a redução do dinitrogênio pela Mo-nitrogenase
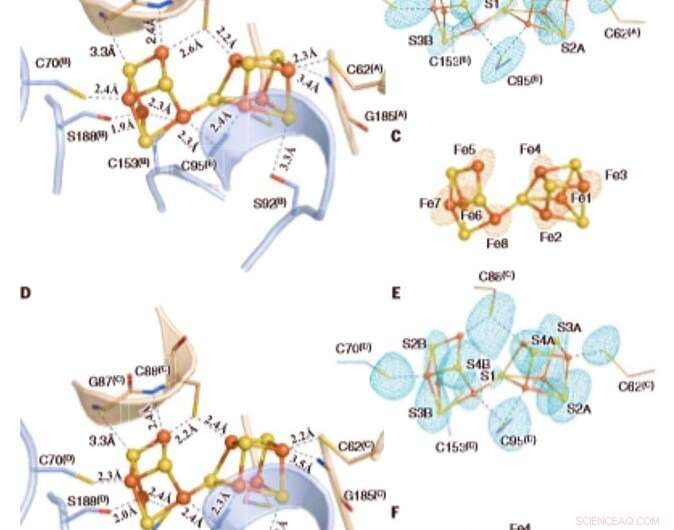 p Os aglomerados P oxidados em Av1 *. Estruturas de P-clusters em (A a C) a interface das cadeias A e B [P-cluster (A / B)] e (D a F) a interface das cadeias C e D [P-cluster (C / D) ] de Av1 *. As cadeias A e C são as subunidades a, e as cadeias B e D são as subunidades b dos dois dímeros ab de Av1 *. [(A) e (D)] Os P-clusters são mostrados na apresentação ball-and-stick, e os resíduos-chave que interagem com os aglomerados P são indicados como bastões. As cadeias A e C são mostradas como fitas de trigo, e as cadeias B e D são mostradas como fitas azuis claras. [(B) e (C)] Grupo P (A / B) e [(E) e (F)] Grupo P (C / D) sobreposto com [(B) e (E)] os mapas de densidade anômala calculado em 7100 eV em uma resolução de 2,18 Å e contornado em 4,0s, mostrando a posição dos átomos de enxofre (malha azul-menta); e com [(C) e (F)] os mapas de densidade anômala calculados em 7141 eV em uma resolução de 2,1 Å e contornados em 15,0s, mostrando a posição dos átomos de ferro (malha vermelha). Os átomos são coloridos da seguinte forma:Fe, laranja; S, amarelo; O, vermelho; N, azul. As abreviações de uma única letra para os resíduos de aminoácidos são as seguintes:C, Cys; G, Gly; H, Seu; R, Arg; S, Ser. Crédito:Ciência, doi:10.1126 / science.aaz6748
p Os aglomerados P oxidados em Av1 *. Estruturas de P-clusters em (A a C) a interface das cadeias A e B [P-cluster (A / B)] e (D a F) a interface das cadeias C e D [P-cluster (C / D) ] de Av1 *. As cadeias A e C são as subunidades a, e as cadeias B e D são as subunidades b dos dois dímeros ab de Av1 *. [(A) e (D)] Os P-clusters são mostrados na apresentação ball-and-stick, e os resíduos-chave que interagem com os aglomerados P são indicados como bastões. As cadeias A e C são mostradas como fitas de trigo, e as cadeias B e D são mostradas como fitas azuis claras. [(B) e (C)] Grupo P (A / B) e [(E) e (F)] Grupo P (C / D) sobreposto com [(B) e (E)] os mapas de densidade anômala calculado em 7100 eV em uma resolução de 2,18 Å e contornado em 4,0s, mostrando a posição dos átomos de enxofre (malha azul-menta); e com [(C) e (F)] os mapas de densidade anômala calculados em 7141 eV em uma resolução de 2,1 Å e contornados em 15,0s, mostrando a posição dos átomos de ferro (malha vermelha). Os átomos são coloridos da seguinte forma:Fe, laranja; S, amarelo; O, vermelho; N, azul. As abreviações de uma única letra para os resíduos de aminoácidos são as seguintes:C, Cys; G, Gly; H, Seu; R, Arg; S, Ser. Crédito:Ciência, doi:10.1126 / science.aaz6748
p A enzima nitrogenase é um catalisador biológico que pode reduzir o dinitrogênio (N2) a amônia na presença de um conjunto de metalocofatores complexos. Contudo, os detalhes mecanicistas da reação permanecem escassos. Em um novo relatório sobre
Ciência , Wonchull Kang e uma equipe de pesquisa em química, biologia molecular e bioquímica da University of California-Irvine, NÓS., relataram uma estrutura de cristal de 1,83 angstrom para a proteína nitrogenase molibdênio-ferro (MoFe), que eles capturaram sob condições fisiológicas de renovação de dinitrogênio. Os resultados do estudo podem avaliar os possíveis mecanismos de N
2 redução e o papel dos locais de enxofre da correia durante o processo. p A nitrogenase é um catalisador para uma etapa crítica no ciclo global do nitrogênio, durante a redução ambiental do dinitrogênio atmosférico (N
2 ) para a amônia biodisponível (NH
3 ) A enzima nitrogenase de molibdênio contém dois componentes de proteína:um contendo a proteína de ferro (Fe) em um enxofre de ferro (Fe
4 S
4 )
agrupar com um local de ligação de trifosfato de adenosina (ATP) dentro de cada subunidade. O outro, proteína de ferro molibdênio (MoFe) contendo um α
2 β
2 heterotetrâmero com dois metaloclusters complexos. Durante a catálise de molibdênio-nitrogenase (Mo-nitrogenase), a associação e dissociação repetida entre os dois componentes da proteína permitiu a transferência de elétrons dependente de ATP do Fe
4 S
4 cluster à proteína MoFe para redução do substrato. A capacidade da nitrogenase de transportar muitos elétrons para seu cofator tornou a enzima altamente versátil durante a redução do substrato.
p
Compreendendo o mecanismo de ação da enzima nitrogenase
p Muitos esforços foram feitos para entender os mecanismos da nitrogenase desde sua descoberta, onde alguns se concentraram nas interações do substrato e do inibidor da enzima. Desses esforços, Kang et al. determinou uma estratégia digna de consideração, limitando o excesso de suprimentos de elétrons que inadvertidamente conduziu o N
2 processo de redução para a frente. Isso reverteu o substrato ou estado de ligação intermediária da enzima a um estado de repouso ou reduziu a enzima a um estado misto indiscernível. O processo foi relevante uma vez que as proteínas nitrogenase são rotineiramente isoladas na presença de excesso de ditionita como um redutor fornecido externamente, e a remoção desta fonte artificial de elétrons na ausência de oxigênio poderia ajudar os cientistas a capturar o dinitrogênio (N
2 ) ou seus intermediários para análise.
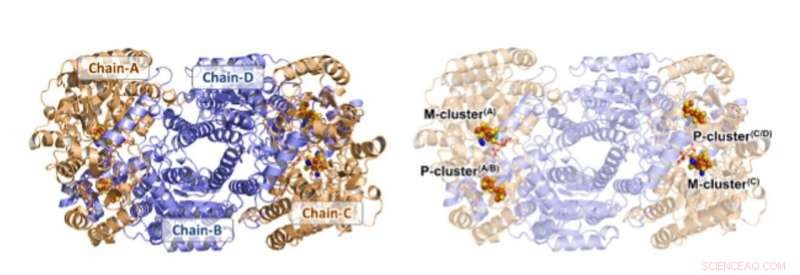 p Estrutura de Av1 *. Apresentações de fita sólidas (esquerda) e transparentes (direita) do heterotetrâmero de Av1 *, com as subunidades a (Cadeia-A e Cadeia-C) e subunidades b (Cadeia-B e Cadeia-D) coloridas em trigo e azul, respectivamente. Os clusters M e P são ilustrados como modelos de preenchimento de espaço. Código de cores dos átomos:Fe, laranja; S, amarelo; O, vermelho; N, azul; Mo, ciano; C, cinzento. Os M-clusters na Cadeia-A e Cadeia-C são designados M-cluster (A) e M-cluster (C), respectivamente. Os P-clusters nas interfaces Chain-A / Chain-B e Chain-C / Chain-D são designados P-cluster (A / B) e P-cluster (C / D), respectivamente. PYMOL foi usado para preparar esta figura. Crédito:Ciência, doi:10.1126 / science.aaz6748
p Estrutura de Av1 *. Apresentações de fita sólidas (esquerda) e transparentes (direita) do heterotetrâmero de Av1 *, com as subunidades a (Cadeia-A e Cadeia-C) e subunidades b (Cadeia-B e Cadeia-D) coloridas em trigo e azul, respectivamente. Os clusters M e P são ilustrados como modelos de preenchimento de espaço. Código de cores dos átomos:Fe, laranja; S, amarelo; O, vermelho; N, azul; Mo, ciano; C, cinzento. Os M-clusters na Cadeia-A e Cadeia-C são designados M-cluster (A) e M-cluster (C), respectivamente. Os P-clusters nas interfaces Chain-A / Chain-B e Chain-C / Chain-D são designados P-cluster (A / B) e P-cluster (C / D), respectivamente. PYMOL foi usado para preparar esta figura. Crédito:Ciência, doi:10.1126 / science.aaz6748
p Como prova de conceito, Kang et al. preparou o extrato bruto de uma cepa bacteriana aneróbia Azotobacter vinelandii com ou sem a adição de ditionito após o rompimento celular. A cepa A. vinelandii expressou ativamente uma Mo-nitrogenase contendo uma proteína MoFe marcada com histidina em ambos os casos. Quando eles analisaram a atividade dessas amostras, as amostras de extrato bruto sem ditionita ficaram quase inativas durante a redução do substrato - devido ao esgotamento de elétrons nos extratos brutos durante a ruptura celular. Kang et al. poderia, portanto, restaurar totalmente a atividade das amostras adicionando ditionita e ATP (ou seja, fornecendo elétrons).
p
Aglomerados de nitrogenase - dois metaloclusters únicos:o cluster P e o cluster M.
p Com base nas condições descritas, quando uma cultura que expressa nitrogenase que realiza ativamente N
2 a fixação é submetida a lise celular sem suprimentos adicionais de elétrons, a nitrogenase permaneceu funcional. Embora potencialmente preso em um estado "dormente" ou de ligação intermediária devido à retirada do fluxo de elétrons em um metalocluster ferro-enxofre conhecido como cluster M, localizado dentro da enzima nitrogenase. Quando Kang et al. purificou o extrato bruto livre de ditionita, a proteína MoFe marcada com histidina (designada como AV1 * no estudo) estava ativa durante N
2 redução e também totalmente funcional. Quando a equipe cristalizou AV1 *, eles observaram cristais marrons que difrataram a uma resolução de 1,83 angstrom (Å). Eles confirmaram o rearranjo estrutural dos dois agrupamentos P de AV1 * usando dados de densidade anômala e usaram ressonância paramagnética de elétrons para observar a atribuição estrutural. Os resultados forneceram-lhes respostas há muito procuradas para a relevância fisiológica deste estado experimental e apontaram para um fluxo limitado de elétrons entre os dois metaloclusters únicos (P- e M-clusters) do composto na ausência de ditionita.
p
 p Um mecanismo plausível de redução do N2 pela Mo-nitrogenase. Crédito:Markus Ribbe e Yilin Hu, Ciência , doi:10.1126 / science.aaz6748
Um mecanismo de ação plausível
p Um mecanismo plausível de redução do N2 pela Mo-nitrogenase. Crédito:Markus Ribbe e Yilin Hu, Ciência , doi:10.1126 / science.aaz6748
Um mecanismo de ação plausível
p Um mecanismo de ação plausível que concordou com as observações experimentais incluiu a redução gradual do dinitrogênio (N
2 )
nos três locais de enxofre do cinturão no catalisador de nitrogenase com base na rotação do cluster M. O mecanismo proposto começa com forte ligação de N
2 em um site específico, seguido pela rotação do N ligado
2 para outro local subsequente (locais designados como S3A a S2B a S5A no composto). Durante o processo, a redução / protonação de N
2 ao nível de diazene ocorreu através de ligações de hidrogênio, seguido por mais redução / protonação para sua conversão em amônia, antes de sua liberação da estrutura. A rotação subsequente do cluster trouxe um novo N
2 molécula para o próximo local para iniciar a próxima rodada de N
2 redução através da rotação contínua do cluster em um mecanismo delicado durante a catálise. Esse ciclo entre diferentes locais de reação era vagamente análogo ao mecanismo da enzima ATP sintase. O metalocluster rotativo, portanto, permitiu efetivamente a redução de múltiplos elétrons de N
2 por meio de uma abordagem de dividir e conquistar.
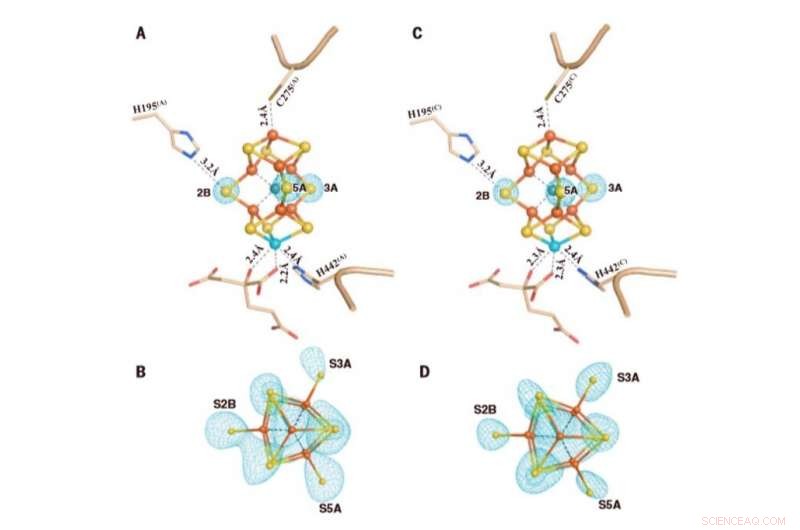 p Os clusters M em Av1 * (TOD). Estruturas de M-clusters em (A e B) cadeia A [M-cluster (A)] e (C e D) cadeia C [M-cluster (C)] refinadas em uma resolução de 1,73 Å. Vista lateral de (A) aglomerado M (A) e (C) aglomerado M (C) com resíduos chave interagindo com os aglomerados indicados como bastões. O agrupamento M (A) e o agrupamento M (C) são sobrepostos com os mapas de omissão de Fo-Fc dos enxofre da correia contornados em 13s (malha azul-menta). View along the Fe1-C-Mo direction of (B) M-cluster(A) and (D) M-cluster(C) superimposed with the anomalous density maps calculated at 7100 eV at a resolution of 2.17 Å and contoured at 4.0s, showing the presence of the anomalous sulfur density (mint-blue mesh) at all belt sulfur positions (S2B, S3A, and S5A) in (B) M-cluster(A) and (D) M-cluster(C). Crédito:Ciência, doi:10.1126/science.aaz6748
p Os clusters M em Av1 * (TOD). Estruturas de M-clusters em (A e B) cadeia A [M-cluster (A)] e (C e D) cadeia C [M-cluster (C)] refinadas em uma resolução de 1,73 Å. Vista lateral de (A) aglomerado M (A) e (C) aglomerado M (C) com resíduos chave interagindo com os aglomerados indicados como bastões. O agrupamento M (A) e o agrupamento M (C) são sobrepostos com os mapas de omissão de Fo-Fc dos enxofre da correia contornados em 13s (malha azul-menta). View along the Fe1-C-Mo direction of (B) M-cluster(A) and (D) M-cluster(C) superimposed with the anomalous density maps calculated at 7100 eV at a resolution of 2.17 Å and contoured at 4.0s, showing the presence of the anomalous sulfur density (mint-blue mesh) at all belt sulfur positions (S2B, S3A, and S5A) in (B) M-cluster(A) and (D) M-cluster(C). Crédito:Ciência, doi:10.1126/science.aaz6748
p To understand the sulfur-displaced conformation of AV1* under limited electron flux, the team formed AV1* turnover with dithionite (designated as AV1*TOD), to yield brown crystals that diffracted to a resolution of 1.73 Å. The observations were consistent with the mechanism of bound dinitrogen species on the compound and illustrated the physiological relevance of the conformation during catalysis. The capacity to displace three different sites by a dinitrogen species was consistent with previous investigations on catalysis-dependent selenium. Kang et al. proposed many mechanisms to explain the observations, however they seek further experimental support to verify them. The team highlighted the possibility for all belt-sulfur sites to be involved in the process of catalysis due to the presence of asymmetric belt-sulfur displacements in the compound. The results aim to provoke a paradigm shift in the mechanistic thinking of nitrogenase activity, ultimately to understand the intricate mechanism of the enzyme. p © 2020 Science X Network



