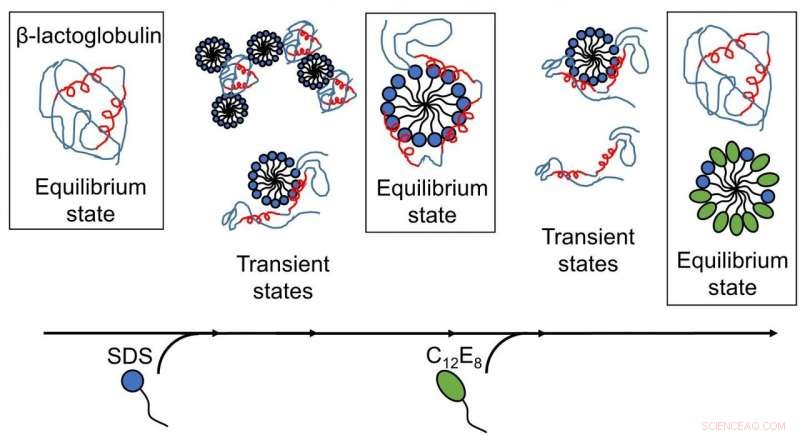
Resultados publicados por pesquisadores da AU revelam que o desdobramento e redobramento de proteínas mediados por surfactante são processos complexos com várias estruturas presentes, e os rearranjos ocorrem em escalas de tempo de submilissegundos a minutos. (Imagem:reproduzida com permissão da Royal Society of Chemistry). Crédito: Chem Sci , copyright 2020 Royal Society of Chemistry.
Compreender as interações entre as proteínas e as moléculas de sabão (surfactantes) tem sido importante para a indústria, particularmente em detergentes e cosméticos. O surfactante aniônico dodecil sulfato de sódio (SDS) é conhecido por desdobrar proteínas globulares, enquanto o surfactante não iônico octaetilenoglicol monododecil éter (C12E8) faz o oposto, ou seja, ajuda as proteínas a voltarem a ter forma.
Para que os pós de lavagem funcionem de forma eficiente, é importante que os surfactantes não alterem a estrutura das proteínas (enzimas), como qualquer mudança na estrutura da enzima mata sua capacidade de quebrar manchas e remover a sujeira. A maioria dos detergentes em pó contém misturas de surfactantes que permitem que as enzimas permaneçam ativas. Também, algumas biotecnologias exploram surfactantes em combinação com proteínas.
As proteínas da membrana geralmente ficam na membrana celular. A fim de extraí-los deste ambiente para diferentes estudos, eles têm que ser solubilizados por surfactante. O surfactante deve ser "suave" e cobrir apenas a parte da proteína inserida na membrana para que sua estrutura seja preservada. Em contraste, ao caracterizar o peso molecular das proteínas em laboratório, uma técnica padrão é desdobrá-los pelo surfactante agressivo carregado negativamente, SDS, e monitorar como eles migram em um gel de polímero em um campo elétrico. Essa técnica só funciona se o surfactante desdobrar completamente as proteínas e destruir sua estrutura.
Ainda há debate sobre qual tipo de interação entre a proteína e o surfactante é mais importante. São as interações eletrostáticas entre as cargas do surfactante e da proteína, ou são simplesmente as propriedades da interface dos agregados (micelas) que os surfactantes formam na água, quais são os responsáveis pelo desdobramento da proteína?
Embora o desdobramento tenha sido estudado em detalhes no nível da proteína, um quadro completo da interação entre proteína e surfactante está faltando nesses processos. Essa falta de conhecimento é abordada no presente trabalho usando a proteína globular β-lactoglobulina (bLG) como proteína modelo.
A combinação certa de técnicas experimentais
Uma visão mais profunda sobre o desdobramento e redobramento de proteínas foi obtida, já que as várias etapas das interações entre o surfactante e as proteínas foram mapeadas em função do tempo. Em primeiro lugar, a proteína modelo, bLG, foi misturado com o surfactante aniônico SDS enquanto acompanhava a evolução temporal da formação de complexos entre proteínas e moléculas de surfactante na escala de milissegundos-minuto. Com isso, os pesquisadores determinaram a estrutura dos complexos em evolução. Posteriormente, eles mapearam o curso do tempo do processo de redobragem quando o surfactante não carregado (C12E8) foi adicionado a uma amostra contendo complexos de SDS e proteína.
A fim de observar como a proteína se reorganiza durante o processo de desdobramento e redobramento induzido por surfactantes, técnicas espectroscópicas complementares, dicroísmo circular e fluorescência de triptofano, foram usados em combinação com espalhamento de raios X de pequeno ângulo resolvido no tempo (SAXS).
O dicroísmo circular e as alterações do monitor de fluorescência de triptofano na estrutura de bLG, enquanto as mudanças na forma geral dos complexos de proteína-surfactante foram seguidas por SAXS síncrotron. Esta combinação de técnicas não foi usada antes para estudar esses processos.
Processos complexos que duram de milissegundos a minutos
O desdobramento da proteína por SDS foi um processo homogêneo, onde todas as moléculas de proteína seguem a mesma rota de desdobramento. Os complexos de SDS (micelas) atacam as moléculas de proteína de frente e, em seguida, desdobram gradualmente a proteína de modo que forme uma concha em torno da micela de SDS. O redobramento começa quando as micelas C12E8 "sugam" SDS do complexo proteína-SDS para formar micelas SDS-C12E8 mistas. Contudo, o processo de redobramento real parece seguir várias rotas, uma vez que várias estruturas foram encontradas se formando em paralelo, a saber, complexos de proteína-surfactante (provavelmente contendo SDS e C12E8), micelas mistas de SDS e C12E8, proteínas "nuas" desdobradas como longas cadeias poliméricas, e proteínas devidamente dobradas. O experimento permitiu acompanhar a interconversão entre essas espécies, so that it could be determined which of the processes are fast and which ones are slow. The folded protein could form both from the naked unfolded proteins (quickly) and from protein-surfactant complexes (more slowly). Assim, the best way in which surfactants can help a protein to fold is to basically get out of the way and let the protein find its own way back to the folded state.
The results have provided deeper insight into the structural changes occurring at the protein-surfactant level. They revealed that surfactant-mediated unfolding and refolding of proteins are complex processes of rearrangements occurring on time scales from below milliseconds to minutes and involve intimate collaboration between surfactant complexes and proteins.